新規インフォマティクスワークフローを使用した mRNA 消化物のオリゴマッピング
要約
LC-MS およびインフォマティクステクノロジーの最近の進歩により、mRNA の重要特性分析の革新的な戦略設計における科学者の取り組みをサポートしています。このアプリケーションノートでは、RNase T2 酵素を使用したサンプル前処理から UPLC™-QTof MS を使用したデータ取り込みまでの、mRNA 配列マッピングのワークフロー、およびデータ分析のための新規インフォマティクスのワークフローについて説明します。新規の RNA 消化酵素、および緑色蛍光タンパク質(GFP)mRNA の消化オリゴマッピングの自動ソフトウェア解析の併用について詳述します。オリゴヌクレオチド消化産物の独自の精密質量に基づいたシーケンスカバー率の向上について説明しています。5' キャッピング効率の分析では、配列確認に加えて、新規の RNase T2 消化アプローチの使用にメリットがあることも実証しました。
アプリケーションのメリット
- waters_connect™ MAP Sequence アプリを使用する新しいインフォマティクスワークフローにより、UPLC QTof MS を用いて取り込まれた mRNA 消化物データのオリゴヌクレオチドマッピングが合理化される
- RNase T2 酵素(MC1 およびクサチビン)によって、ユニークな切断特異性とオーバーラップする消化産物を生成できる可能性が得られ、従来の RNase T1 消化と比較して、高いシーケンスカバー率が得られる
はじめに
新型コロナウイルスに対する最近の 2 種類の mRNA ベースのワクチンの開発および承認により、RNA ベースの医薬品がバイオ医薬品業界の最前線に押し出されました1-3。 迅速な製品開発の必要性により、配列および修飾のインテグリティなどの mRNA の重要品質特性(CQA)の精密な特性解析に用いる分析法の開発が必要になっています。以前、waters_connect INTACT Mass アプリを利用して mRNA の CQA を直接評価する 2 種類のワークフロー(5' キャッピング効率の測定とポリ(A)テールの不均一性分析)が、Waters™ によって開発されました。また、MAP Sequence アプリを利用する別のワークフローが最近、100 mer 一本鎖ガイド RNA のオリゴマッピングについて実証されました4,5,6。
Sanger 法シーケンシングや次世代シーケンシング(NGS)などの従来のオリゴヌクレオチドシーケンシング手法は、その費用対効果とスループットにより、mRNA のシーケンス解析に適用されてきましたが、これらには製造中に発生する可能性のある多くの修飾や mRNA 分子の分解を評価する機能がありません。一方、卓越した特異性、感度、定量性能で知られる LC-MS ベースのアプローチがより一般的になりつつあり、塩基および骨格の修飾に容易に対処することができます7-10。 全体として、複雑なバイオ医薬品の特性解析では、製品品質の包括的な視点を得るために直交する手法が望まれています。
従来、RNA 消化物オリゴマッピングの LC-MS ワークフローは、相当量の手作業でのデータ分析およびキュレーションを伴う、手間と時間のかかる作業でした。最近のアプリケーションノートで、RNase T1 酵素と RNase T2 酵素(RapiZyme MC1 およびクサチビン)や hRNase4 などの一連の酵素による個別の消化の後に sgRNA 配列を自動的にマッピングする UPLC-MS およびインフォマティクスワークフローについて説明しました6。 このアプリケーションノートでは、このワークフロー(図 1 に示す)が、複数の RNA 消化酵素によって消化されたより大きな mRNA 分子にまで適用できることについて説明します。これには、最近発売された 2 種類の RNase T2 酵素(RapiZyme MC1 および RapiZyme クサチビン)が含まれており、シーケンスカバー率を高めるユニークな切断パターンを有しています6,11。 sgRNA 消化産物の迅速な(2 分未満の)自動割り当て、並びにデータ解析およびレビューのための合理化されたインターフェースは、sgRNA における大きな利点となり、より複雑な mRNA の特性解析の効率がさらに高まると考えられます。さらに、RapiZyme MC1 および RapiZyme クサチビンによる消化では、一般に使用されている RNase T1 による消化アプローチと比較して、mRNA のキャッピング効率の共測定において優れた結果が得られることが示されています。
実験方法
試薬およびサンプル前処理
ジプロピルエチルアミン(DPA、純度 99%、カタログ番号 D214752–500ML)および 1,1,1,3,3,3-ヘキサフルオロ-2-プロパノール(HFIP、純度 99%、カタログ番号 105228-100G)は、Millipore Sigma(ミズーリ州セントルイス)から購入しました。メタノール(LC-MS グレード、カタログ番号 34966-1L)は Honeywell(ノースカロライナ州シャーロット)から入手しました。HPLC グレードのタイプ I 脱イオン水(DI)は、Milli-Q システム(Millipore、マサチューセッツ州ベドフォード)を使用して精製しました。移動相は毎日新しく調製しました。mRNA 消化用のヌクレアーゼフリー超高純度水(カタログ番号 J71786.AE)は、Thermo Fisher Scientific(マサチューセッツ州ウォルサム)から購入しました。
クラゲ緑色蛍光タンパク質(GFP)配列に基づく mRNA コンストラクトは、Biosynthesis(テキサス州、ルイスヴィル)により IVT(in vitro 転写)合成でカスタム合成されたものです。この mRNA 分子は、(配列 7MeGpppA(2'-OMe)、元素組成 C32H42N15O26P5)の Cap1 構造、その後に 1019 ヌクレオチドが続き、ポリ(A)テール配列なしで合成されています。
Aspergillus oryzae から単離され、クロマトグラフィー精製された、動物質が含まれないリボヌクレアーゼ T1(カタログ番号 LS01490、500 kU)は、Worthington Biochemical Corporation(ニュージャージー州、レイクウッド)から購入しました。凍結乾燥した酵素を、5 mL の 100 mM 重炭酸アンモニウム(カタログ番号 5.33005-50G、Millipore Sigma)に溶解し、100 単位/µL の溶液を調製しました。RNase T1 による mRNA 消化では、5 µL の 5 µM GFP mRNA を、25 µL のヌクレアーゼフリー水および 10 µL RNase T1 酵素(1000 単位)と混合し、37 ℃ で 15 分間消化しました。消化混合液は、QuanRecovery™ MaxPeak™ 300 µL バイアル中に調製しました。消化物 5 µL を注入し、LC-MS によって直ちに分析しました。
RapiZyme MC1(製品番号:186011190、10000 単位/チューブ)および RapiZyme クサチビン(製品番号:186011192、10000 単位/チューブ)は、ウォーターズコーポレーションが最近発表した 2 種類の新規 RNA 消化酵素です6,7。 RapiZyme MC1 とクサチビンに使用した GFP mRNA 消化プロトコルは、非常に類似しています。RapiZyme MC1 の場合、GFP mRNA(10 µL、5 µM 溶液)を、200 mM 酢酸アンモニウム(pH 8.0)を含むバッファー中、90 ℃ で 2 分間変性しました。RapiZyme クサチビンの場合、mRNA(10 µL、5 µM 溶液)を、200 mM 酢酸アンモニウム(pH 9.0)を含むバッファー中、90 ℃ で 2 分間変性しました。いずれのサンプルも氷上で冷却し、微量遠心してサンプルの液滴を回収しました。50 単位の消化酵素(RapiZyme MC1 または RapiZyme クサチビンのいずれかを 1 µL)とヌクレアーゼフリー水 8 µL を添加して最終量を約 20 µL にした後、mRNA を Eppendorf サーモミキサー中、37 ℃ で 60 分間消化しました。70 ℃ で 15 分間加熱して酵素消化を停止させ、酵素を不活性化しました。消化物 5 µL を注入し、LC-MS によって直ちに分析しました。
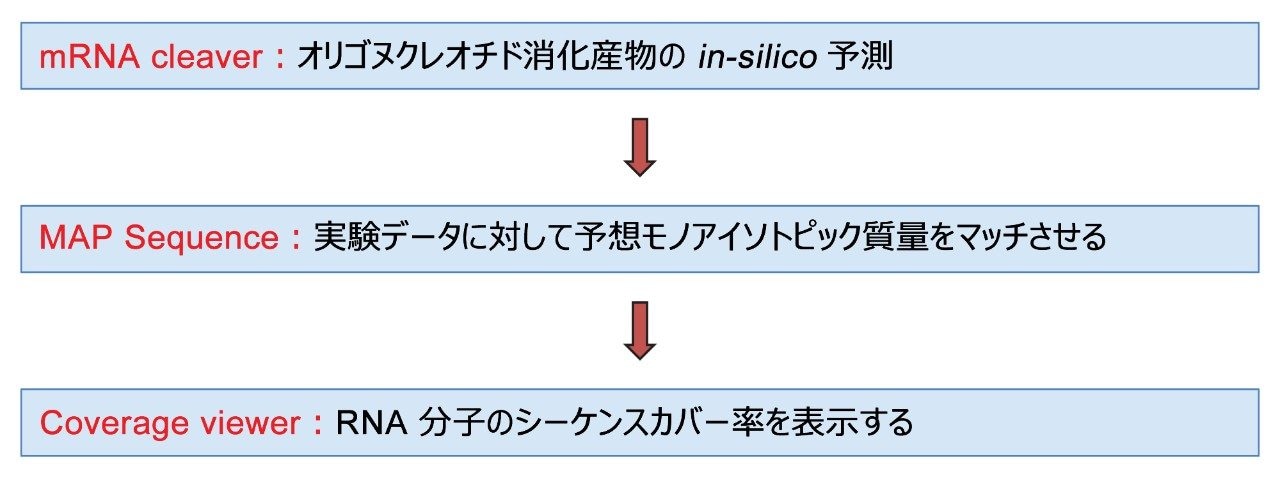
データセットはすべて、waters_connect UNIFI™ アプリバージョン 3.6.0.21 を使用して取り込んだ後、mRNA Cleaver MicroApp および Coverage Viewer MicroApp を補助とする MAP Sequence アプリを使用して解析しました。
LC 条件
|
LC-MS システム: |
ACQUITY™ Premier UPLC(バイナリー)システムを搭載した Xevo™ G3 QTof LC-MS |
|
カラム: |
ACQUITY Premier Oligonucleotide BEH™ C18 FIT カラム、130 Å、1.7 µm、2.1 × 150 mm(製品番号:186009486) |
|
カラム温度: |
60 ℃ |
|
流量: |
300 µL/分 |
|
移動相: |
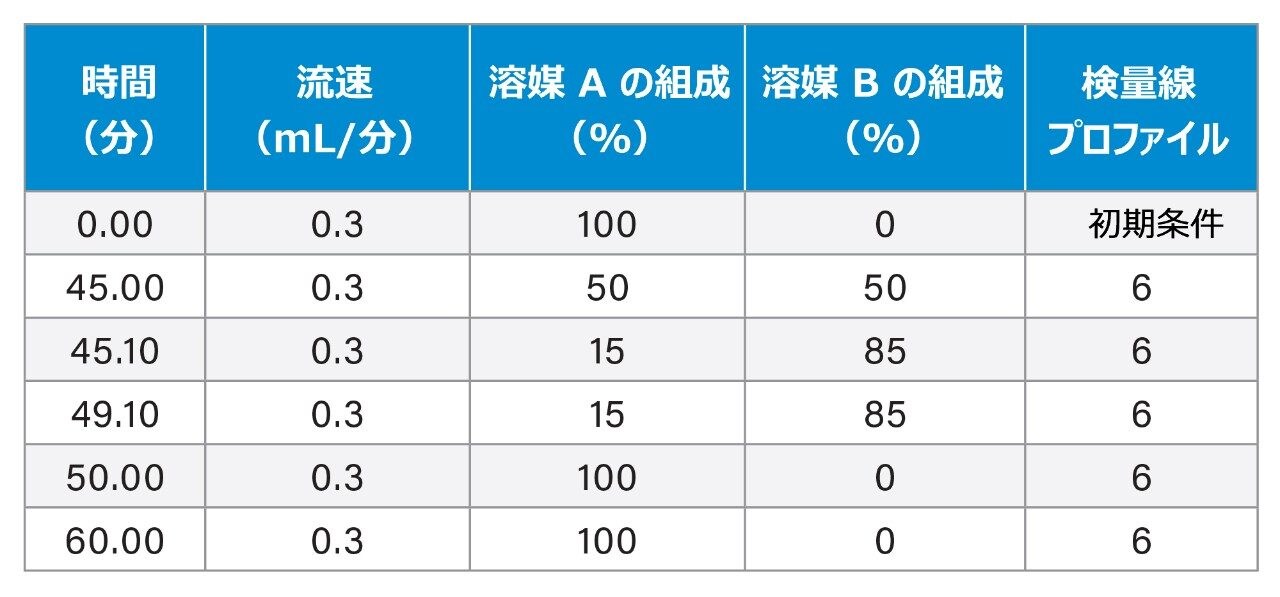
溶媒 A:10 mM DPA(ジプロピルエチルアミン)、40 mM HFIP(1,1,1,3,3,3-ヘキサフルオロイソプロパノール)含有脱イオン水、pH 8.5 溶媒 B:10 mM DPA、40 mM HFIP 含有 50% メタノール |
|
サンプル温度: |
8 ℃ |
|
サンプルバイアル: |
QuanRecovery MaxPeak HPS バイアル(製品番号:186009186) |
|
注入量: |
5 µL |
|
洗浄溶媒: |
パージ溶媒:50% メタノール サンプルマネージャー洗浄溶媒:50% メタノール シール洗浄:20% アセトニトリル含有脱イオン水 |
グラジエントテーブル
MS 条件
|
MS システム: |
Xevo™ G3 QTof 質量分析計 |
|
イオン化モード: |
ESI(-) |
|
取り込みモード: |
MSE |
|
取り込み速度: |
1 Hz |
|
キャピラリー電圧: |
2.5 kV |
|
コーン電圧: |
40 V |
|
イオン源オフセット: |
60 V |
|
イオン源温度: |
120 ℃ |
|
脱溶媒温度: |
550 ℃ |
|
コーンガス流量: |
50 L/時間 |
|
脱溶媒ガス流量: |
600 L/時間 |
|
ToF 質量範囲: |
340 ~ 4000(MSE 取り込み) |
|
低エネルギー CE: |
6 V |
|
高エネルギー CE ランプ: |
25 ~ 50 V |
|
ロックマス: |
50 pg/µL Leu Enk |
|
データ取り込み: |
waters_connect 3.6.0.21 |
|
データ解析: |
waters_connect 3.6.0.21 |
|
データ解析: |
mRNA Cleaver MicroApp v1.1.0 |
結果および考察
バイオ医薬品企業が製品開発パイプラインを進める上で、RNA 配列解析に LC-MS ベースのメソッドの採用を増やすことは、より徹底した初期特性解析と信頼性の高い継続的な配列確認を行う上で有用です。クロマトグラフィーの再現性と分解能の改善、高質量分解能 MS の使いやすさの向上、MS システムの感度と精度の改善により、確実な配列確認と塩基修飾の検出が可能になりました。最も重要なこととして、新しいオリゴヌクレオチドインフォマティクスツールにより、RNA マップのデータ分析という面倒な作業がより迅速かつ簡単に行えるようになりました。
バイオ医薬品タンパク質の消化ペプチドマッピング用 LC-MS はルーチンで行われますが、RNA に対してこれと同等のアプローチを使用することはより困難です。20 種類のアミノ酸で構成されるタンパク質とは異なり、mRNA には 4 つの構成要素(A、C、G、T)しか含まれないため、mRNA にマッピングできる固有の質量を持つオリゴヌクレオチド消化産物の生成はより困難になります。G で切断するという特異性を有する RNase T1 のような高頻度切断酵素では、割り当てがあいまいになる異性体や同一の mRNA 消化産物が多く生成します。低頻度の切断部位を有する酵素は、特に非特異的切断したオリゴヌクレオチド集団を生成する場合に、より長い消化産物を生成することができます。本研究で使用した新規の RNase タイプ 2 酵素(RapiZyme MC1 および RapiZyme クサチビン)では、一意の質量を持つ割り当て可能な消化産物が生じる可能性が高くなります。
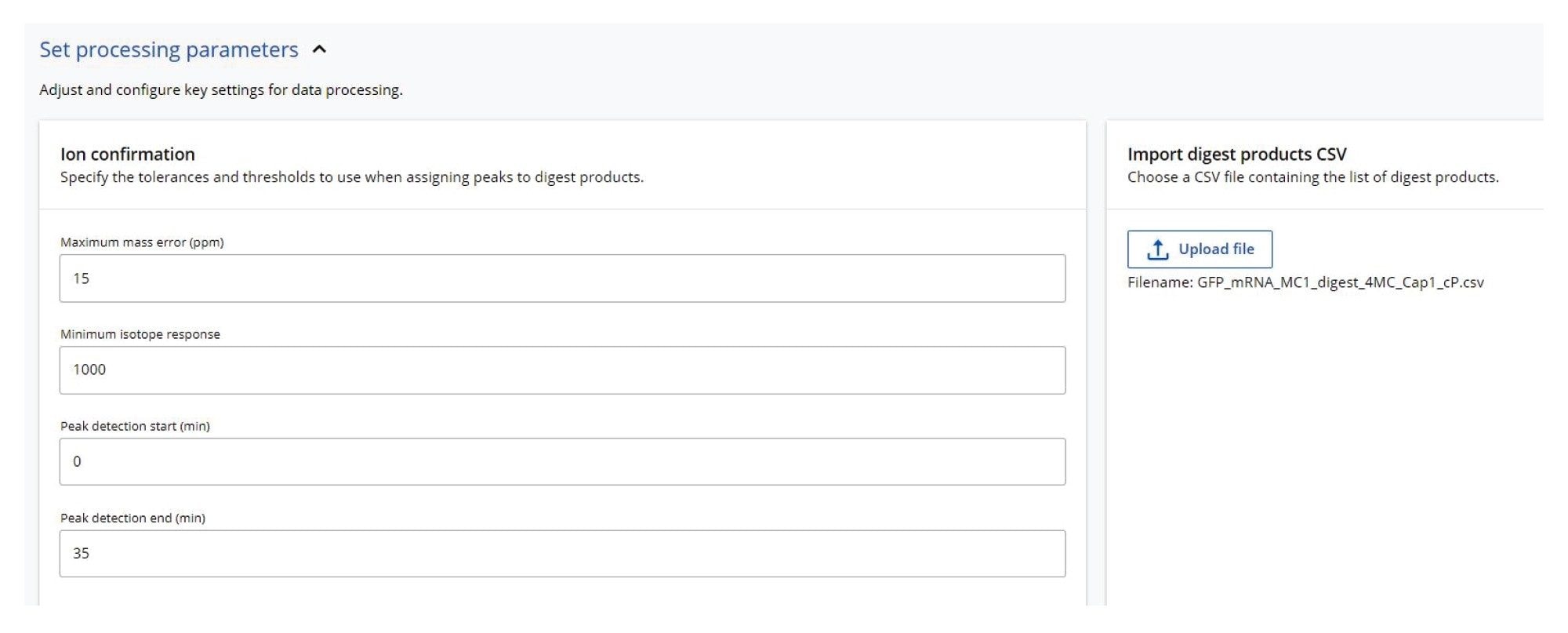
データ解析および配列割り当て用の waters_connect MAP Sequence アプリを使用するインフォマティクスワークフロー(図 1)は、酵素消化および UPLC-MS データ取り込みの後に取り込み済み LC-MS データセットの迅速かつ自動のデータ解析が容易に行えるように設計されています6。 最初のステップでは、mRNA Cleaver MicroApp を使用して、指定した mRNA 分子の予測 in-silico 消化オリゴヌクレオチド産物を生成します。2 番目のステップでは、waters_connect MAP Sequence アプリによって UPLC-MS データを解析し、消化されたオリゴの予測中性モノアイソトピック質量を、実験的に得られた MS1 データとマッチングします。最後のステップでは、mRNA 消化に関して得られたシーケンスカバー率をまとめて、Coverage Viewer MicroApp を使用して視覚化します。
このワークフローではオリゴマッピングに RNA 消化産物の精密質量測定を利用するため、酵素消化によって明確で一意の生成物を生成して、割り当てのあいまいさを解消する上で MS2 フラグメンテーションデータに関するユーザー操作にあまり頼らないようにすることが重要です。RapiZyme MC1 酵素および RapiZyme クサチビン酵素では、そのユニークな切断特異性と、酵素量と消化時間を調整することによって意図的に生成する非特異的切断の組み合わせにより、より長い一意の消化産物を生成することができます。オリゴヌクレオチド消化産物の割り当ての確認には、データ非依存的 MSE 取り込みを使用します。高エネルギー MSE フラグメンテーション情報が、アプリの今後のリリースに取り入れられる予定です。これは特に、その他のあいまいな割り当てに対処するためです。
mRNA オリゴマッピング
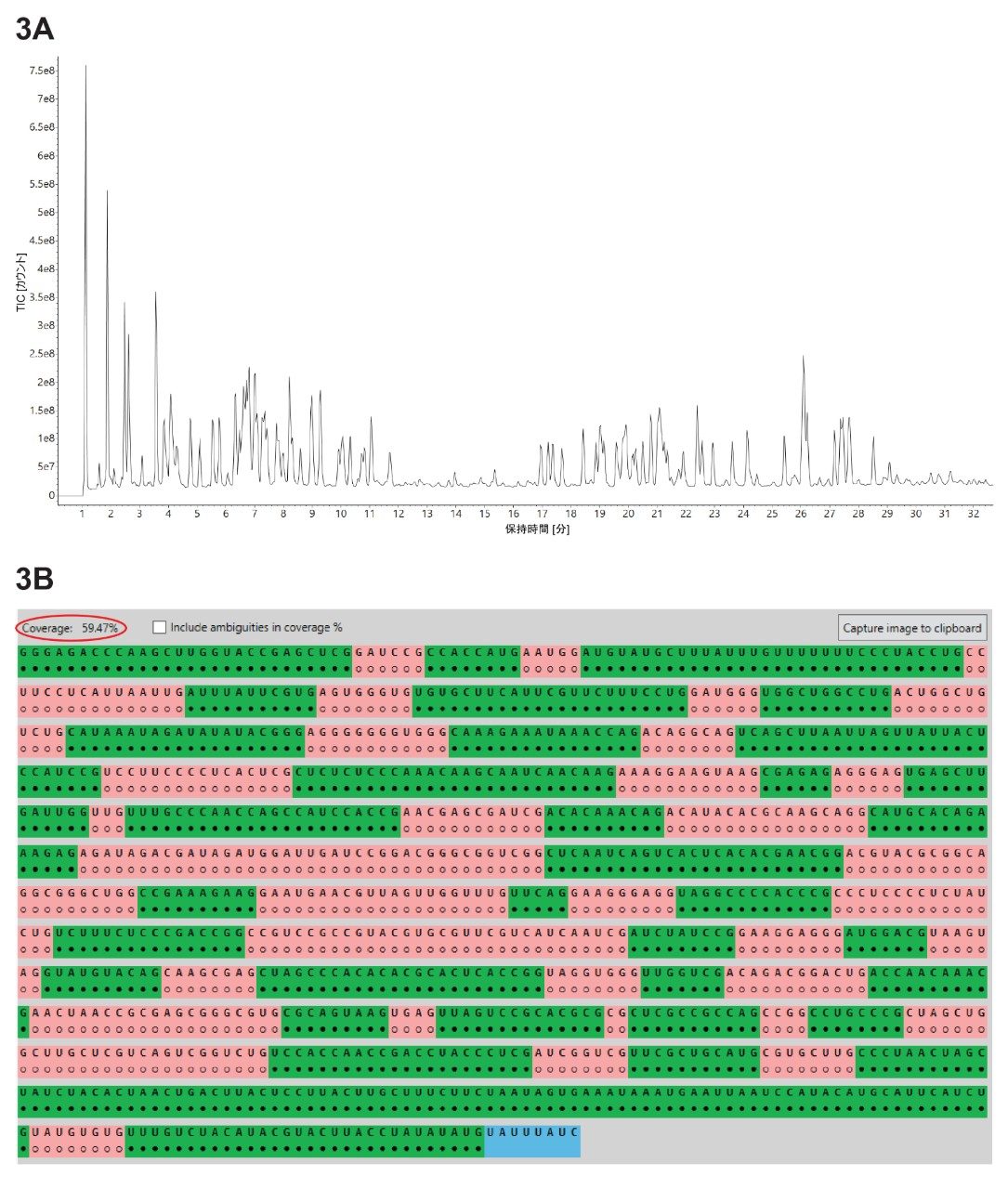
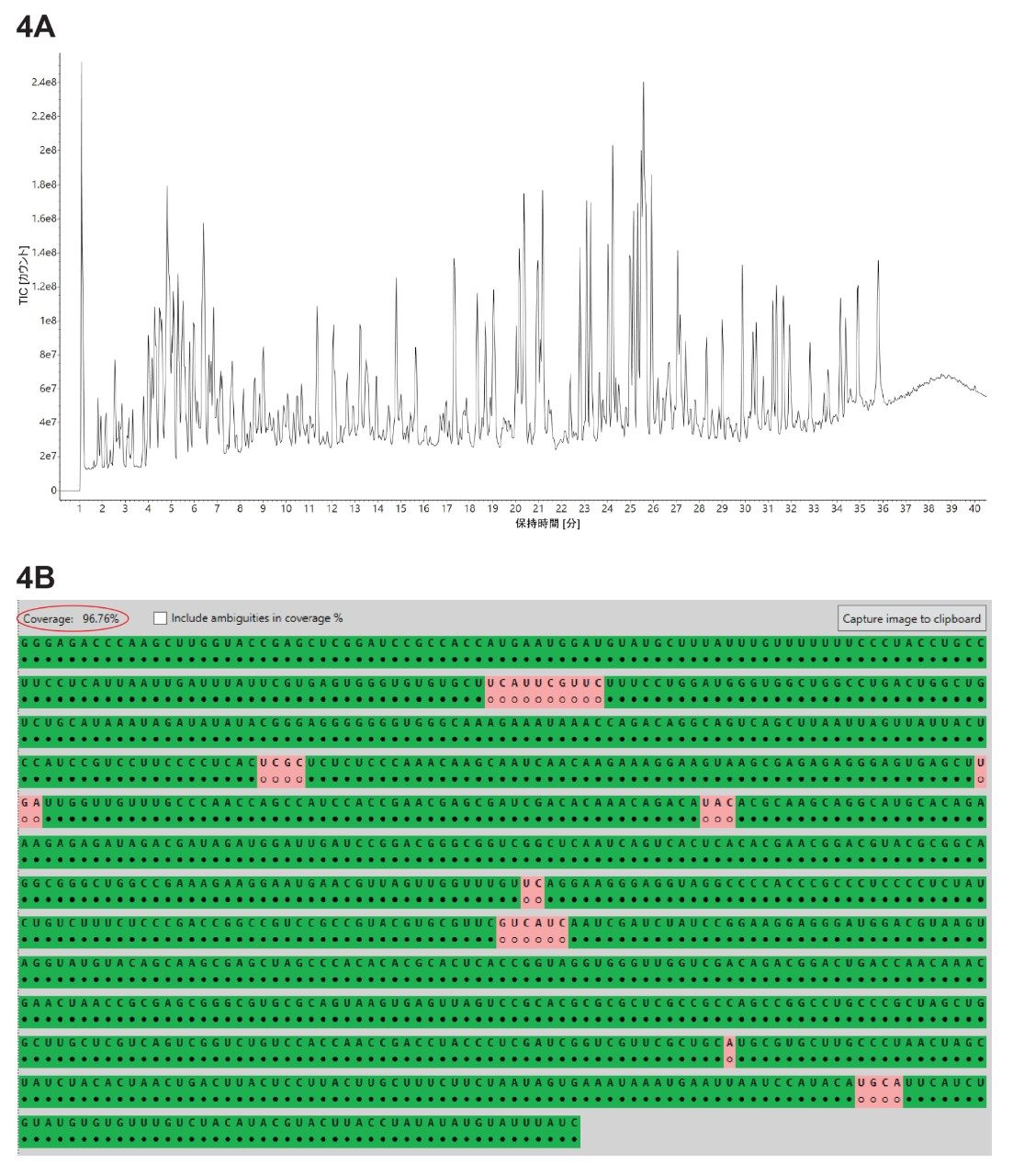
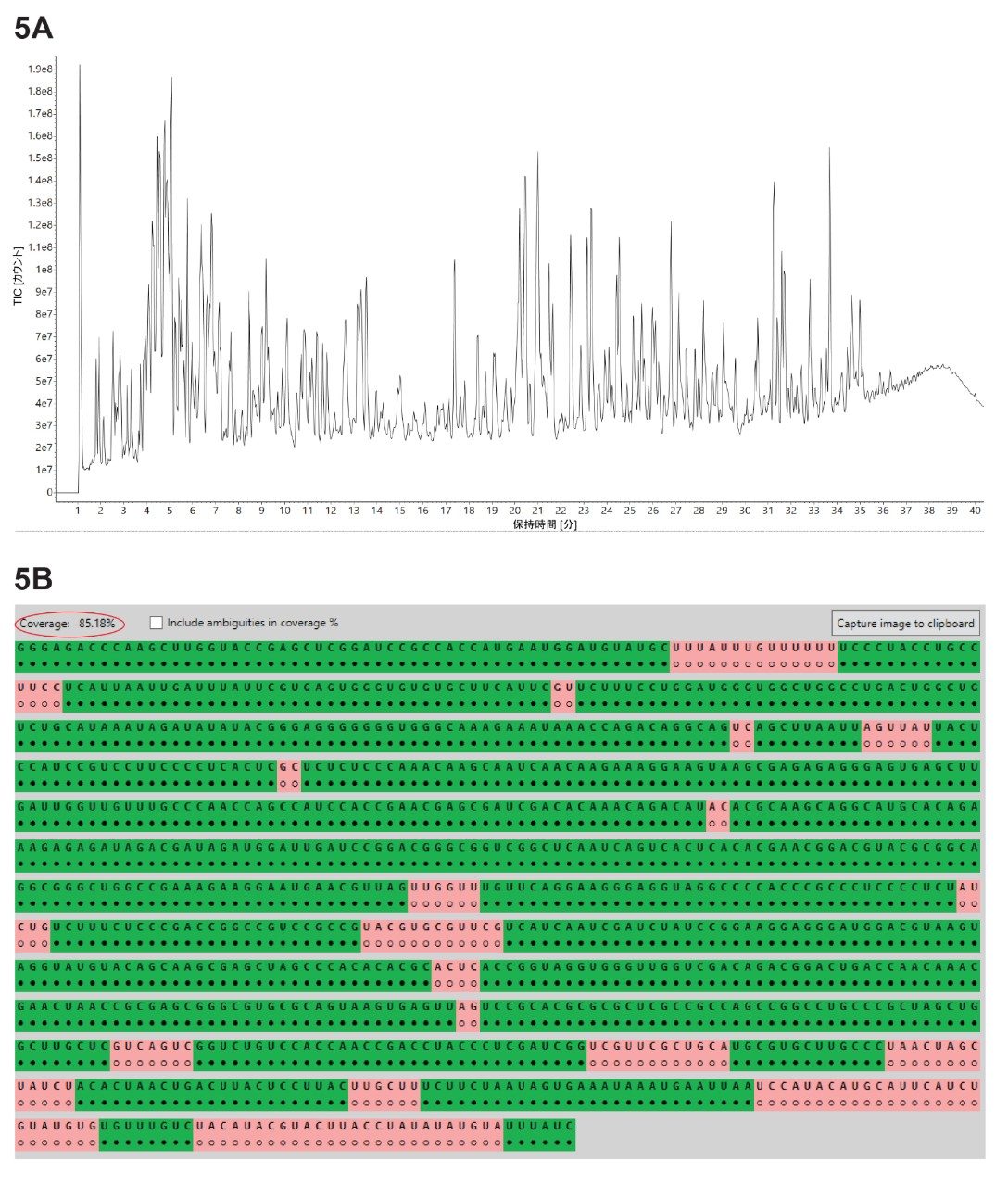
3 種類のリボヌクレアーゼ(RNase T1、RapiZyme MC1、RapiZyme クサチビン)で消化した GFP mRNA サンプルについて、3 つの独立した LC-MSE オリゴマップデータセットを取り込みました。MAP Sequence アプリを用い、図 2 に示したパラメーターを使用して 3 つの UPLC-MSE データセットを解析し、結果を Coverage Viewer MicroApp にエクスポートして、シーケンスカバー率が速やかに視覚化されました。RNase T1(図 3A)、Rapizyme MC1(4A)、RapiZyme クサチビン(5A)について記録された 3 つの TIC クロマトグラムを示しています。MC1 およびクサチビンは、RNase T1 と比較してかなり多数の非特異的切断を生じることが知られており、この酵素特性は図 4A および 5A に示したクロマトグラムの複雑さに明確に反映されています6-9。
配列マッピングの結果(図 3B、4B、5B)の比較により、MC1(約 97%、4B)およびクサチビン(約 85%、5B)での分析と比較して、RNase T1 では一意のカバー率が大幅に低いことがわかります(約 60%、3B)。これは、割り当てがあいまいな消化産物の数がはるかに多かったためです。これは、非常に特異的なジヌクレオチドモチーフに依存する MC1 およびクサチビンの切断特異性とは対照的に、RNase T1 の特異性が非常に広い(すべての G 残基の後で切断)ことに直接起因しています6-9。 MC1 はウリジン残基の 5' 末端で切断し、3 つの主要な切断部位(A_U/C_U/U_U)と 2 つのマイナーな切断部位(C_A/C_G)があります。クサチビンはシチジン残基の 3' 末端で切断し、4 つの主要な切断部位(C_A/C_G/C_U/U_A)と 3 つのマイナーな切断部位(A_U/G_U/U_U)があります。RNase T1 は、そのすべての消化産物の 3' 末端に直線状リン酸を付加するのに対し、MC1 とクサチビンではいずれも、主に 3' 環状リン酸を含む消化産物が生じます。
MC1 とクサチビンは、RNase T1 と比較して、制御された非特異的切断を行う能力が高いため、mRNA Cleaver 予測ツールでは、これらの 2 種の酵素について、最大 4 箇所の非特異的切断が許容されましたが、RNase T1 では非特異的切断は 2 箇所にのみ適用されました。非特異的切断が多いことで、MC1 とクサチビンはいずれも、RNase T1 と比較して長い消化産物を生成しました。これらの長いオリゴヌクレオチド産物では、一意の質量を有する可能性が高く、あいまいな割り当てになる可能性が減ります。このような酵素特性の結果から、MC1 およびクサチビンのマッピングの結果には、より良好なカバー率が見られます。
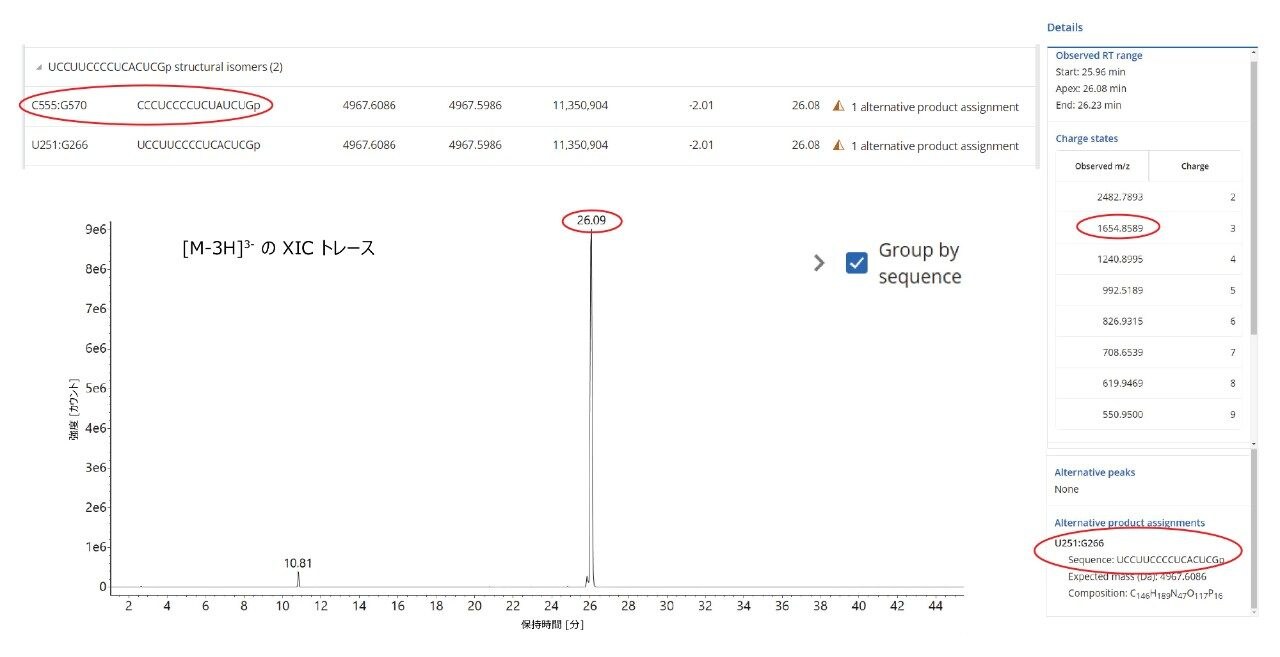
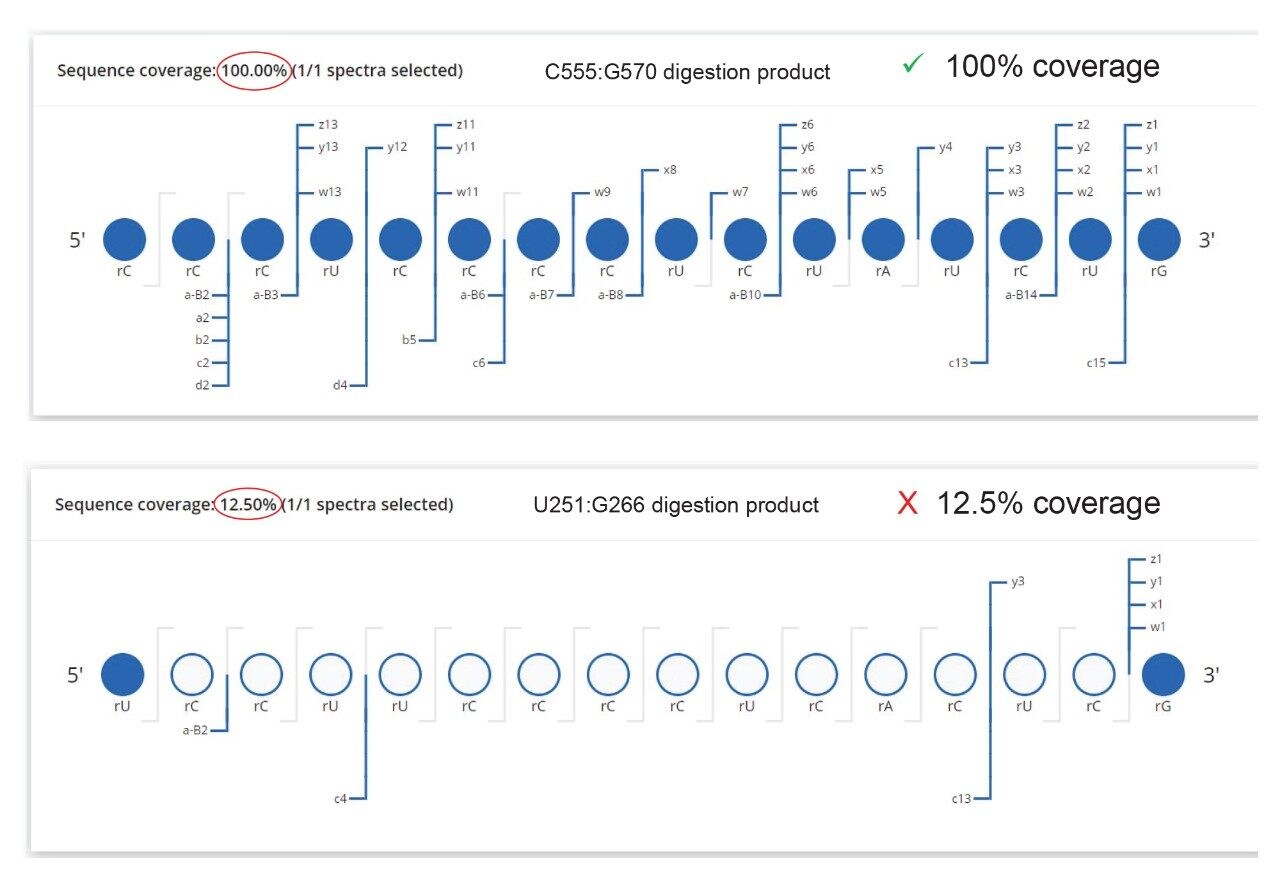
あいまいな配列割り当ての場合、CONFIRM Sequence waters_conect アプリを使用して、高エネルギー MSE データをさらに調査し、フラグメンテーションイオンを最も可能性の高いオリゴヌクレオチド配列に割り当てることができます12。このアプローチを説明するために、GFP mRNA の RNase T1 消化によって生成した 16 mer の消化産物のペアを選択しました。このペアは、同じヌクレオチド組成の構造異性体 U251:G266(配列 UCC UUC CCC UCA CUC G)と C555:G570(CCC UCC CCU CUA UCU G)によって成り立っています。挿入図に示すように(図 6 上)、MAP Sequence は、複数のプリカーサー(チャージ状態 2 ~ 9)に基づいて、これらの 2 つの配列を自動的に見つけ、「別の産物割り当て」とラベル付けしています。対応する三価プリカーサー(m/z=1654.86)(図 6 にも示す)の抽出質量クロマトグラムは、一方または両方のオリゴヌクレオチド産物に属する可能性のある、単一の高存在量のクロマトグラフィーピークの存在を示しています。CONFIRM Sequence を使用して、このオリゴヌクレオチド消化産物について記録された高エネルギー ESI-MSE スペクトル(図 7)中に検出されたフラグメントイオンを自動的に割り当て、C555:G570 配列のマッチをシーケンスカバー率 100% で確実に明らかにしました。
mRNA のキャップ分析のいくつかの例が、以前に公開されたウォーターズからのアプリケーションノートに記載されています5,13。mRNA のキャップは、mRNA の 5' 末端に付加され、通常は完全に修飾された 7 N-メチルグアノシンで構成され、三リン酸エステル(m7Gppp)を介して mRNA に 5'-5' 結合しています。GFP mRNA の場合、mRNA に結合したアデノシン残基の 2'-O-メチル化を含むバリアントの Cap1 構造が使用されています。そのため、GFP mRNA の Cap1 構造は m7GpppAm と表記されます。
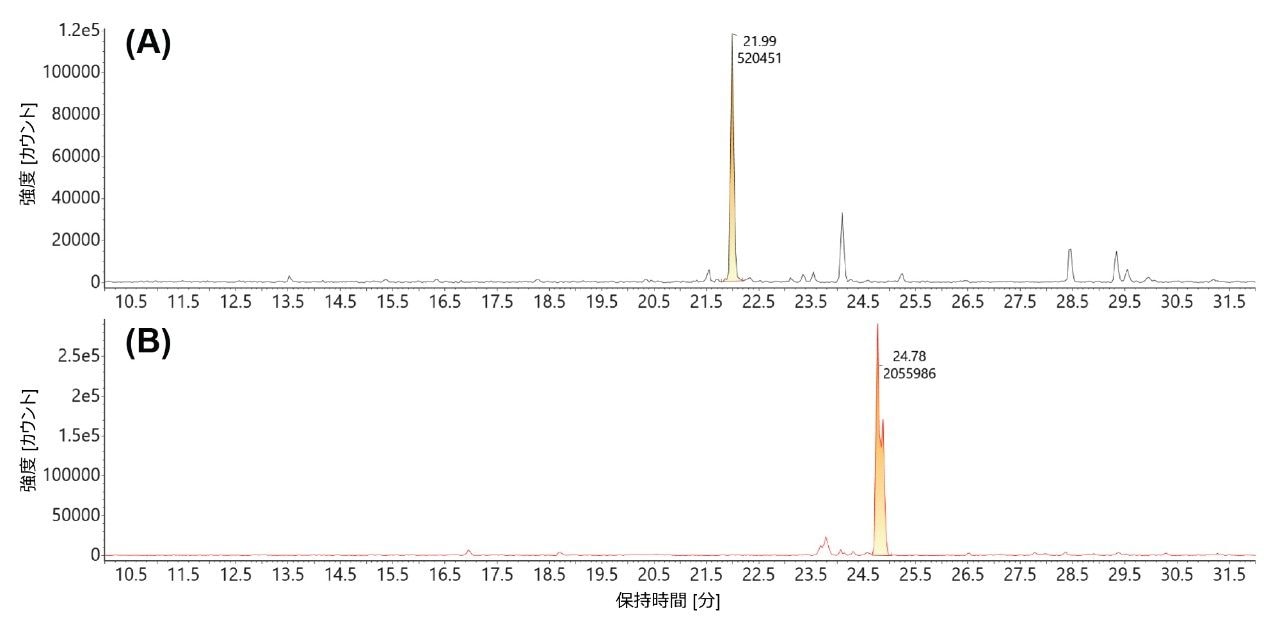
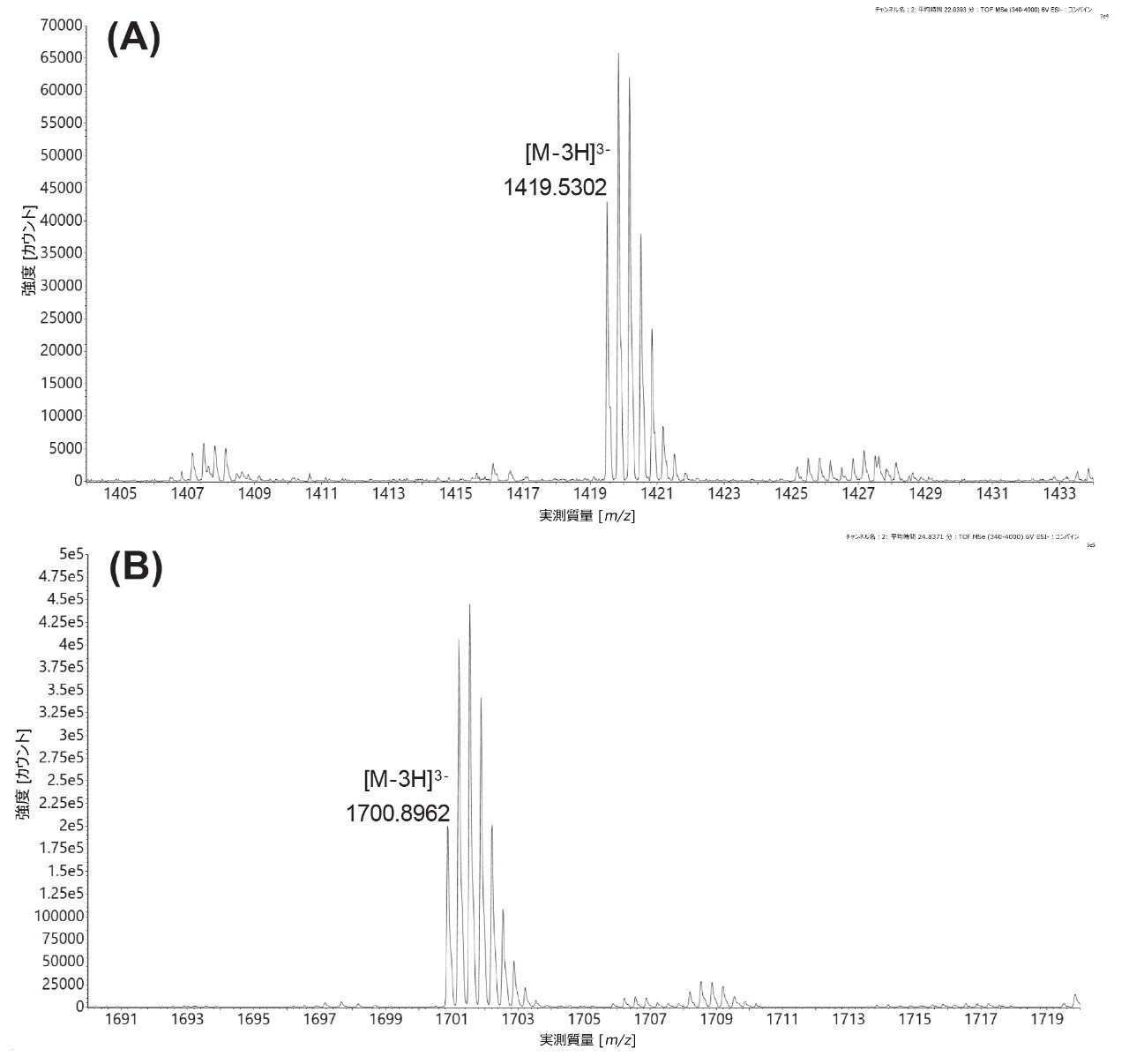
図 8 に示す抽出質量クロマトグラムでわかるように、MC1 mRNA 消化物中には、キャップなしおよびキャップ付きの両方の GFP 5' mRNA 消化産物が検出されています。MC1 消化後、キャップなしバージョン(m/z=1419.5302、-3)は上のトレースの 22.0 分に溶出し(図 8A)、Cap1 修飾分子種(m/z=1700.8962、-3)は下のトレースの 24.7 分に溶出しています(図 8B)。これらの三価のチャージ状態の XIC ピークから得られた波形解析済みピーク面積を使用して、キャッピング効率は 79.8% と算出されました。
これらのオリゴヌクレオチド分子種のアイデンティティーを、検出された最も存在量の多いチャージ状態([M-3H]3-)について記録された同位体分布(図 9)によってさらに確認します。配列が GGA AGA CCC AAG C の遊離(キャップなし)13 mer MC1 消化オリゴヌクレオチドのモノアイソトピックピークを図 8A に示しています。Cap1 修飾 13 mer オリゴヌクレオチドのモノアイソトピックピークを下のパネル(図 8B)に示しています。このケースでは、MC1 とクサチビンの両方で、GFP mRNA の 5' 末端を切断した後に、同じ消化産物(配列 GGA AGA CCC AAG C)が生じるため、この mRNA キャッピング効率の測定が可能になります。一方、RNase T1 消化の場合、mRNA の最初の 5' 末端残基がグアノシンであるため、キャップなしの消化産物は短すぎて(単一ヌクレオチド)、使用している IP-RP 条件下では保持されません。このため、Cap1 消化産物のみが検出され(配列は m7GpppAmGp、最も存在量が多いモノアイソトピックイオンは m/z=602.5482、-2、データは示していません)、mRNA キャッピング効率を測定できませんでした。したがって、GFP mRNA の場合、MC1 とクサチビンのみがキャッピング効率の計算に使用できました。
これらの結果により、高分子 mRNA の配列確認における質量分析の現在の有用性が浮き彫りになっていますが、ワークフローの効率と自動化にはまだ実現すべき点が残っており、この分野ではワークフローの開発が引き続き活発に行われています。このような複雑なデータセットを解析するためのソフトウェアの自動化を継続的に開発することは、消化酵素のツールセットの拡張とともに、この新たな方法論の有用性を高めるために重要です。
結論
- waters_connect MAP Sequence Waters アプリを活用した、新たなインフォマティクスワークフローにより、UPLC-MS 取り込みデータを使用した mRNA 消化物の MS1 オリゴマッピングが簡単になることを実証しました
- 2 種の新規の RNase T2 酵素(RapiZyme MC1 および RapiZyme クサチビン)では、RNase T1 とは異なるそのユニークな消化特異性により、完全消化オリゴヌクレオチドと非特異的切断オリゴのより大きな集団が生成し、より高度にオーバーラップしたより信頼性の高いシーケンスカバー率が得られました。
- 異性体の消化オリゴヌクレオチド産物を、waters_connect CONFIRM Sequence アプリを使用して区別し、高エネルギーフラグメントイオンを異性体オリゴの配列にマッチングすることができます
参考文献
- Xu S, Yang K, Li R, Zhang L. mRNA Vaccine Era-Mechanisms, Drug Platform and Clinical Prospection, Intl J Mol Sci Chem, 2020, 21 (18), 6582, doi: 10.3390/ijms21186582.
- Verbeke R, Lentacker I, De Smedt SC, Dewitte H. The dawn of mRNA vaccines: The COVID-19 case.J Controlled Release, 2021, 333, 511–520, doi: 10.1016/ j.jconrel.2021.03.043.
- Jackson NA, Kester KE, Casimiro D, Gurunathan S, DeRosa F. The Promise of mRNA Vaccines: A Biotech and Industrial Perspective.npj Vaccines, 2020, 5, https://doi.org/10.1038/s41541-020-0159-8.
- Synthetic mRNA Oligo-Mapping Using Ion-Pairing Liquid chromatography and Mass Spectrometry, 2022, Waters application note.June, 2022. 720007669.
- RNA CQA Analysis using the BioAccord LC-MS System and INTACT Mass waters_connect Application, 2023, Waters application note. November, 2023. 720008130.
- RNA Digestion Product Mapping Using an Integrated UPLC-MS and Informatics Workflow, 2024, Waters application note. September, 2024. 720008553.
- Jiang T, Yu N, Kim J, Murgo JR, Kissai M, Ravichandran K, Miracco E, Presnyak V, Hua S. Oligonucleotide Sequence Mapping of Large Therapeutic mRNAs via Parallel Ribonuclease Digestions and LC-MS/MS, Anal Chem, 2019, 91, 8500–8506, doi:10.1021/acs.analchem.9b01664.
- Vanhinsbergh CJ, Criscuolo A, Sutton JN, Keely M, Williamson AJK, Cook K, Dickman M. Characterization and Sequence Mapping of Large RNA and mRNA Therapeutics using Mass Spectrometry, Anal Chem, 2022, 94, 7339–7349, doi:10.1021/acs.analchem.2c00765.
- Gau B, Dawdy AW, Wang HL, Bare B, Castaneda CH, Friese OV, Thompson MS, Lerch TF, Cirelli DJ, Rouse JC.Oligonucleotide Mapping via Mass Spectrometry to Enable Comprehensive Primary Structure Characterization of an mRNA Vaccine Against SARS CoV-2, Sci Rep, 2023, 13, No 9038, doi:10.1038/s41598-023-36193-2.
- Tang S, Liu GY, Yan Y, Wang S, Li N. Development of a Flow Through-Based Limited Digestion Approach for High-Throughput and High-Sequence Coverage Mapping of Therapeutic mRNAs, Anal Chem, 2024, 96, 16944–17003, doi:10.1021/acs.analchem.4c04384.
- Tunable Digestion of RNA Using RapiZyme RNases to Confirm Sequence and Map Modifications, 2024, Waters application note. September, 2024. 720008539.
- CONFIRM Sequence: A waters_connect Application for Sequencing of Synthetic Oligonucleotide and Their Impurities, 2022, Waters application note.July, 2022. 720007677.
- Rapid Analysis of a Synthetic mRNA Cap Structure Using Ion-Pairing RPLC with the BioAccord LC-MS System, 2021, Waters application note.August, 2021. 720007329.
ソリューション提供製品
720008677JA、2025 年 1 月