UPLC-MS とインフォマティクスを統合したワークフローを使用した RNA 消化産物のマッピング
要約
LC-MS 分析は、その感度、特異性、およびさまざまなオリゴヌクレオチド修飾を検出する能力により、シングルガイド RNA(sgRNA)分析に必須のツールです。このアプリケーションノートでは、sgRNA の酵素消化後に取り込んだ LC-MS データセットの高速データ解析用に設計された新しいインフォマティクスツールを紹介します。この新しいワークフローには、waters_connect™ MAP Sequence アプリなどのデータ解析ソフトウェアが含まれています。酵素消化済みオリゴヌクレオチド生成物を、mRNA Cleaver MicroApp を使用して in-silico で生成し、MAP Sequence アプリによって実験データとマッチングして、結果が Coverage Viewer MicroApp で視覚化されます。今回説明するワークフローでは、一連の RNase T1、T2(RapiZyme™ MC1 およびクサチビン)、hRNase4 などのリボヌクレアーゼを使用したサンプル前処理から LC-MS 分析およびデータ解析までに焦点を当てます。さまざまな酵素特異性によって得られる一意の切断部位と(精密質量測定を使用する)新しいインフォマティクスツールの組み合わせにより、迅速かつ合理的により良好な sgRNA の配列確認が可能になり、医薬品開発の効率や、臨床やカスタマーに対する RNA ベースの製品のリリースの効率が向上します。
アプリケーションのメリット
- 規制対応の新しいインフォマティクスにより、UPLC-MS を使用した RNA 生成物のマッピングを効率化
- RNase T2 酵素は一意の切断部位を提供し、従来の RNase T1 消化と比較して、消化産物がオーバーラップする可能性が高い
- 酵素消化のパネルから得られる結果を組み合わせることにより、質量フィンガープリントベースのアプローチの正確さに対する信頼性が全体的に高まり、より高いまたは完全なシーケンスカバー率が得られる可能性が高まる
はじめに
シングルガイド RNA(sgRNA)は、二本鎖 DNA の切断に必要な 2 種の RNA 分子(Cas9 酵素の足場である tracRNA および DNA ターゲットの認識に関与する crRNA)をシングル RNA 構造に融合した際に、2012 年に発表された CRISPR の原著論文に初めて記載されました1。sgRNA は、CRISPR-Cas9 遺伝子編集システムの主要なコンポーネントです1-2。 sgRNA 分子は、Cas9 ヌクレアーゼに DNA 二本鎖を切断することを指示する分子であり、これによって遺伝子編集への扉が開かれました。2020 年にノーベル化学賞を受賞した CRISPR テクノロジーの発見により、遺伝子編集アプリケーションの分野に変革がもたらされました。その主要な用途の 1 つとして、2020 年における世界的なパンデミックの時期におけるシンプルで迅速な新型コロナウイルス感染症(COVID-19)検査の開発が挙げられますが、CRISPR テクノロジーにはその他、遺伝病、がん、感染症の治療など、研究や個別化医療における多くの用途があります³。sgRNA は通常、固相オリゴヌクレオチド合成によって生成するため、その分析的特性解析では、インタクト分子量の確認および完全な配列確認を行います4,5。
sgRNA の消化産物マッピングの現行のワークフローは、手動データ分析を含む、手間と時間のかかる作業です。RNA 消化の戦略はまだ最適化されておらず、多くの特異的でないオリゴヌクレオチド消化産物が生成されるため、RNA 分子の配列確認にはほとんど役立ちません。2023 年に発表したアプリケーションノートで、RNase T1 消化後に sgRNA および mRNA の配列を自動的にマッピングするために開発した UPLC-MS・インフォマティクスワークフローについて説明しました6。 今回のこのアプリケーションノートでは、複数の消化酵素を独立した反応で使用して、累積的で特異的な消化産物割り当てによりシーケンスカバー率を高める、改良したワークフローについて詳細に説明します。今回、RNase T1、T2(RapiZyme MC1 およびクサチビン)、hRNase4 などの一連の酵素による消化産物のマッピングの結果を報告します。このワークフローのもう 1 つの大きな改善点は、消化済み RNA 生成物マップの迅速(3 分未満)かつ効率的な自動割り当てに使用する、新規の waters_connect アプリ(MAP Sequence)の導入によるものです。
実験方法
試薬およびサンプル前処理
ジプロピルエチルアミン(DPA、純度 99%、カタログ番号 D214752–500ML)および 1,1,1,3,3,3-ヘキサフルオロ-2-プロパノール(HFIP、純度 99%、カタログ番号 105228-100G)は、Sigma Aldrich(ミズーリ州セントルイス)から購入しました。メタノール(LC-MS グレード、カタログ番号 34966–1L)は Honeywell(ノースカロライナ州シャーロット)から入手しました。HPLC グレードのタイプ I 脱イオン水(DI)は、Milli-Q システム(Millipore、マサチューセッツ州ベッドフォード)を使用して精製しました。移動相は毎日新しく調製しました。sgRNA/mRNA 消化用のヌクレアーゼフリー超高純度水(カタログ番号 J71786.AE)は、Thermo Fisher Scientific(マサチューセッツ州ウォルサム)から購入しました。
HPRT1 酵素(ヒポキサンチンホスホリボシルトランスフェラーゼ 1)をコードする 10 nmol の 100 mer の sgRNA オリゴは、Integrated DNA Technologies(アイオワ州コーラルビル)から購入しました。sgRNA オリゴヌクレオチドの配列は 5’ - A*A*A* UCC UCA GCA UAA UGA UUG UUU UAG AGC UAG AAA UAG CAA GUU AAA AUA AGG CUA GUC CGU UAU CAA CUU GAA AAA GUG GCA CCG AGU CGG UGC U*U*U* U-3’ です。この RNA オリゴヌクレオチドには、最初の 3 つの 5' 末端ヌクレオチド(A*A*A*)および最後の 3' 末端ヌクレオチド(U*U*U*)が 2'-OMe 修飾されており、アスタリスク(*)でこれら 6 ヌクレオチドすべてがホスホロチオエート化されていることを示しています。sgRNA オリゴヌクレオチドのストック溶液は、濃度 5 µM になるように脱イオン水中に調製しました。
糸状菌由来アニマルフリー精製リボヌクレアーゼ T1(カタログ番号 IFGRNASET1AFLY 500KU)は、Innovative Research(ミシガン州ノバイ)から注文し、凍結乾燥酵素を 5 mL の 100 mM 重炭酸アンモニウム(カタログ番号 5.33005-50G、Millipore Sigma)に溶解して 100 単位/µL の溶液を調製しました。hRNase4 消化酵素(カタログ番号 M1284S)は、New England Biolabs(NEB、マサチューセッツ州イプスウィッチ)から購入しました。RapiZyme MC1(製品番号:186011190、10000 単位/チューブ)および RapiZyme クサチビン(製品番号:186011192、10000 単位/チューブ)の 2 つは、ウォーターズコーポレーションによって最近導入された新規の RNA 消化酵素です7。
RNase T1 を使用した sgRNA 消化では、5 µM sgRNA 2 µLを、ヌクレアーゼフリーの水 28 µL および RNase T1 酵素 10 µL と混合し、37 ℃ で 15 分間消化させました。消化混合液は、QuanRecovery MaxPeak 300 µL バイアル中に調製しました。この消化物を、LC-MS によって直ちに分析しました(5 µL 注入)。
hRNase4 を使用した sgRNA 消化では、3M 尿素中の 5 µM sgRNA 2 µL を 90 ℃ で 5 分間熱変性させ、続いて 25 ℃ に急速冷却しました。この変性 sgRNA を、4 µL の酵素バッファー(10X 濃度)、33 µL のヌクレアーゼフリー水および 1 µL の hRNase4 酵素(50 単位/µL)と混合して、37 ℃ で 60 分間消化させました。hRNase4 消化を 1 µL の NEB RNase 阻害剤(NEB カタログ番号 M0314S)により停止した後、室温で 10 分間インキュベーションしました。
RapiZyme MC1 とクサチビンに使用した消化プロトコルは非常に似ています。RapiZyme MC1 の場合、sgRNA(10 µL、2 ~ 5 µM 溶液)を、200 mM 酢酸アンモニウム(pH 8.0)を含むバッファー中で、90 ℃ で 2 分間変性させました。RapiZyme クサチビンの場合、sgRNA(10 µL、2 ~ 5 µM 溶液)を、200 mM 酢酸アンモニウム(pH 9.0)を含むバッファー中で 90 ℃ で 2 分間変性させました。いずれのサンプルも、氷上で冷却し、マイクロ遠心分離機でスピンして、サンプルの液滴を回収しました。50 単位の消化酵素(1 µL の RapiZyme MC1 またはクサチビン)とヌクレアーゼフリー水 8 µL を添加して最終容量を約 20 µL にした後、Eppendorf サーモミキサー中で sgRNA を 37 ℃ で 60 分間消化しました。70 ℃ に 15 分間さらして酵素を不活性化して酵素消化を停止させました。この消化物を、LC-MS によって直ちに分析しました(5 µL 注入)。
すべてのデータセットを、UNIFI™ 科学情報システムアプリのバージョン 3.6.0.21 を使用して取り込んだ後、mRNA Cleaver および Coverage Viewer MicroApp の助けを借りて、waters_connect インフォマティクスプラットフォーム内の MAP Sequence アプリを使用して解析しました。
LC 条件
|
LC-MS システム: |
ACQUITY™ Premier システムを搭載した Xevo™ G3 QTof LC-MS |
|
カラム: |
ACQUITY™ Premier Oligonucleotide BEH C18 FIT カラム 130 Å、1.7 µm、2.1 × 150 mm(製品番号:186009487) |
|
カラム温度: |
60 ℃ |
|
流速: |
300 µL/分 |
|
移動相: |
移動相 A:10 mM DPA(ジプロピルエチルアミン)、40 mM HFIP(1,1,1,3,3,3-ヘキサフルオロイソプロパノール)含有脱イオン水(pH 8.5) 移動相 B:10 mM DPA、40 mM HFIP 含有 50% メタノール |
|
サンプル温度: |
8 ℃ |
|
サンプルバイアル: |
QuanRecovery MaxPeak HPS バイアル(製品番号:186009186) |
|
注入量: |
5 µL |
|
洗浄溶媒: |
パージ溶媒:50% メタノール サンプルマネージャー洗浄溶媒:50% メタノール シール洗浄:20% アセトニトリル含有脱イオン水 |
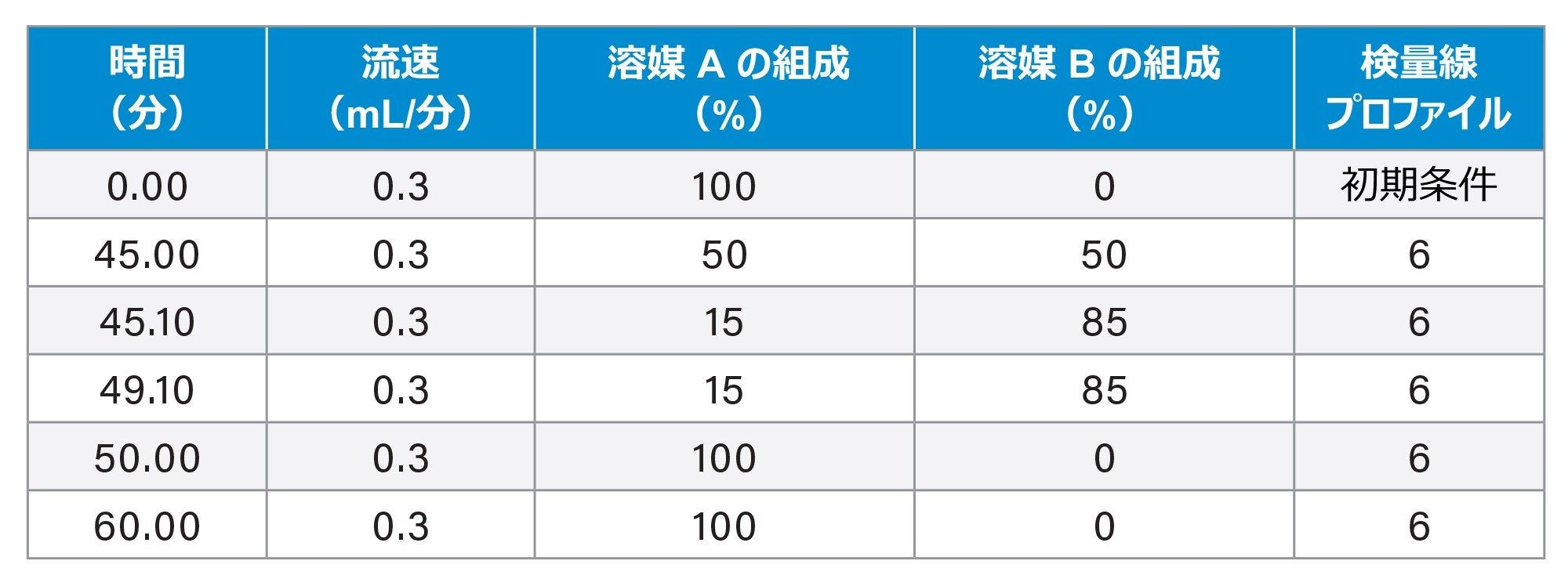
グラジエントテーブル
MS 条件
|
MS システム: |
Xevo™ G3 QTof 質量分析計 |
|
イオン化モード: |
ESI(-) |
|
取り込みモード: |
MSE |
|
取り込み速度: |
1 Hz |
|
キャピラリー電圧: |
2.5 kV |
|
コーン電圧: |
40 V |
|
イオン源オフセット: |
60 V |
|
イオン源温度: |
120 ℃ |
|
脱溶媒温度: |
550 ℃ |
|
コーンガス流量: |
50 L/時間 |
|
脱溶媒ガス流量: |
600 L/時間 |
|
ToF 質量範囲: |
340 ~ 4000(MSE 取り込み) |
|
低エネルギー CE: |
6 V |
|
高エネルギー CE ランプ: |
25 ~ 50 V |
|
ロックマス: |
50 pg/ µL Leu Enk |
|
データ取り込み: |
waters_connect 3.6.0.21 |
|
データ解析: |
waters_connect 3.6.0.21 |
|
データ解析: |
mRNA Cleaver 2.0 MAP Sequence v 1.0 Coverage Viewer v 2.0 |
結果および考察
RNA 配列確認のための LC-MS ベースのアプローチが、バイオ医薬品組織によってより一般的に採用されてきています。クロマトグラフィーの再現性と分離能の向上、高質量分解能 MS の使いやすさの向上とともに、MS システムの感度と精度の向上により、確実な配列確認と塩基修飾の検出が可能になっています。最も重要な点として、新しいオリゴヌクレオチドインフォマティクスツールにより、RNA マッピングのデータ分析という面倒な作業がはるかに簡単になりました。
ワークフローの特徴
図 1 に、さまざまなリボヌクレアーゼによって消化された sgRNA から取り込まれた LC-MSE データセットの解析のために導入したワークフローを示しています。最初のステップで、mRNA Cleaver MicroApp を使用して in-silico 消化したオリゴ生成物を生成します。ワークフローの 2 番目のステップでは、waters_connect MAP Sequence アプリを使用して、予測されるオリゴ消化産物を LC-MS データに自動的に割り当てます。図 1 のワークフローの最終ステップでは、複数の酵素で消化した sgRNA について得られたシーケンスカバー率が、Coverage Viewer MicroApp を使用してまとめられています。
図 2 に、修飾 sgRNA の RNase T1 消化に使用した設定を示す mRNA Cleaver MicroApp の GUI(グラフィカルユーザーインターフェース)の例を示しています。mRNA Cleaver MicroApp では、MazF、RNase A、hRNase4、Colicin E5 などのその他の酵素、およびウォーターズコーポレーションが最近導入した 2 つの酵素(RapiZyme™ MC1 および RapiZyme™ クサチビン)をデフォルトで使用できます。
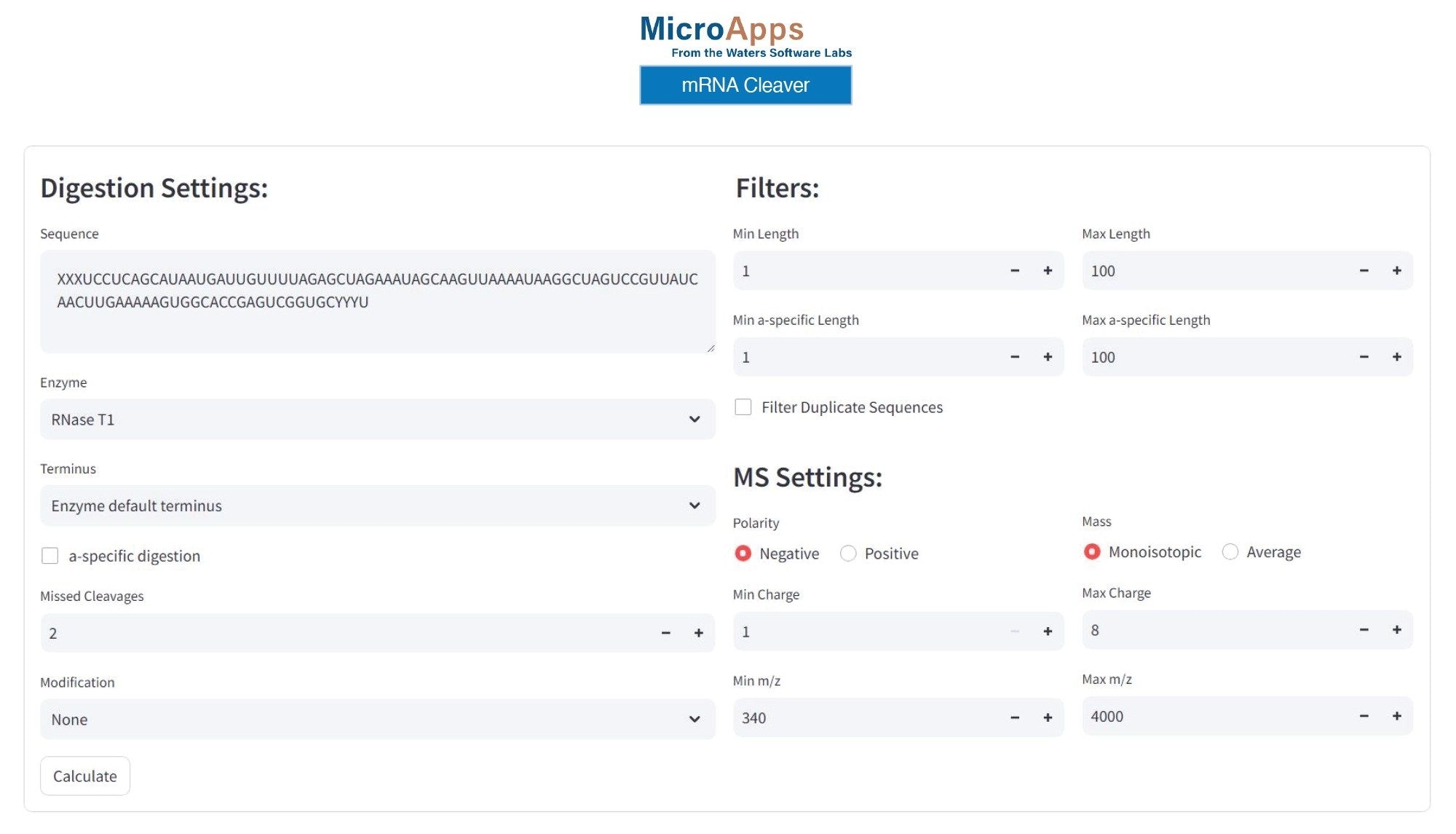
「実験方法」セクションに記載したように、RNA オリゴヌクレオチドには、図 2 に示す RNA 配列の最初の 3 つの 5' 末端ヌクレオチド(A*A*A*)に、XXX の文字で示した 2'-OMe 修飾およびホスホロチオエート化リンカーが含まれています。さらに、図 2 に示す配列中の文字 YYY で示した最後の 3 つの 3' 末端ヌクレオチド(U*U*U*)は、ホスホロチオエート化され、2'-OMe 官能基で修飾されています。図 2 に示した設定を使用して予測される in-silico 消化産物には、最大 2 つの RNase T1 酵素によるミスクリーベージ、および 3' 位置に直鎖状リン酸を含むオリゴ生成物の生成が想定され、生成物はわずか 1 ヌクレオチドと考えられました。修飾ヌクレオチドの追加は、mRNA Cleaver MicroApp で直接サポートされています。
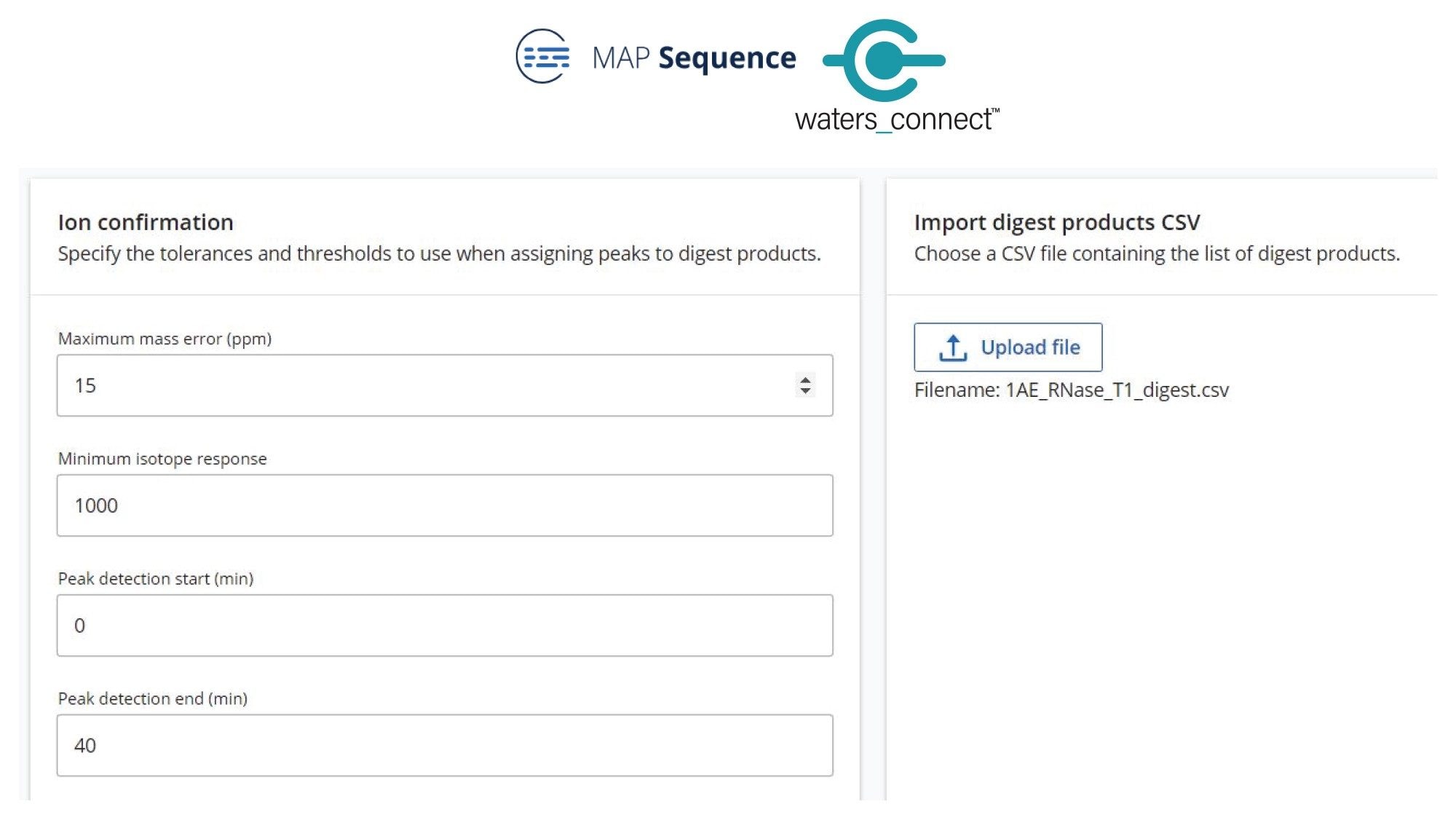
MAP Sequence アプリ(図 3)のデータ解析に必要なシンプルな解析パラメーターには、クロマトグラフィー保持時間(RT)の範囲、最小同位体レスポンス、予測オリゴ生成物と実測オリゴ生成物を関連付けるために使用する質量精度許容値が含まれます。解析後に得られた MAP Sequence 結果の一部を、図 4 内に示しています。実験的に測定したモノアイソトピックオリゴヌクレオチド生成物を予測モノアイソトピック質量に対してマッチングしたところ、一意(緑色の円)および非特異的な(黄色の三角形)オリゴ生成物の割り当てが得られました。
酵素のパネルからのマッピングの結果
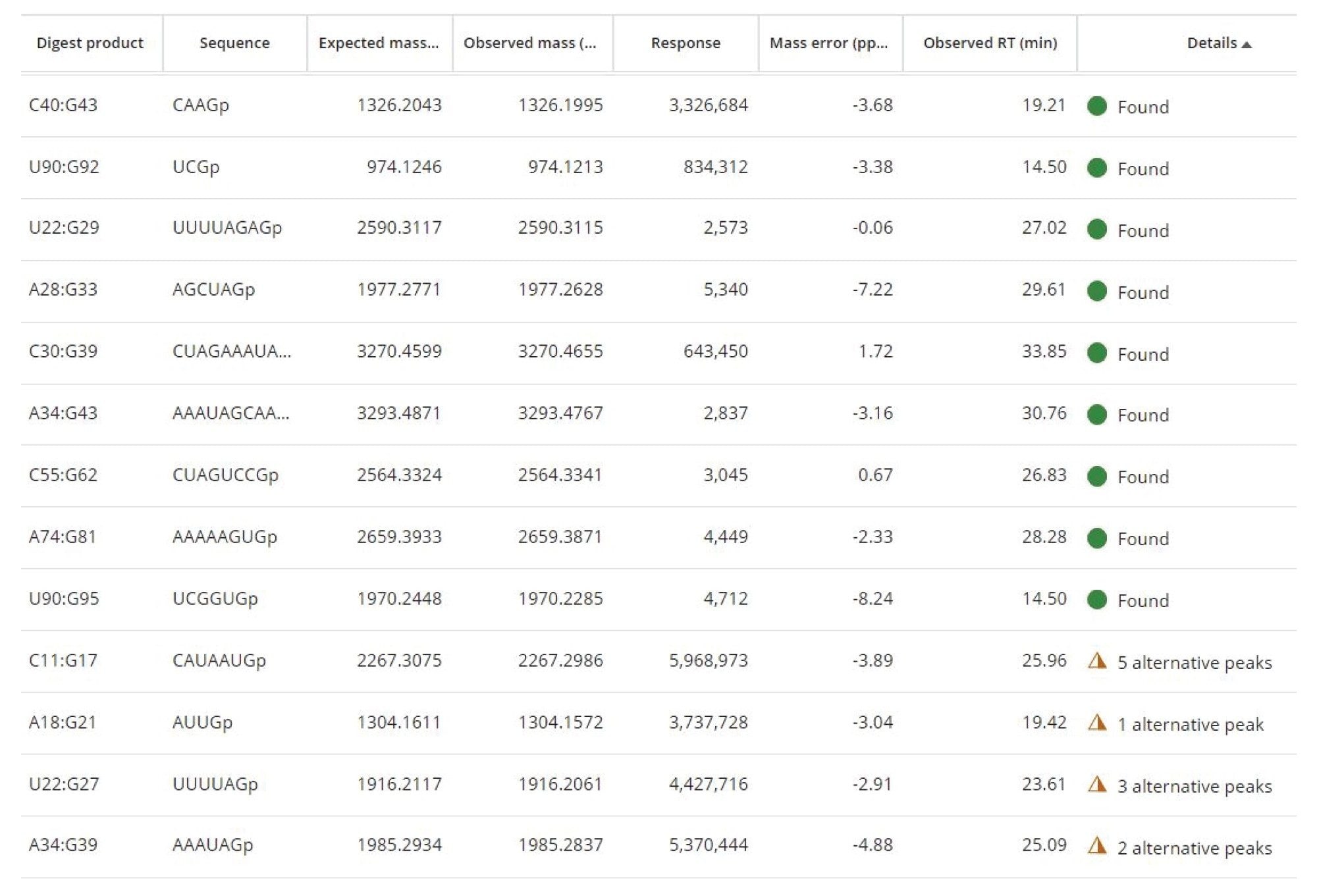
RNase T1 sgRNA 消化について記録されたトータルイオンクロマトグラム(TIC)を図 5 に示します。mRNA Cleaver MicroApp は、末端のグアノシン(G)残基を含むオリゴ生成物を生成する可能性のある酵素切断をすべて想定して、この図にリストされている 22 種のオリゴ生成物を予測しています。この図に示すように、黄色でラベル付けしたクロマトグラフィーピークで示した 3 対の同一のオリゴ生成物(配列:AG - ピーク 5/18、UG - ピーク 15/21 および CUAG - ピーク 6/11)があり、これらによって配列割り当てが非特異的になっています。この欠点が、RNase T1 の限界を示しています。つまりこの酵素には、100 mer sgRNA のような比較的短い基質の場合でも、完全に一意の生成物を生成するのに必要な特異性がありません。すべての G 残基の後で切断が起きると(RNA 中に非修飾ヌクレオチドが 4 つしか存在しないことを考慮すると、理論的な切断の確率は 25%)、RNA 配列の長さが増加するにつれて、同一のオリゴ生成物の配列を持つ可能性が高い予測オリゴ生成物が明らかに多くなりすぎます。
図 5 に、MAP Sequence 割り当ての結果をまとめています。左側に示した sgRNA 配列内で、一意のオリゴ生成物に属するすべてのクロマトグラフィーピークを緑色でラベル付けし、非特異的な配列を黄色でラベル付けし、欠落している(マッチしない)生成物を赤色でリストしています。このクロマトグラム中に検出された存在量が多いクロマトグラフィーピークのほとんどは、予測生成物に割り当てられています。その他の唯一の非特異的な生成物(上記の 3 つの同一の対以外)は、最初(5' 末端)のオリゴ生成物(OMEA* OMEA* OMEA* UCC UCA G)です。これには、最初の 3 ヌクレオチドのホスホロチオエートリンカー中に硫黄のキラル中心が存在するために、複数のジアステレオマーが含まれる可能性があります。唯一欠落している(マッチしない)オリゴヌクレオチド生成物は、使用する IP -RP 条件下でカラムに保持されない単一の G 残基(生成物番号 10、16、20)です。このシングルヌクレオチドの G 残基は、sgRNA の 100 nt 組成全体の LC-MSE データセットのうち唯一検出されなかった残基であり、最大 2 箇所の RNase T1 ミスクリーベージを含めてもこの結果は改善されませんでした。ただし、特筆すべき点として、図 5 に示すように、DPA イオン対試薬により、2 つのジヌクレオチド分子種(UG - 15/21 の対と AG - 5/18 の対)の分離が可能になりました。
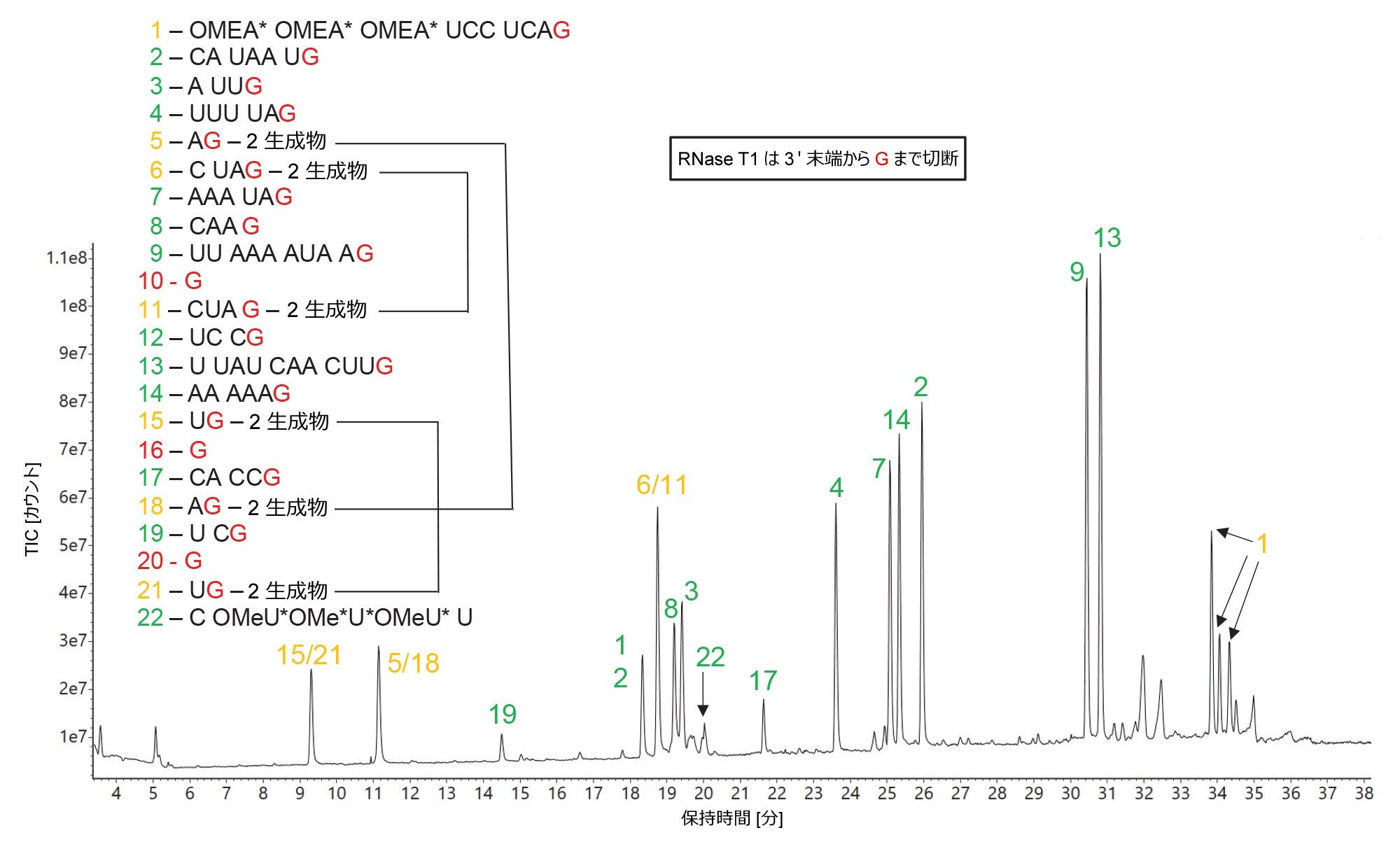
HPRT1 sgRNA を、RNase T1 消化に加えて、最近製品化された酵素 hRNase4 で個別に消化しました7。 RNase T1 とは異なり、hRNase4 はウリジン(U)エンドオリボヌクレアーゼであり、プリン(A および G)残基が後に続く場合にのみ、U 残基の後で切断します。hRNase4 の 2 つの切断部位は、mRNA Cleaver MicroApp で U_A および U_G と定義されており、その RNA 基質切断の理論的確率は 12.5% と算出され、RNase T1 の理論的切断確率の半分です。この結果の例は、同じ sgRNA 基質の hRNase4 消化について記録された図 6 の TIC クロマトグラムに見られます。予測オリゴ生成物の数は、RNase T1 消化と比較して大幅に少なく(14 対 22)、ランダムな 100 mer RNA 配列の切断について予測されるオリゴ生成物の予想数(12 ~ 13 生成物)とよく一致しています。RNase T1 の場合と同様に、同一の予測配列はありませんが、hRNase4 では 1 対の 6 mer の構造異性体(ペア 5/9、AG AGC U と A AGG CU)が生成しており、これが非特異的な配列割り当ての原因でした。これは、使用した IP-RP 条件(RNase T1 消化物の分離に使用した条件と同じ)では、これらがクロマトグラフィー分離されなかったためです。この非特異的な割り当ては、クロマトグラフィー条件を調整して 2 つの 6 mer オリゴ生成物を分離し、(高エネルギー MSE フラグメンテーションデータに記録されている)対応するフラグメントを使用して waters_connect CONFIRM Sequence アプリで正しい配列を割り当てることで解決することができます8。
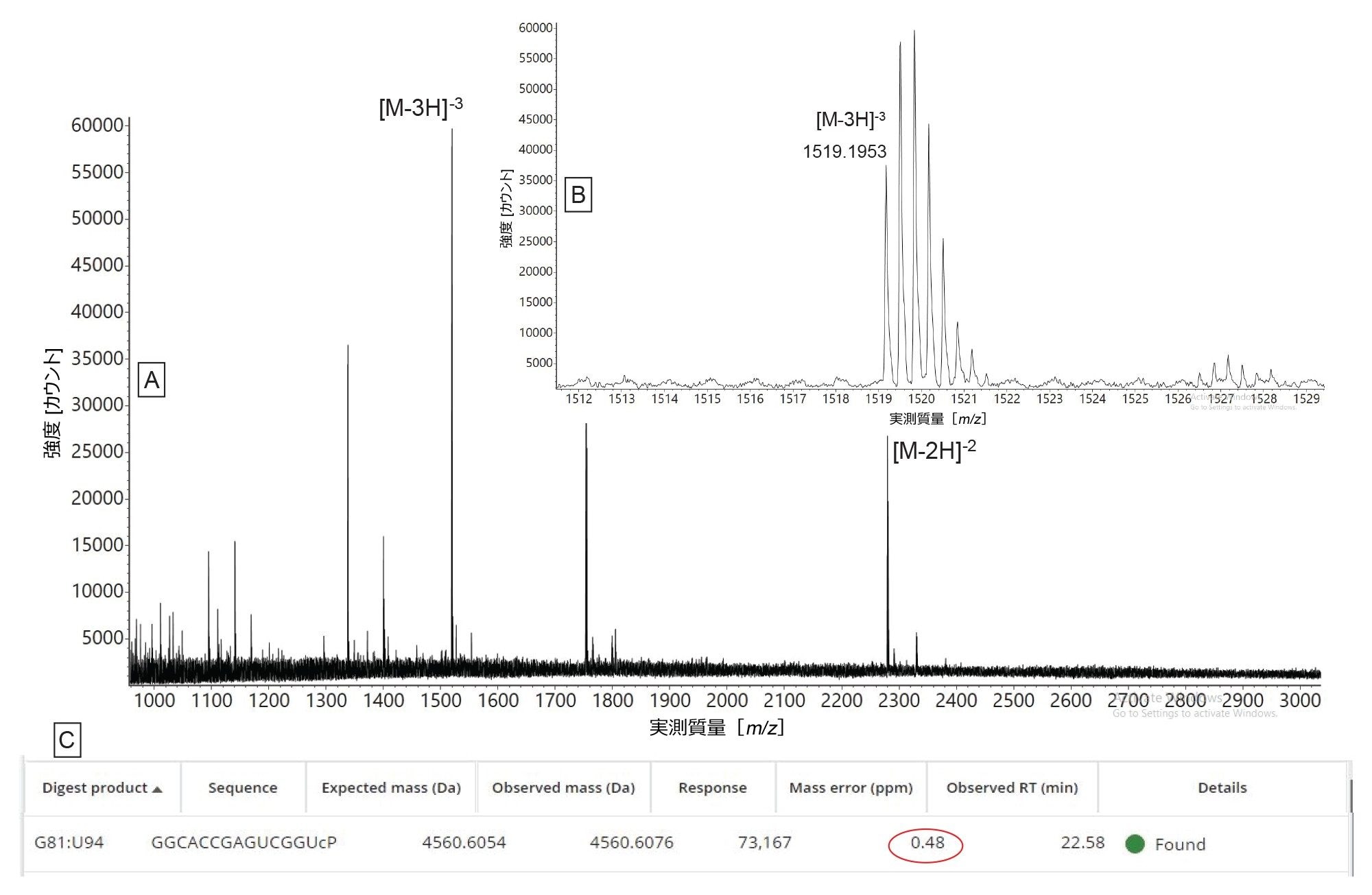
その他の唯一の非特異的な割り当ては、最初の 5' 末端のオリゴ生成物(OMEA* OMEA* OMEA* UCC UCA GCA U)に対応する、遅く溶出するピークダブレットによって生じたもので、これも複数のキラル中心が存在するために不均一性を示しました。低存在量のオリゴ生成物による ESI-MS スペクトル(最も存在量の多い生成物による ESI-MS スペクトルの強度と比較して存在量 0.6%)を図 7 に示します。このスペクトル中に検出された二価および三価のオリゴプリカーサーにより、14 mer の予測オリゴ生成物(配列:G GCA CCG AGU CGG U)の存在が確認されました。この生成物は、MAP Sequence でイオン同位体レスポンスのしきい値を 1000 カウントに下げることで検出されました。RNase T1 と hRNase 4 の生物学的活性に関して最後に付け加えると、いずれの酵素も優先する残基(G/U 残基の 3' 側)の後で切断し、RNase T1 は消化オリゴ生成物の 3' 末端に直鎖状のリン酸を付加するのに対して、hRNase4 は、ソフトウェアで検出された 3' 直鎖状および環状のリン酸の両方をほぼ同レベルで含む消化産物を生成します。RNase T1 と hRNase4 はいずれも、TIC クロマトグラムに未消化の sgRNA が存在する徴候がないことから、RNA 基質を完全に切断します(データは示していません)。
RapiZyme MC1 と RapiZyme クサチビンの 2 つは、ウォーターズコーポレーションによって最近導入された新しい酵素です。MC1 はウリジンに対する特異性が高く、クサチビンはシチジンに対する特異性が高いという特徴があります9,10。
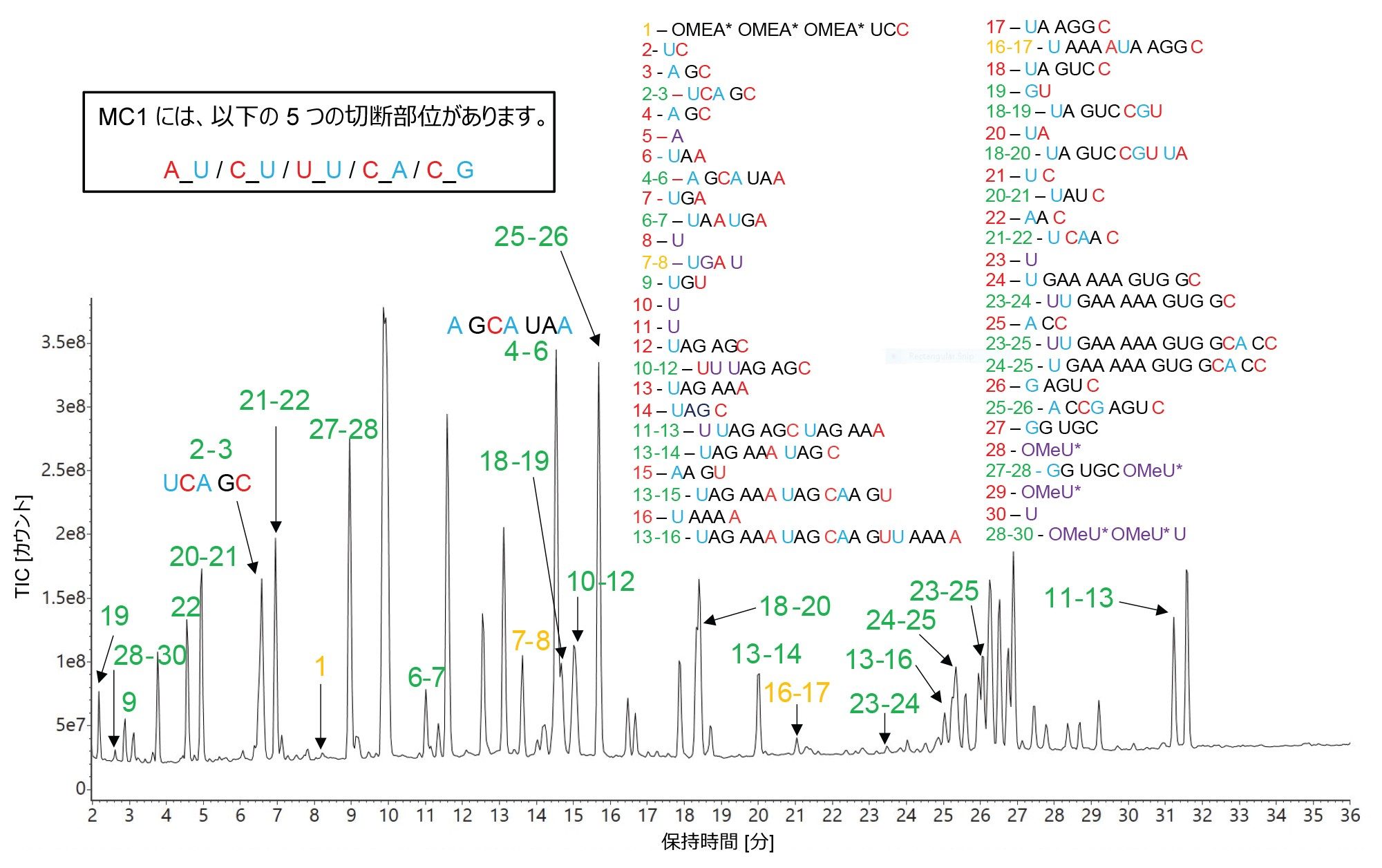
HPRT1 sgRNA 基質の MC1 消化物の LC-MS 分析で得られた TIC クロマトグラムを、MAP Sequence 解析後に得られた対応する配列割り当てとともに、図 8 に示します。mRNA Cleaver アプリにより、MC1 の 5 つの予想切断部位(A_U / C_U / U_U / C_A / C_G)の結果として、また最大 2 箇所のミスクリーベージも考慮して、比較的多数の予測オリゴ生成物が生じました。予測された短いオリゴ生成物(いくつかのシングルヌクレオチドとジヌクレオチドを含む)の多くは実験データでは検出されず、割り当てられたシーケンスカバー率のほとんどに、ミスクリーベージを含むオリゴヌクレオチド生成物が使われていました。これは、トリプシン(高特異性)とペプシン(低特異性)によるタンパク質の消化に似ています。前者ではよりシンプルなペプチドマップが得られ、後者ではオーバーラップしたシーケンスカバレッジを持つマップの可能性を生成します。
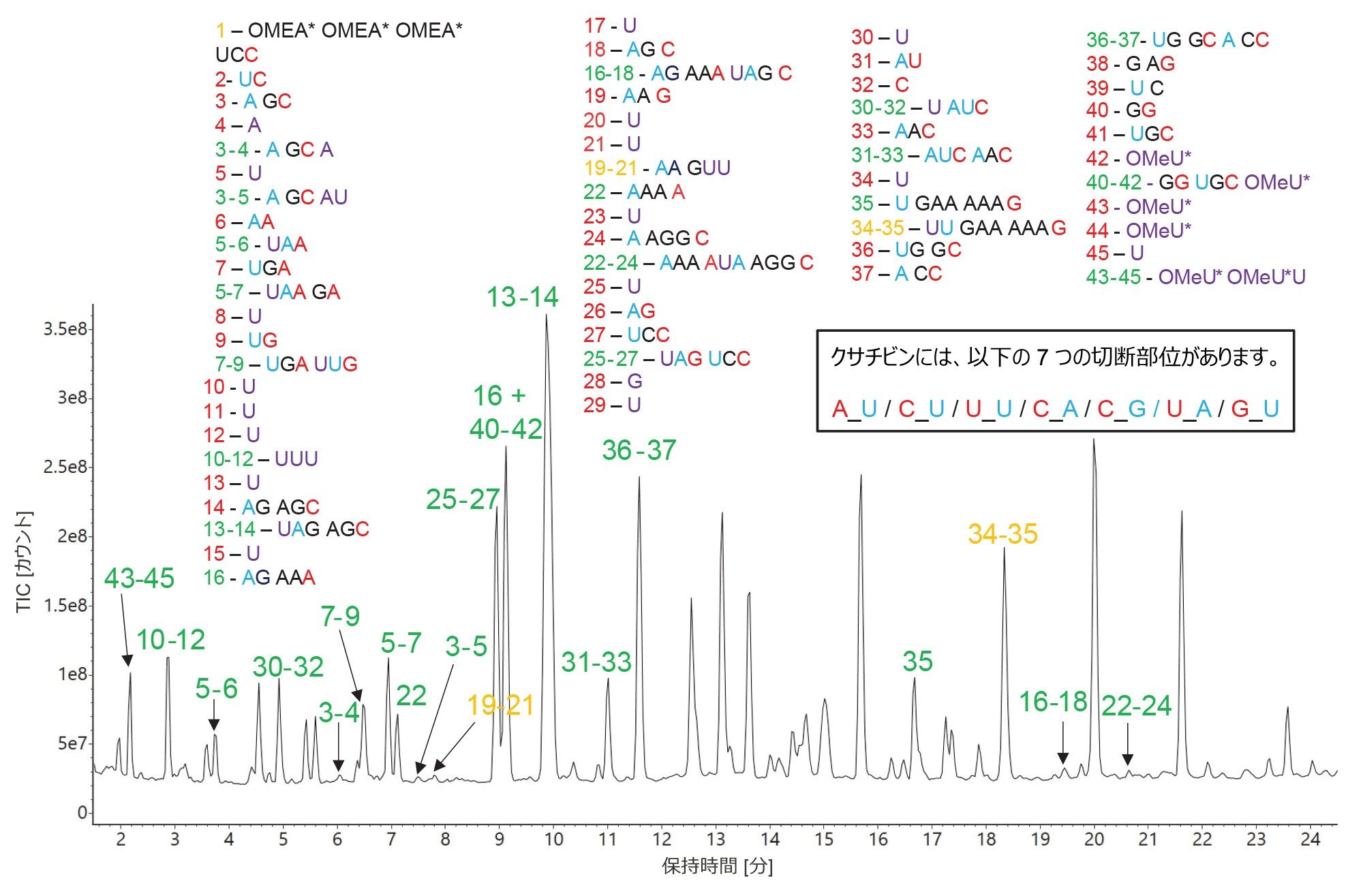
MC1 は、RNase T1 および hRNase4 と異なり、特定のターゲットの前(U 残基の 5' 末端)で切断し、主に環状リン酸を消化されたオリゴの 3' 末端に付加することも付け加えておきます。クサチビンは主にシチジンの後(C 残基の 3' 末端)で切断しますが、他にもマイナーな切断部位が 4 つあります。全体として、クサチビンには 7 種類の切断部位が予想され、これらは mRNA Cleaver MicroApp で C_U / C_A / C_G / U_U / A_U / U_A / G_U と定義されています。MC1 と同様、クサチビンは主に各オリゴ消化産物の 3' 末端に環状リン酸を付加しますが、ソフトウェアでは両方のフォーム(環状および直鎖状)が検出されました。
多くの割り当てられたオリゴヌクレオチド生成物にミスクリーベージが含まれることから、クサチビンの特異性が、HPRT1 sgRNA 消化の TIC クロマトグラムに反映されていることがわかります(図 9)。ミスクリーベージのない対応する生成物は短すぎて(シングルヌクレオチド、ジヌクレオチドまたはトリヌクレオチド)IP-RP UPLC で分離するのは困難であることから、検出されない可能性のある sgRNA 配列の部分をカバーするために、ミスクリーベージの数を比較的多くすることには明らかなメリットがあります。MC1 とクサチビンはいずれも、TIC クロマトグラムに未消化の sgRNA の徴候がないことから、RNA 基質を完全に切断します(データは示していません)。RapiZyme MC1 および RapiZyme クサチビンに焦点を当てた別のアプリケーションノートでは、これらのユニークな酵素およびその RNA 消化への利用の詳細について記載しています11。
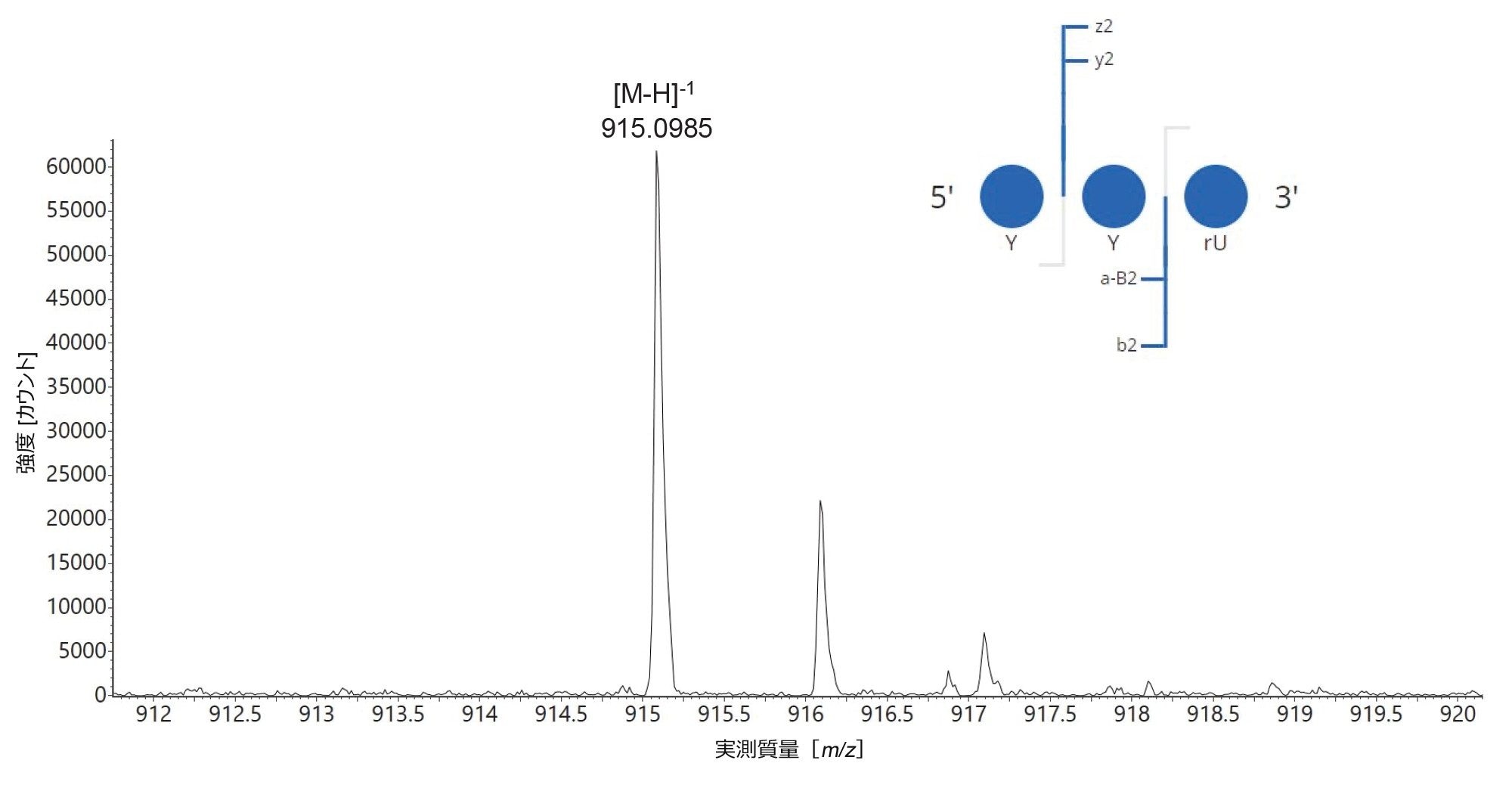
上記の結果に基づいて、これらの酵素の完全なシーケンスカバー率を得る試みとして、MC1 およびクサチビンのミスクリーベージの数を増やし、最大 3 箇所のミスクリーベージを許容しました。さらに、mRNA Cleaver で定義された切断部位を拡張し、sgRNA 配列中に存在するいくつかの修飾残基の周囲の切断を含めました。例えば、MC1 の場合、3 つの切断部位(C_Y / Y_Y / Y_U)を追加しました(Y は 2'-OMe ホスホロチオエート化ウリジンを示します)。これらを行った結果、配列 YYU を含む新規の予測オリゴ生成物が、MAP Sequence 解析によって両方の消化物中に検出されました。この生成物のアイデンティティーは、CONFIRM Sequence アプリを使用して高エネルギーフラグメンテーションスペクトルを解析することで確認されました8。 その一価プリカーサーの同位体分布を、CONFIRM Sequence アプリによって生成されたドットマップダイヤグラムとともに、図 10 に示します。このトリヌクレオチドの配列を検証したところ、MC1 とクサチビンはいずれも修飾されたウリジン残基の間で切断することが示されました(Y_Y モチーフの切断)。これはユニークで有用な酵素機能です。hRNase4 酵素は、修飾されたウリジンの後で切断できませんが、MC1 とクサチビンは、2' ウリジン修飾の影響を受けないため、一意の消化産物を生成することができます7。
RNA 分析ワークフローの最終ステップ(図 1)で、Coverage Viewer MicroApp を使用して、RNase T1、hRNase4、RapiZyme MC1 および RapiZyme クサチビンで消化した同じ sgRNA について得られたシーケンスカバー率をまとめました(図 11)。RNase T1 および hRNase4 について個別に得られた一意のシーケンスカバー率は 80 ~ 90% の範囲ですが、MC1 消化およびクサチビン消化では、この分子について 100% のカバー率が得られました。全体として、これらの結果により、複数の酵素の結果を組み合わせることで、オリゴ生成物のマッピングにおける信頼性が高まると言えます。

高分子 RNA(sgRNA、mRNA)の配列確認における質量分析の使用については、依然として積極的にワークフロー開発が行われています。これらの複雑なデータセットを解析するためのソフトウェア自動化の開発、および今回検討したより大規模な消化酵素ツールセットの開発は、この新しい分析法の有用性を高めるための重要な要素になると考えられます。
結論
- 新しいソフトウェアツールによって、sgRNA の複数の酵素消化物から取得した UPLC-MS データを使用して、効率的な RNA 消化産物マッピングを行えることが実証されました。この新しい RNA 消化産物マッピングアプリケーション(MAP Sequence)を使用して、各酵素によって得られた sgRNA 消化データを正常に解析して生成物マップを生成し、予測配列を確認できるようになりました。
- 2 つの新規の RNase T2 酵素(RapiZyme MC1 および RapiZyme クサチビン)では、そのユニークな消化特異性により、より多数のオリゴ消化産物とミスクリーベージが生じます。これらの酵素は、重複する配列の領域を含むオリゴ生成物のマップを得るのに役立つ可能性があります。
- すべての酵素について高いシーケンスカバー率が得られ、MC1 およびクサチビンでは、この分子のオリゴ消化産物のレベルで 100% の配列確認が得られました。実際には、すべての sgRNA 分子、またより大きな mRNA 配列でこれが得られるわけではありません。酵素消化のパネルから得られる結果を組み合わせることにより、質量フィンガープリントベースのアプローチの正確さに対する信頼性が全体的に高まり、RNA ベースの医薬品について、完全なシーケンスカバー率が得られる可能性が最大限に高まります。
- オリゴマッピングは現在主に開発で使用されていますが、規制対応の waters_connect インフォマティクスプラットフォームの、統合された UPLC-MS データ取り込みワークフローおよびデータ解析ワークフローにより、これらのワークフローを製造や品質管理部門に活用できる可能性があります。
参考文献
- Jinek M, Chylinsky K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A Programmable Dual-RNA-Guided DNA Endonuclease in Adaptive Bacterial Immunity, Science, 2012, 337, 816–821.
- Jiang F, Doudna JA.CRISPR-Cas9 Structures and Mechanisms, Annu Rev Biophys, 2017, 46, 505–529.
- Ganbaatar U, Liu C. CRISPR-Based COVID-19 Testing: Toward Next Generation Point of Care Diagnostics, Front Cell Infect Microbiol, 2021, 11.https://doi.org/10.3389/fcimb.2021.663949
- Catalin E. Doneanu, Patrick Boyce, Henry Shion, Joseph Fredette, Scott J. Berger, Heidi Gastall, Ying Qing Yu.LC-MS Analysis of siRNA, Single Guide RNA and Impurities using the BioAccord System with ACQUITY Premier System and New Automated INTACT Mass Application.Waters Application Note.720007546.2022
- Goyon A, Scott B, Kurita K, Crittenden CM, Shaw D, Lin A, Yehl P, Zhang K. Full Sequencing of CRISPR/Cas9 Single Guide RNA (sgRNA) via Parallel Ribonuclease Digestions and Hydrophilic Interaction Liquid Chromatography High-Resolution Mass Spectrometry Analysis, Anal Chem, 2022, 93, 14792–14801. doi: 10.1021/acs.analchem.1c03533
- Rebecca J. D'Esposito, Catalin E, Doneanu Heidi Gastall, Scott J. Berger, Ying Qing Yu.RNA CQA Analysis using the BioAccord LC-MS System and INTACT Mass waters_connect.Waters Application Note.720008130.2023.
- Wolf EJ, Grunberg S, Dai N, Chen T-H, Roy B, Yigit E, Correa IR.Human RNase 4 Improves mRNA Sequence Characterization by LC-MS/MS, Nucleic Acid Res, 2022, 50, e106. DOI:10.1093/nar/gkac632
- Catalin E. Doneanu, Chris Knowles, Matt Gorton, Henry Shion, Joseph Fredette, Ying Qing Yu.CONFIRM Sequence: A waters_connect Application for Sequencing of Synthetic Oligonucleotide and Their Impurities.Waters Application Note.720007677.2022.
- Grunberg S, Wolf EJ, Jin J, Ganatra MD, Becker K, Ruse C, Taron CH, Correa IR, Yigit E. Enhanced Expression and Purification of Nucleotide-specific Ribonucleases MC1 and Cusativin, Protein Expr Purif Acid Res, 2022, 190, 105987.doi:10.1016/j.pep.2021.105987
- Thakur P, Atway J, Limbach PA, Addepalli B. RNA Cleavage Properties of Nucleobase-Specific RNase MC1 and Cusativin Are Determined by the Dinucleotide-Binding Interactions in the Enzyme-Active Site, Int J Mol Sci, 2022, 23, 7021.
- Balasubrahmanyam Addepalli Tatiana Johnston, Christian Reidy, Matthew A. Lauber.Tunable Digestions of RNA Using RapiZyme™ RNases to Confirm Sequence and Map Modifications.Waters Application Note.720008539.2024.
720008553JA、2024 年 9 月