本应用纪要使用离子淌度高分辨率质谱法对多种血清样品进行了分析。使用软件包Mass-MetaSite和WebMetabase处理数据,自动检测并显示肽分解代谢物。通过结合具备高准确度和高选择性的HDMSE数据采集方法(离子淌度增强型HRMS数据非依赖性采集),有助于可靠地鉴定和追踪分解代谢物。
肽类化合物具有许多引人注目的属性,可作药用,但其药理性质(例如,渗透性和稳定性)往往较差,因此在开发方面存在较大挑战。为克服这些局限性,研究人员向肽类化合物中引入更复杂的结构和非天然修饰,从而改善其结构并增强药用功能。
在药物开发过程中,务必要鉴定这些肽的分解代谢物,以便了解清除率和代谢物去向,尤其是独特的人体分解代谢物更为需要。实现可靠鉴定在分析和计算方面具有挑战性;即使简单的序列也包含许多可水解的键,从而产生数百种潜在的分解代谢物。对于体外或体内研究,各种氨基酸或经过结构设计的肽片段还可能存在其他转化途径,例如氧化、脱酰胺、偶联和解离。使用电喷雾法LC-MS分析采集的肽具有多电荷性质,可产生大量不同m/z值的碎片,这些全都需要经过筛选才能确保鉴定出所有分解代谢物。以上所有因素均增加了肽分解代谢物鉴定的复杂性。
生长抑素是一种天然存在的肽激素,具有生长抑制作用,一直以来都是众多研究的重点。Riera等人发表的文章中详细描述了生长抑素以及多种序列修饰在人血清中对稳定性的影响3。 本应用纪要使用离子淌度高分辨率质谱法对多种这类血清样品进行了重新分析。
使用软件包Mass-MetaSite和WebMetabase (Molecular Discovery Ltd.)处理数据,自动检测并显示肽分解代谢物。通过结合具备高准确度和高选择性的HDMSE数据采集方法(离子淌度增强型HRMS数据非依赖性采集),有助于可靠地鉴定和追踪分解代谢物4。
使用在英国运行WebMetabase的集中式服务器对研究进行分析,该服务器使英国和美国的多个全球用户和实验室可以同时上传、处理、查看数据,并可用作数据集的单一资源库。此外,经证明,Webmetabase的HELM notation (Pistoia Alliance)功能非常强大,能够显示复杂的肽分解代谢物并实现碎片鉴定。
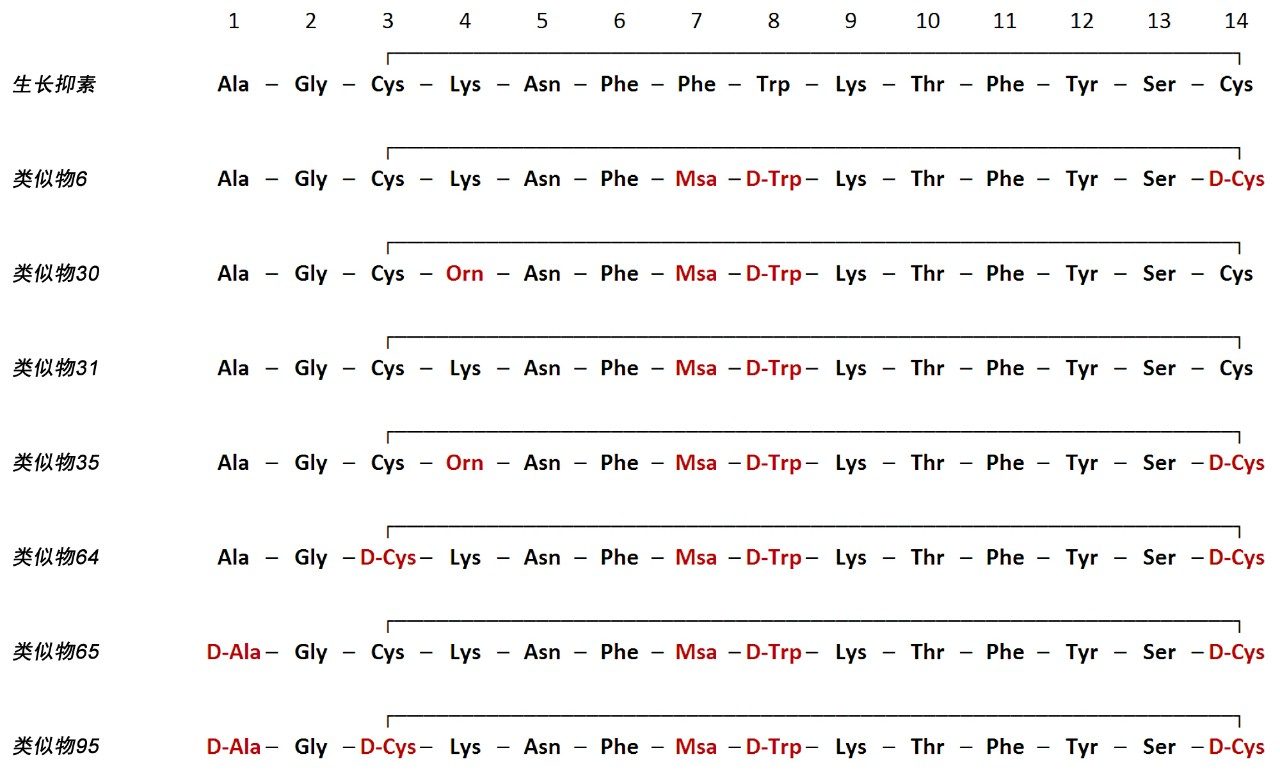
对8种含14个氨基酸的生长抑素类似物进行了研究(图1)。将化合物置于人血清中孵育11个时间点(0 min、5 min、10 min、30 min、1 h、2 h、4 h、8 h、24 h、30 h和48 h),评估稳定性和主要代谢物的形成。样品由西班牙巴塞罗那科学技术研究院生物医学研究所Antoni Riera教授的实验室提供。
|
系统: |
ACQUITY UPLC I-Class |
|
样品瓶: |
聚丙烯样品瓶,300 μL[部件号:186002628] |
|
色谱柱: |
ACQUITY CSH C18, 1.7 μm, 2.1 × 100 mm[部件号:86005297] |
|
柱温: |
45 °C |
|
样品温度: |
8 °C |
|
进样体积: |
1 μL |
|
流速: |
0.4 mL/min |
|
流动相A: |
水+0.1%甲酸 |
|
流动相B: |
乙腈+0.1%甲酸 |
|
梯度: |
流动相B在7 min内从2%增加至30%,然后在第8 min增加至100%,保持至第10 min |
|
系统: |
Vion IMS QTof质谱仪 |
|
电离模式: |
ESI正离子模式 |
|
采集模式: |
HDMSE(使用CCS) |
|
采集范围: |
m/z 50~2000 |
|
毛细管电压: |
1.0 kV |
|
碰撞能量: |
低能量6 eV,高能量15~55 eV递增 |
|
扫描时间: |
0.2 s |
启用API(应用程序接口)的UNIFI 1.9.4
Mass-MetaSite 3.4.2、WebMetabase 4.0 (Molecular Discovery Ltd.)
常规数据,启用TEC(两种碰撞能量)模式
监测的代谢物生成反应:酰胺水解、氧化脱酰胺、二硫化物还原
质量数,MS峰,模式筛选,容差(%):30
质量数,代谢物鉴定,代谢物生成:2
质量数,代谢物鉴定,底物键断裂限值:1
质量数,代谢物鉴定,分解代谢物:真,1
TEC,质谱仪:Waters QTof质谱仪
TEC,算法阈值:色谱图/MS/MSMS筛选:0.95/0.95/0.95
TEC,信号阈值:40
外部LC-MS文件转换器:2.1.9版UNIFI msvc64
HDMSE数据由运行UNIFI的Vion IMS QTof质谱仪进行采集,该软件具有完全集成的自动化CCS报告功能。使用Mass-MetaSite处理数据,并使用MetaSite批处理器将数据上传至WebMetabase进行显示1。
研究的所有类似物均包含Msa对Phe[7]的取代以及D-Trp对Trp[8]的取代。评估Ala[1]、Cys[3]和Cys[14]被其D-氨基酸等效物取代以及Lys[4]被鸟氨酸取代时各种排列的稳定性。对于所有类似物,利用WebMetabase监测底物的消失情况、评估关键代谢物形成(尤其是-Ala和-AlaGly)的调节情况并监测其他主要的代谢途径(包括开环)。之前曾有文献报道,天然生长抑素的代谢主要表现为分子尾部失去Ala (-71 Da)和-AlaGly (-128 Da)2,3。
WebMetabase中的一个代表性示例(肽31)在48 h孵育过程中被大部分清除。通过在Chromatogram(色谱图)概览(图2A)和Compounds(化合物)概览(图2B)之间进行切换,用户可以访问汇总的叠加色谱图、RT值、m/z值、与母体底物的质量数差异、MS峰面积/响应以及整个样品组或各组分中单个物质的底物实测范围的百分比。CCS值也直接集成在Mass-MetaSite和WebMetabase中。离子淌度法可用于谱图净化,去除无关离子,并确保追踪的峰不仅具有相同的m/z,而且具有相同的CCS。此法在发生峰漂移或同分异构体峰相邻洗脱的情况下特别有用。
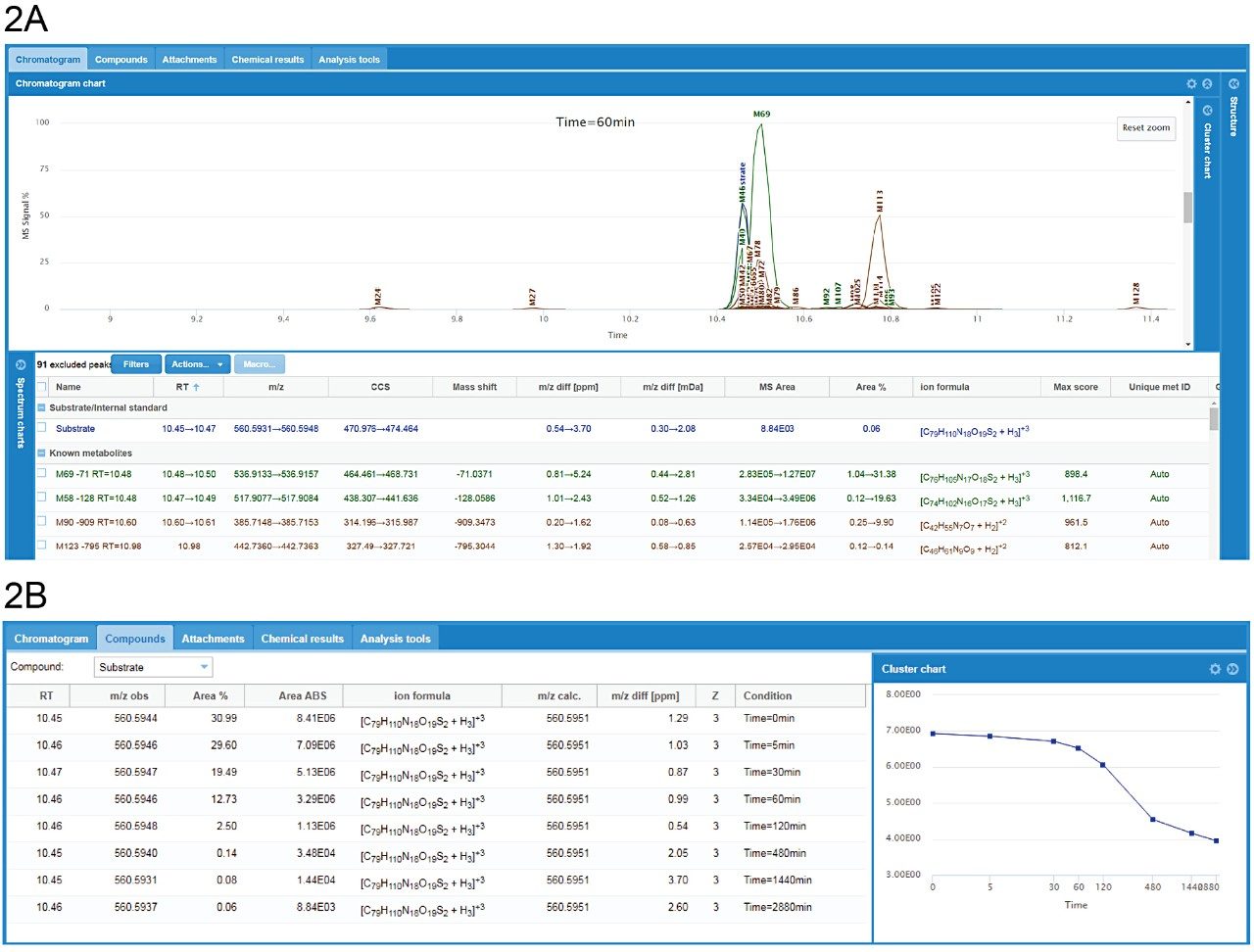
WebMetabase可直观显示所有检出的代谢物以及对所有鉴定出的代谢物进行评估的评分算法。主要代谢物及其结构如图3A所示。代谢物预测得分显示在各结构的左上角(均高于800),并提供了匹配和不匹配谱图鉴定的相应数量。图3B展示了这些化合物的响应-时间曲线。从曲线图可以看到,10 min后开始形成-71 (-Ala)和-128 (-AlaGly)代谢物,其含量在30 min和60 min之间达到最高水平。60 min后,可以看到开始形成后续的开环产物Asn-Phe-Msa-D-Trp-Lys-Thr和Phe-Msa-D-Trp-Lys-Thr(分别缩写为NF-Msa-dWKT和F-MSa-dWKT)。

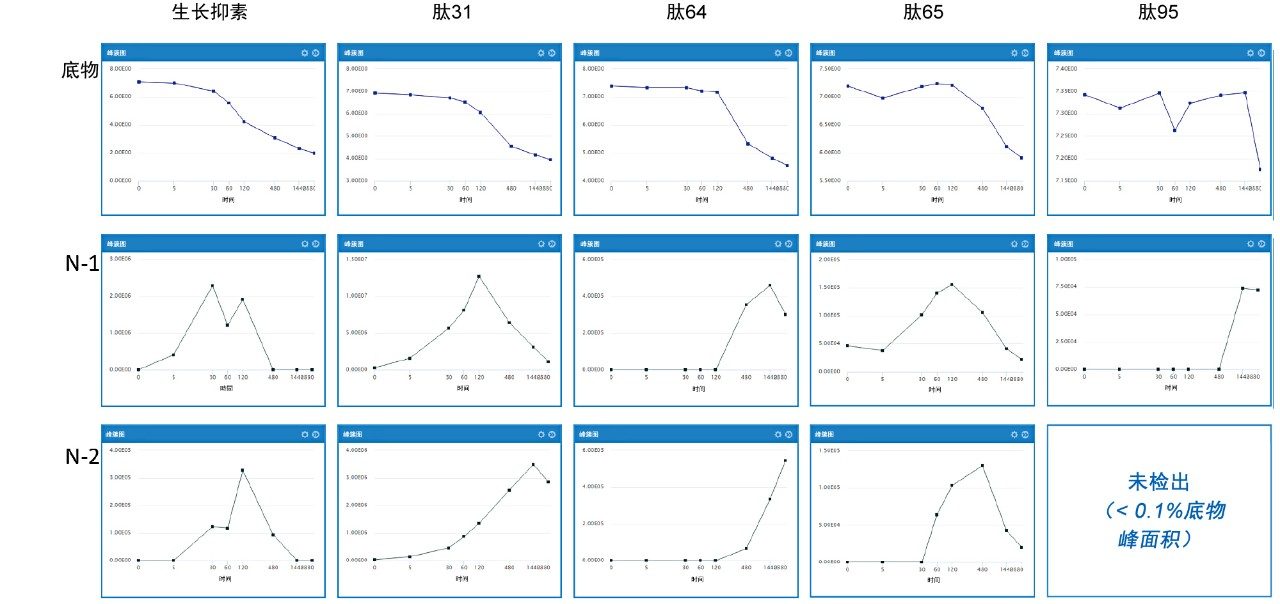
针对所有底物绘制了生长抑素以及四种经Msa/D-Trp修饰的肽段(31、64、65和95)的响应图,并对N-1、N-2分解代谢物损失进行了详细比较(图4)。大部分代谢可归因于失去N-1和N-2氨基酸(-Ala和-AlaGly)。末端丙氨酸或桥接的半胱氨酸分子经过修饰后,清除率差异非常大。底物的时间/响应曲线以及主要的N-1和N-2代谢物展示了药物的消失情况及其与主要代谢物出现(或消失)的相关性(图4)。
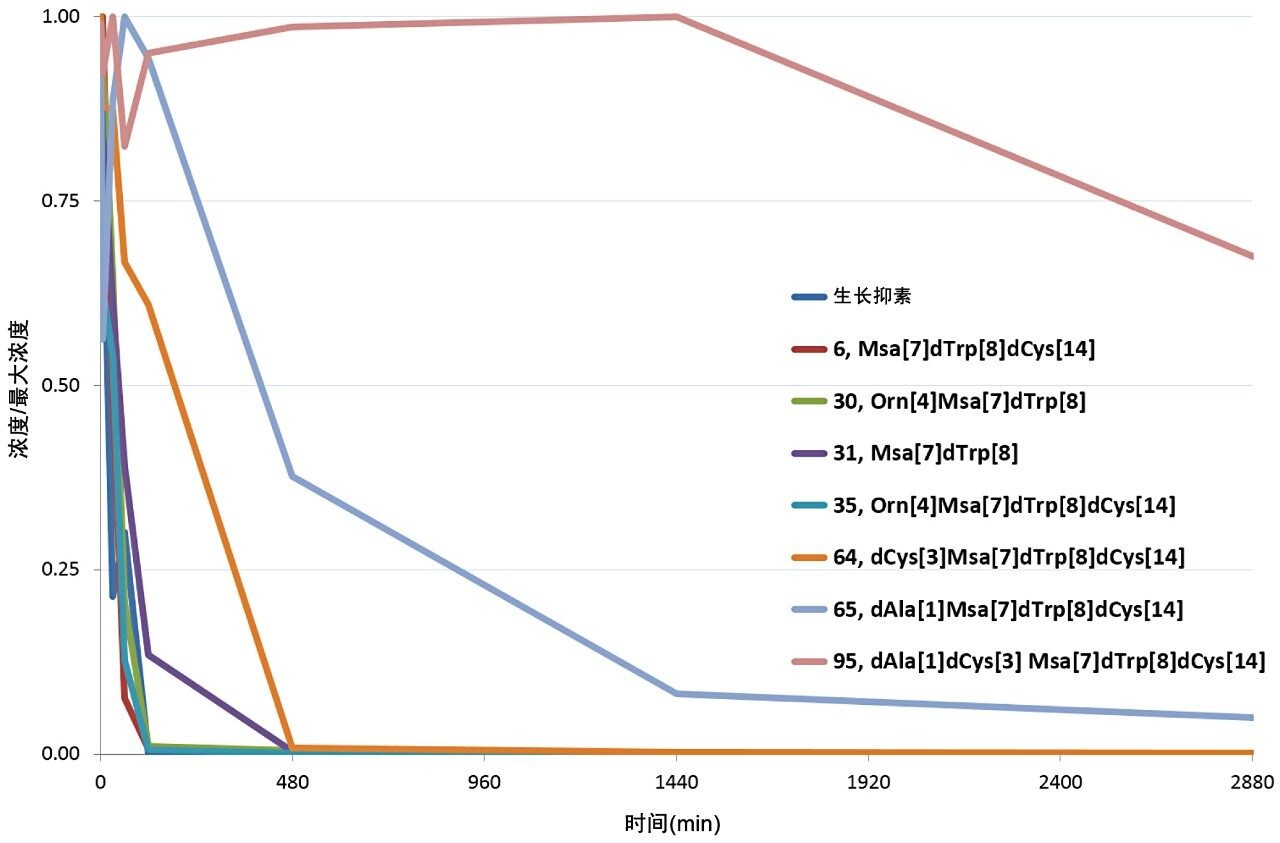
使用WebMetabase完成所有肽类似物的评估后,绘制稳定性曲线(使用Excel),展示两日内的相对比较结果(图5)。经WebMetabase处理的分解代谢物信息表明,D-氨基酸取代Ala[1]、Cys[3]和Cys[14]后,不仅能够增强各自的稳定性,而且具有协同作用。某些修饰(尤其是将Cys[14]修饰为D-Cys[14])本身无显著影响,如同将肽31修饰为肽6一样,但与D-Cys[3]结合后,有效性出现极大提升,如同将肽6修饰为肽64,或者将肽65修饰为肽95一样。用Orn[4]取代Lys[4],以及用Msa[7]D-Trp[8]取代Phe[7]Trp[8]后,其总体稳定性相对于天然生长抑素的差异较小(详细数据未显示)。总体而言,肽95看起来非常稳定,与其他类似物相比,其所有代谢均有延迟和减少,进一步证明了这一结论。
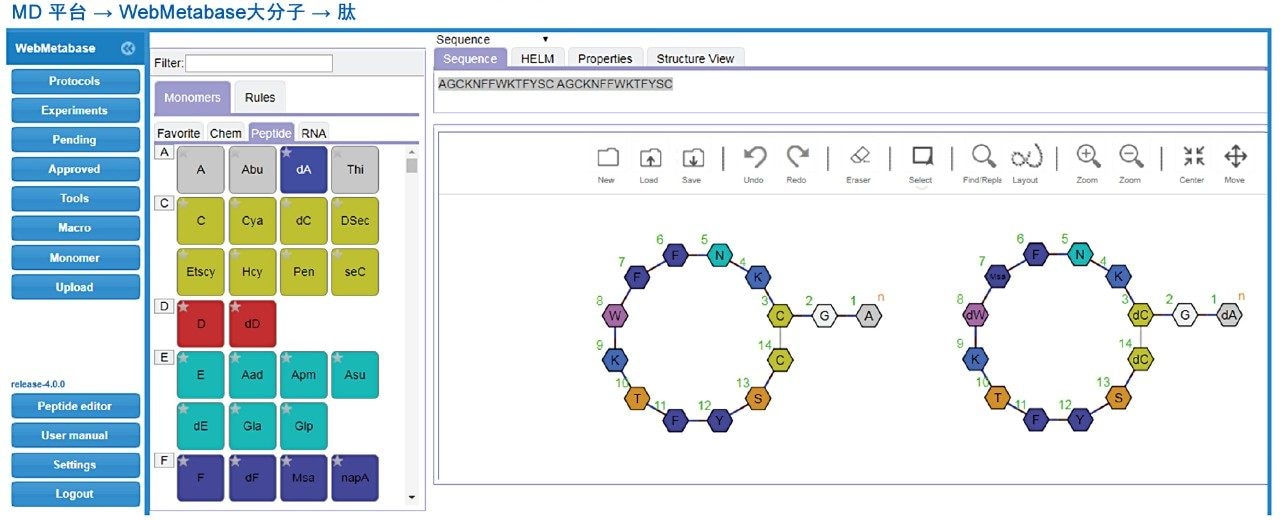
HELM notation使分析人员能够以单体形式查看复杂大分子结构(Pistoia Alliance, https://www.pistoiaalliance.org/projects/current-projects/helm/)。此功能保留了单体的化学结构信息,并将序列结构标注与大分子结构的复杂分子图(例如,.mol或.SDF格式)之间联系起来。图6展示了一个相关示例,其中使用HELM notation展示了生长抑素和肽95的分子结构。
利用WebMetabase处理和显示所有8种生长抑素类似物的数据。分解代谢主要发生在化合物结构中二硫键环外部的末端氨基酸上。主要代谢途径及其发生率通过观察母体稳定性和监测关键裂解的出现来监测。L-半胱氨酸和末端L-丙氨酸残基的取代最大程度地提高了稳定性。观察到某些变化具有协同效应,例如D-Cys[3]与D-Cys[14]偶联可显著提高观察到的稳定性。经过WebMetabase处理的Vion数据能够高效鉴定这些修饰及其对减少生长抑素降解的影响,从而揭示何时通过取代来增强或阻断特定的裂解。
应用离子淌度和生成的CCS值,能够为各种肽提供额外的追踪特性、改善经过淌度筛选的XIC以及进一步挖掘特定取代或裂解中CCS对分子大小的影响,从而提高数据质量。使用Mass-MetaSite和WebMetabase处理离子淌度数据,提供了一种高效、简单直接的方法来查看复杂的肽分解代谢数据。
整合Molecular Discovery等第三方供应商提供的功能后,能够快速获取IMS增强型工作流程、HELM integration和云服务可视化软件包等功能。有效利用HELM integration功能,可为生物学家提供一种更熟悉的视角(而不是.mol或.SDF格式),随着后续开发出结构更复杂的分子和杂化物,这一功能将非常有用。利用云服务存储、处理和共享数据,可使全球多地的多名项目用户都能够使用集中式服务器(位于英国)。各站点之间定期共享数据有助于讨论观察结果并分享某些研究人员的知识。通过浏览器访问WebMetabase可支持从多台PC和多个地点轻松查看,减少了客户端PC所需的昂贵硬件需求以及安装本地软件副本的要求。
720006586ZH,2019年8月