Peptid- und Oligonukleotid-Mapping sind unerlässliche Prozesse zur Bestätigung der Identität, Reinheit und Sequenz komplexer Biomoleküle wie Proteine und RNA-Therapeutika. RapiZyme Enzyme wurden für die LC-MS-Analyse moderner Biotherapeutika entwickelt und liefern schnelle, reproduzierbare Verdauungen für:
Diese Tools ermöglichen LC-MS-Anwendern robuste, hochauflösende Charakterisierungsworkflows für moderne Biotherapeutika. Verbessern Sie die Durchlaufzeit Ihrer Proben, verbessern Sie den Laborbetrieb und treffen Sie wichtige Entscheidungen sicher und schnell mit den Lösungen von Waters.

Vergleich von 1:5-Verdauungen zwischen einem branchenführenden Mitbewerber (oben) und RapiZyme Trypsin (unten), mit vergrößertem Ausschnitt des Retentionszeitfensters von 14 bis 40 Min. Rote Pfeile kennzeichnen Trypsin-Autolyse und unbekannte Peaks.
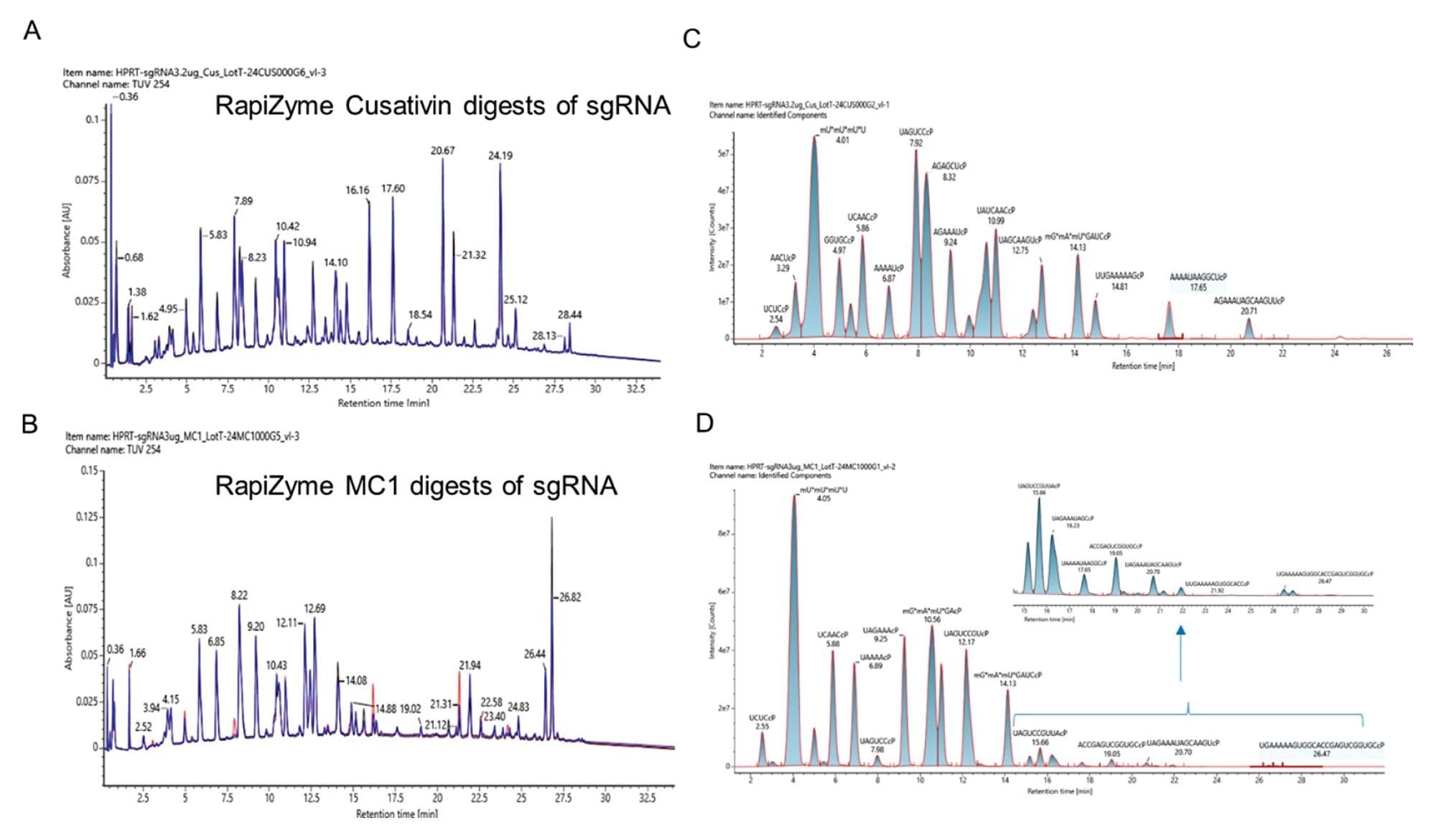
LC-UV-MS-Analyse der HPRT-sgRNA, die mit rekombinantem RapiZyme Cusativin und MC1 verdaut wurde. Drei unabhängige rekombinante Enzymchargen von RapiZyme Cusativin (A) und RapiZyme MC1 (B) wurden zum Verdau von HPRT sgRNA verwendet. Die drei UV-Chromatogramme (schwarze, blaue und rote Kurven) sind übereinandergelegt, um das reproduzierbare Verdauverhalten der drei Präparationen aufzuzeigen. (C) Extrahierte Ionenchromatogramm-Überlagerung der identifizierten RapiZyme Cusativin Verdauprodukte, die ihre Häufigkeit in der repräsentativen TUV-Spur zeigt. (D) Extrahierte Ionenchromatogramm-Überlagerung der identifizierten RapiZyme MC1 Verdauprodukte, die ihre Häufigkeit in der repräsentativen TUV-Spur darstellt. Das Profil der RapiZyme MC1-Verdauprodukte mit geringer Häufigkeit ist in der nebenstehenden Abbildung dargestellt.
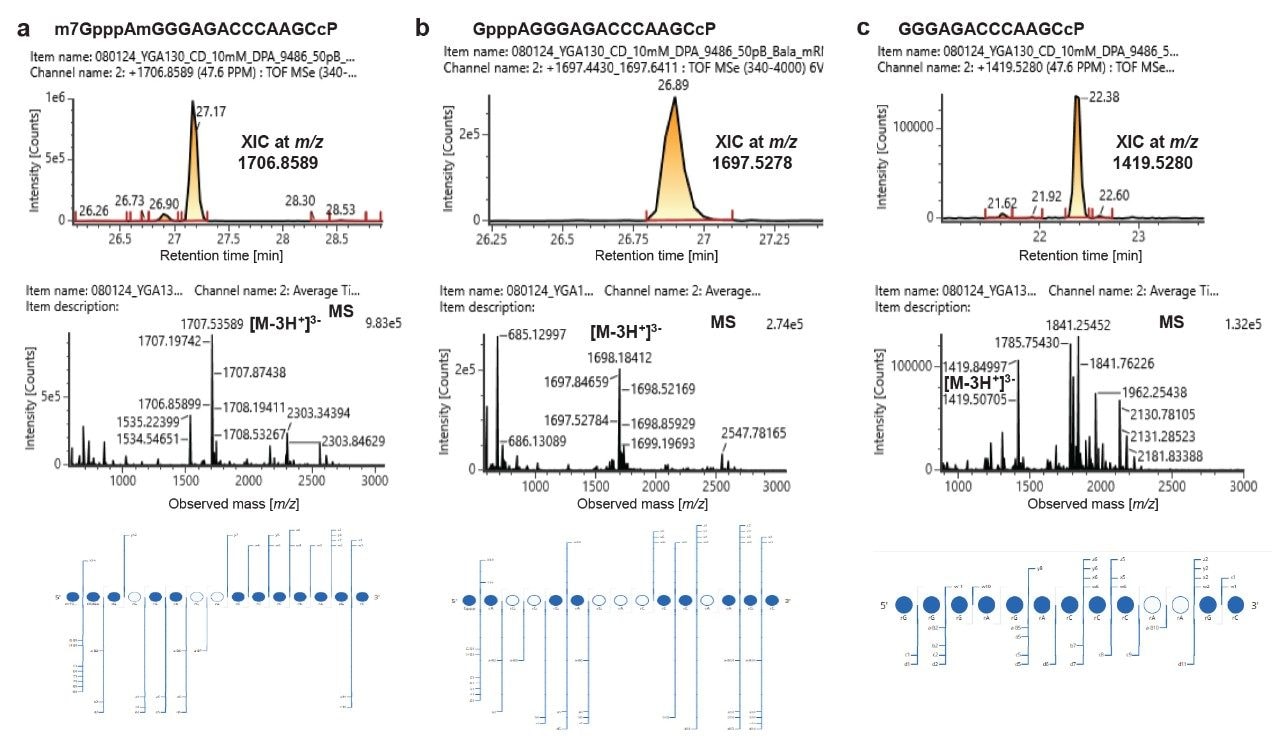
Charakterisierung verschiedener Formen von 5'-gecappten Oligonukleotiden im RNase-MC1-Verdau von mRNA 1 (XIC ist der obere Bereich, das Massenspektrum der mittlere Bereich, das Dotmap der MSE-Fragmentionen der untere Bereich). Detektion eines mit einer Cap versehenen Oligonukleotids (a), der unmethylierten Form der Cap-Version (b) und einer Version ohne Cap (c).
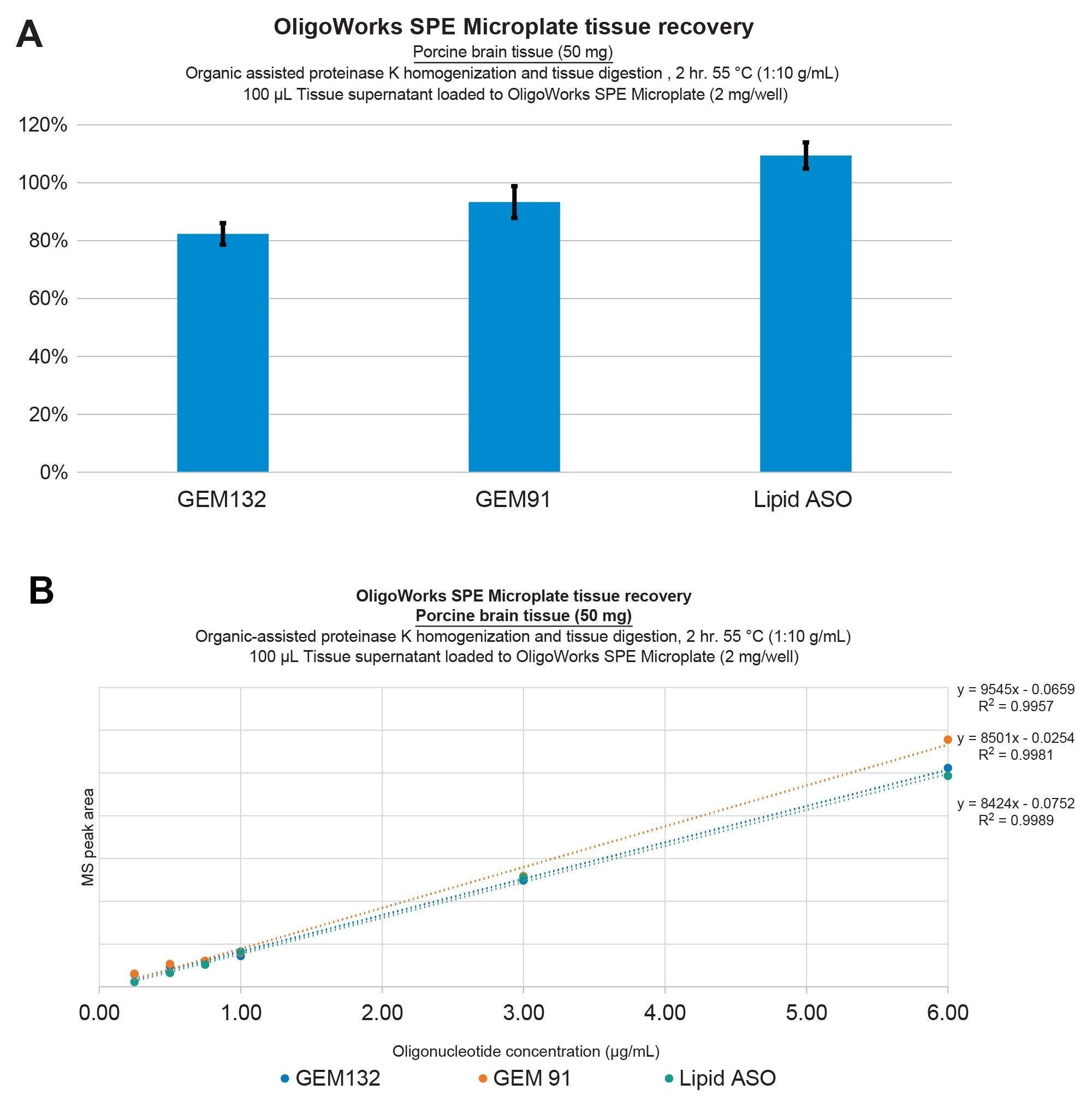
Demonstration der Oligonukleotid-Extraktionsleistung für GEM91, GEM132 und das lipidkonjugierte ASO aus Schweinehirngewebe. Die Wiederfindungsraten wurden zu ≥80 % bei hervorragender Wiederholbarkeit (SDs ≤10 %) bestimmt (A). Die Extraktionslinearität wurde mittels lösungsmittelunterstützter Proteinase-K-Gewebehomogenisierung und Verdau, gefolgt von einer Mixed-Mode-SPE-Reinigung mit der OligoWorks SPE Mikrotiterplatte und Reagenzien, auf ≥0,99 ohne interne Standardkorrektur bestimmt (B).
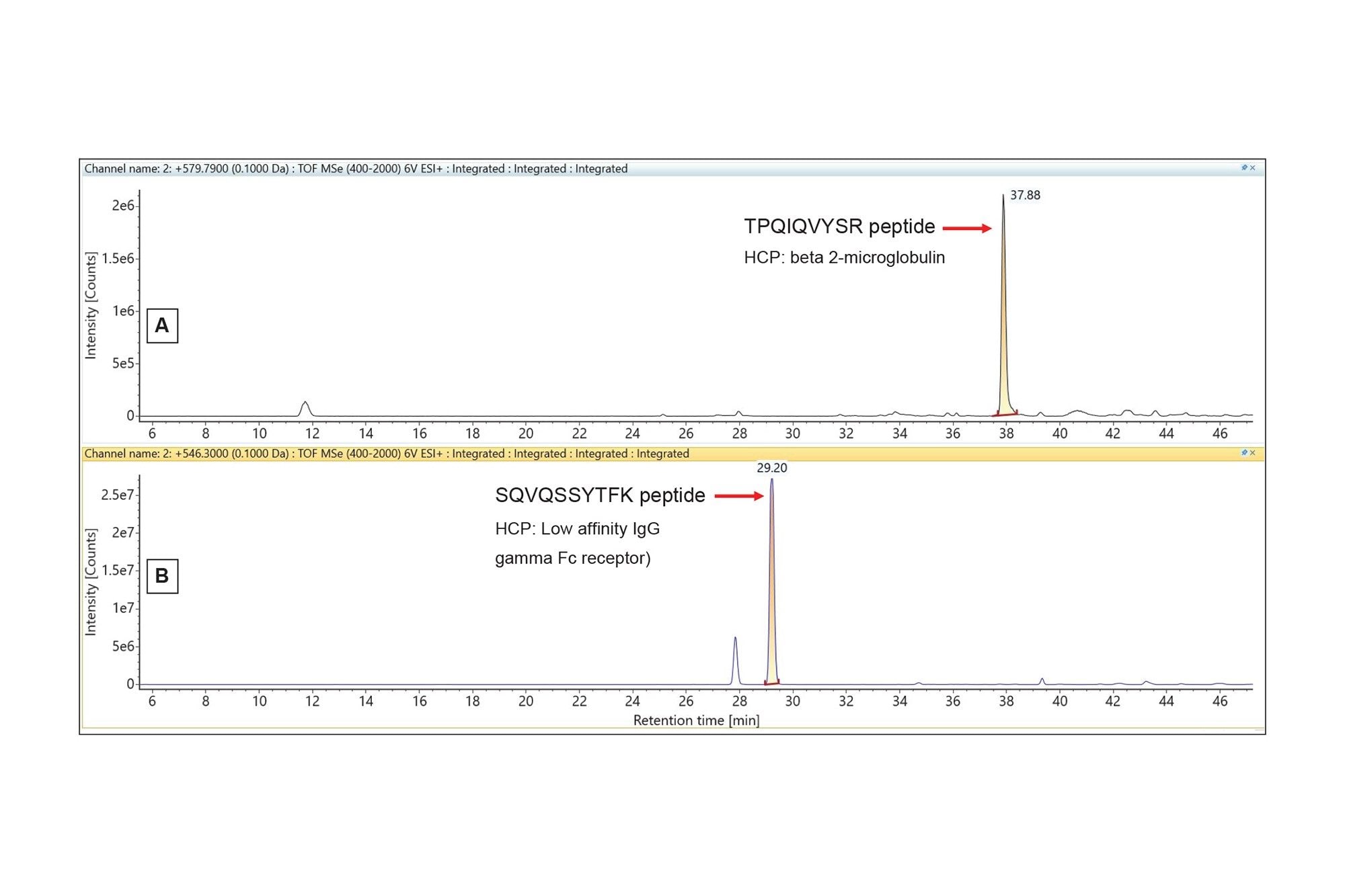
Extrahierte Massenchromatogramme für zwei HCP-Peptide mit geringer Häufigkeit, die im NIST mAb-Verdau mit dem Discovery HCP Assay identifiziert wurden.