El mapeo de péptidos y oligonucleótidos es un proceso esencial para confirmar la identidad, la pureza y la secuencia de biomoléculas complejas, como proteínas y ARN terapéuticos. Diseñadas para el análisis LC-MS de productos bioterapéuticos modernos, las enzimas RapiZyme ofrecen digestiones rápidas y reproducibles para:
Estas herramientas permiten flujos de trabajo de caracterización robustos y de alta resolución para bioterapéuticos modernos destinados a los usuarios de LC-MS. Mejore el tiempo de respuesta de las muestras y las operaciones de laboratorio, y tome decisiones críticas con confianza y rapidez con las soluciones de Waters.

Comparación de digestiones 1:5 entre otro competidor líder en la industria (panel superior) y tripsina RapiZyme (panel inferior), con la sección ampliada de la ventana de tiempo de retención de 14 a 40 min. Las flechas rojas resaltan la autólisis de tripsina y los picos desconocidos.
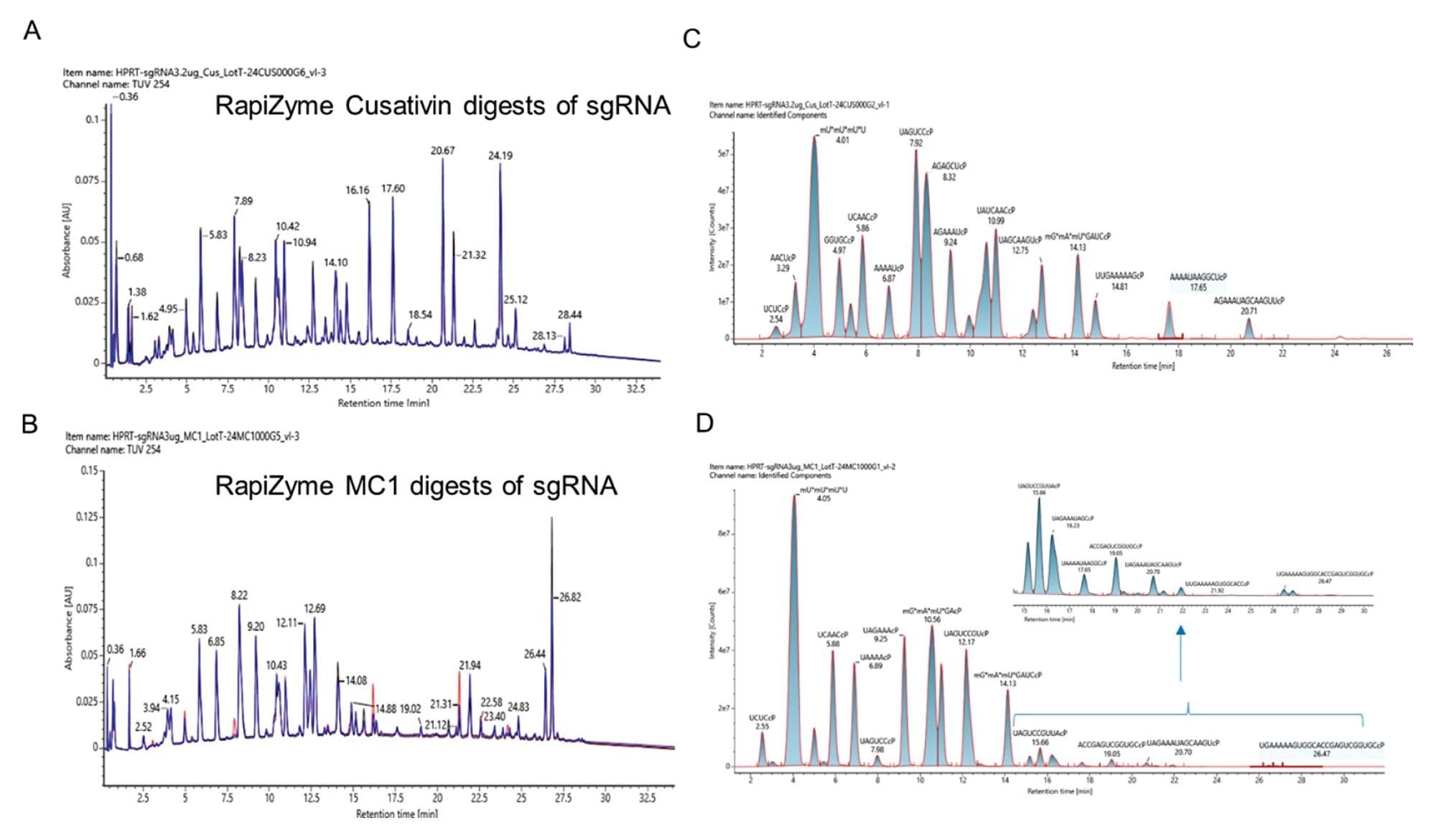
Análisis LC-UV-MS del sgRNA de HPRT digerido con RapiZyme Cusativin recombinante y MC1. Se utilizaron tres lotes de enzimas recombinantes independientes de RapiZyme Cusativin (A) y RapiZyme MC1 (B) para digerir el sgRNA de HPRT. Los tres cromatogramas UV (trazos negros, azules y rojos) se superponen para indicar el comportamiento de digestión reproducible de tres preparaciones. (C) Superposición del cromatograma de iones extraídos de los productos de digestión de RapiZyme Cusativin identificados, que indica su abundancia en el trazado de TUV representativo. (D) Superposición del cromatograma de iones extraídos de los productos de digestión RapiZyme MC1 identificados, que indica su abundancia en el trazado TUV representativo. En la figura del recuadro se muestra el perfil de los productos de digestión de baja abundancia de RapiZyme MC1.
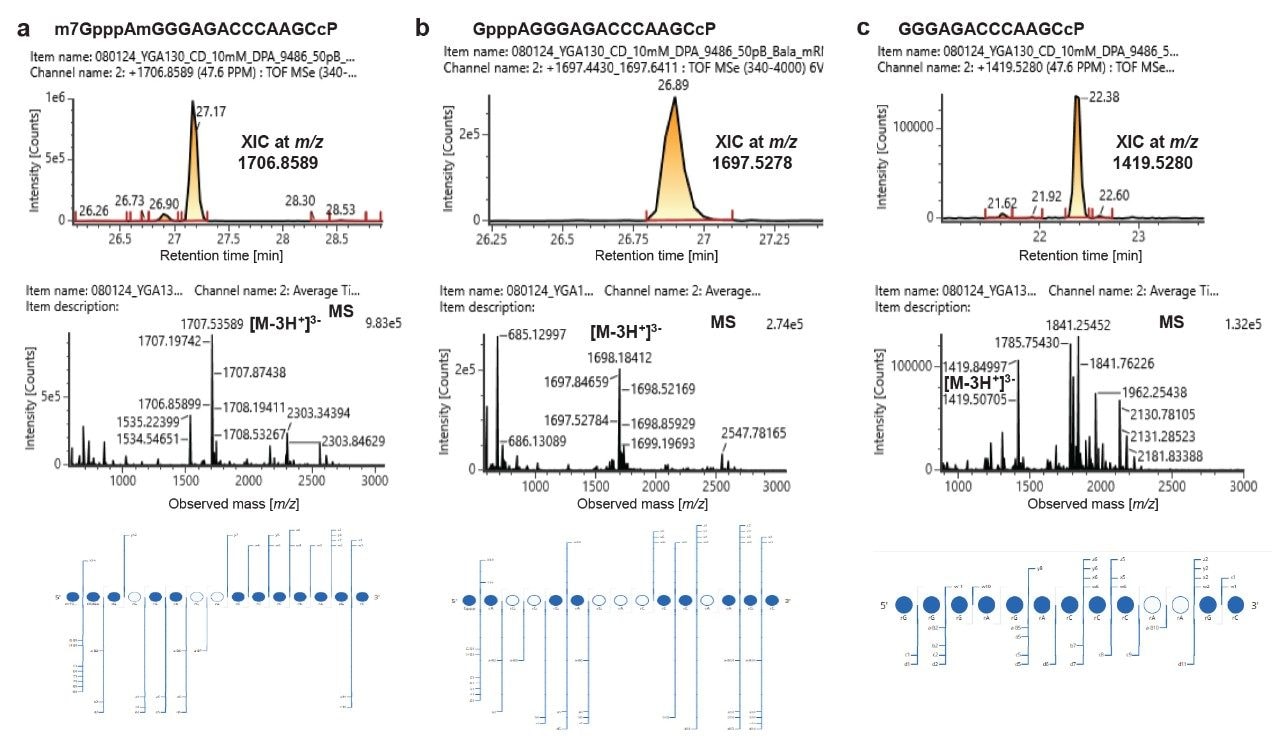
Caracterización de diferentes formas de oligonucleótidos con caperuza 5' en la digestión con ARNm 1 con ribonucleasa MC1 (XIC es el panel superior, el espectro de masas es el panel central, el mapa de puntos de los iones del fragmento MSE es el panel inferior). Detección del oligonucleótido con caperuza (a), la forma no metilada de la versión con caperuza (b) y la versión sin caperuza (c).
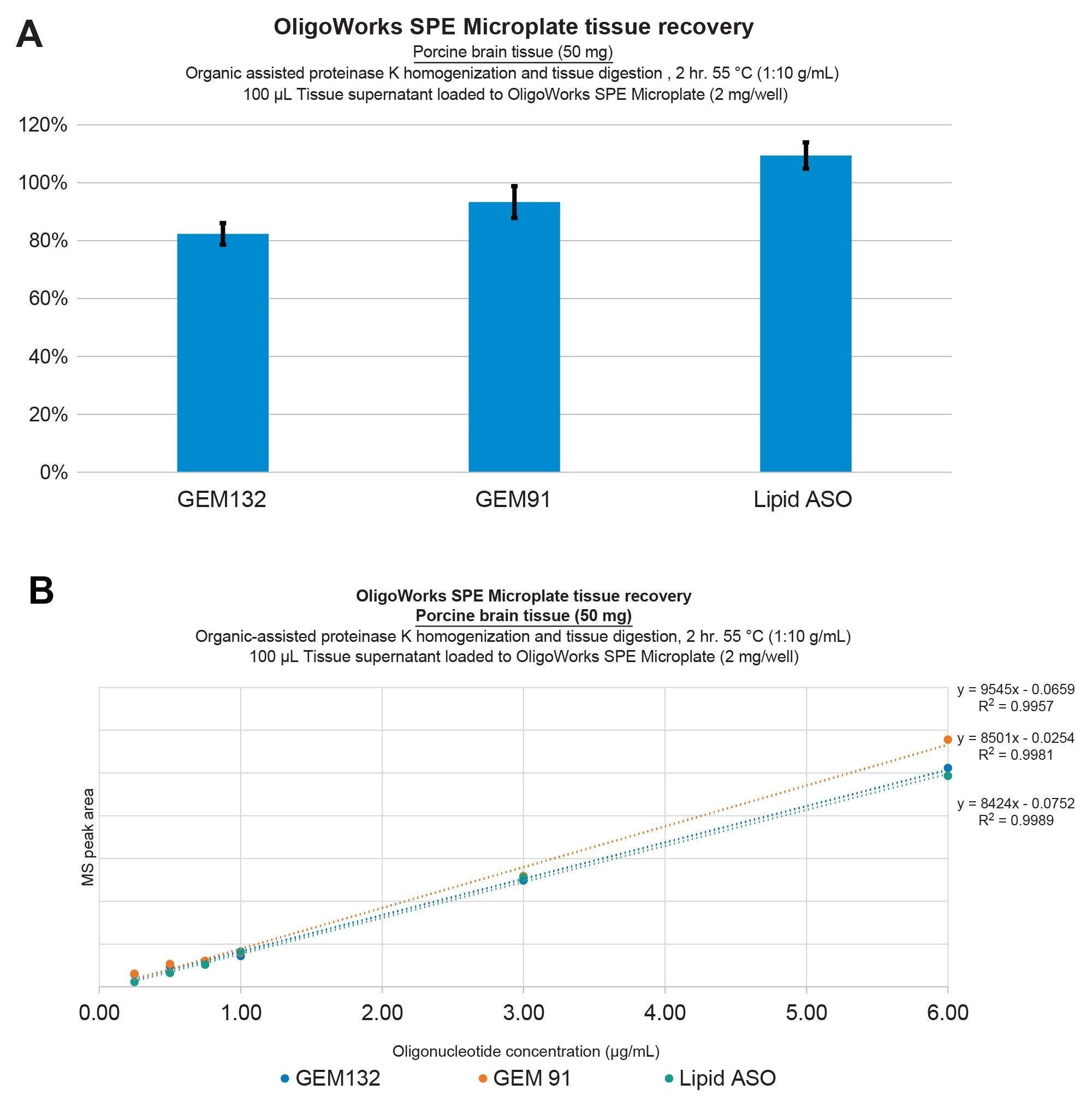
Demostración del rendimiento de la extracción de oligonucleótidos para GEM91, GEM132 y el ASO conjugado con lípidos de tejido cerebral porcino. Se determinó que las recuperaciones eran ≥80 % con una repetibilidad excelente (SD ≤10 %). (A) Se determinó que la linealidad de la extracción era ≥0,99 sin corrección de patrón interno (B) utilizando homogeneización de tejidos con proteinasa K asistida por eluyente y digestión, seguida de purificación de SPE en modo mixto con la microplaca y los reactivos de SPE OligoWorks.
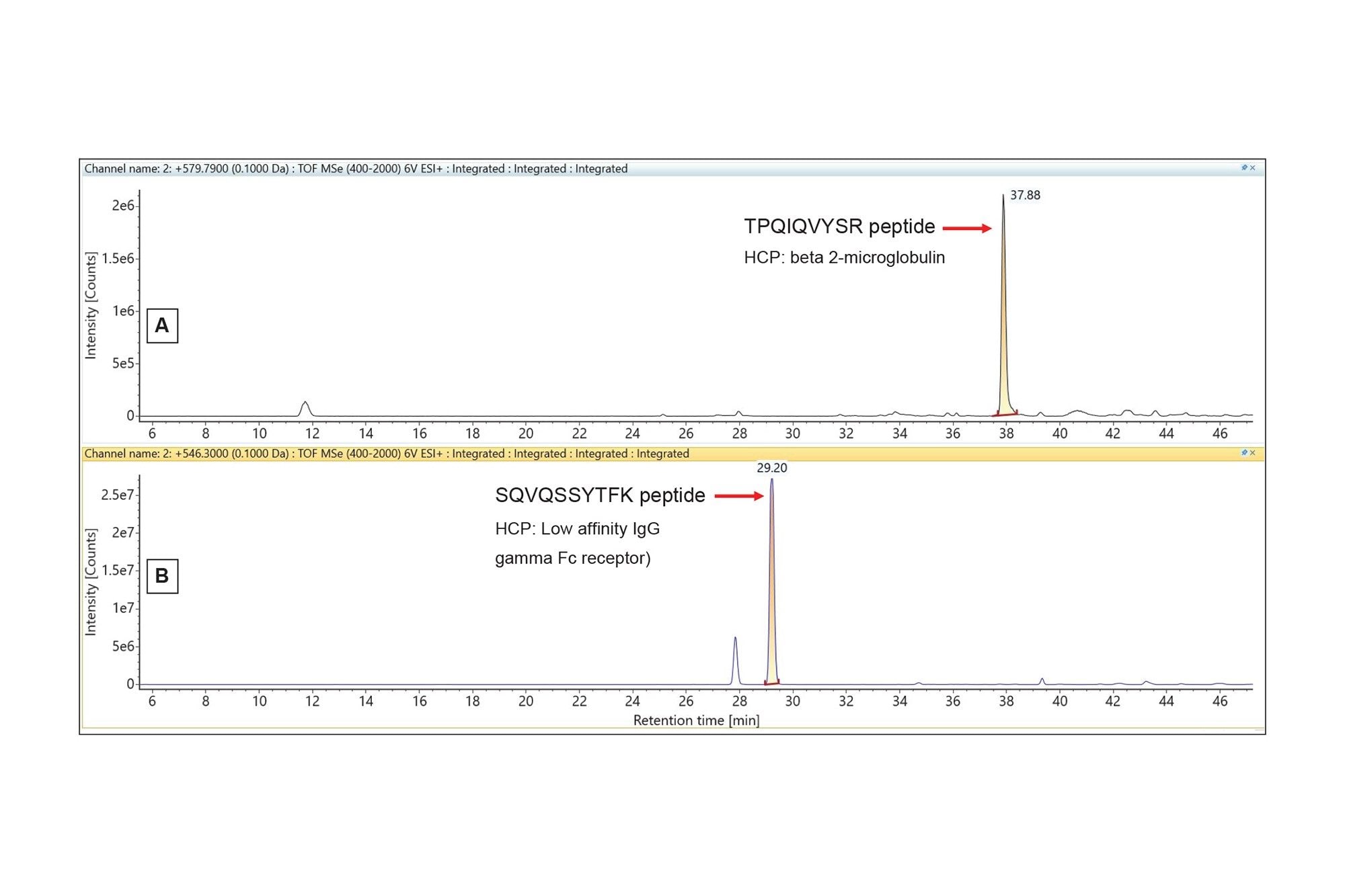
Cromatogramas de masas extraídos para dos péptidos de HCP de baja abundancia identificados en la digestión con mAb NIST utilizando el ensayo de HCP de descubrimiento.