二本鎖 siRNA、脂質結合 ASO、CRISPR sgRNA などのオリゴヌクレオチド医薬品の HILIC UV/MS 分析に用いる移動相の検討
要約
オリゴヌクレオチド(ON)分離における従来のイオン対逆相(IP-RP)メソッドに代わる可能性のある有望な代替手段として、親水性相互作用液体クロマトグラフィー(HILIC)が登場しました。この試験では、ON のジアステレオマー成分、二本鎖成分、一本鎖成分の分離への HILIC の応用について検討し、質量分析(MS)との適合性によってイオン対試薬が不要になることに焦点を当てています。塩濃度、温度、移動相組成などの主なパラメーターを実験計画法(DoE)を用いて系統的に最適化し、感度を高めました。ブレークスルー効果やピーク割れを解消するための、シーケンス注入手順も提案しています。
アプリケーションのメリット
- HILIC により、さまざまな濃度のアンモニウムベースの移動相に簡単に適用できる、イオン対試薬が不要なクロマトグラフィーメソッドを提供
- イオン化効率は HFIP ベースの IP-RP LC-MS より低いが、25 mM という低塩濃度の移動相を使用して信頼性の高い LC-MS メソッドを適用可能
- GTxResolve™ Premier BEH™ Amide 300 Å 1.7 µm カラムは、核酸とタンパク質の両方を分離する性能についてバッチテストおよび選定済み
- MaxPeak™ Premier カラムハードウェアにより回収率が向上し、使用前にカラムを不動態化する必要性が最小限に
はじめに
オリゴヌクレオチド分離用 HILIC
最近、オリゴヌクレオチド(ON)分離において、親水性相互作用液体クロマトグラフィー(HILIC)に対する関心が高まっています1,2,3。 HILIC モードは、最も一般的に使用されているイオン対逆相(IP-RP)メソッドの代替になる、感度が高く頑健で魅力的な手段を提供します。HILIC は、ON のジアステレオマーの分離において特に有望であることがわかりました。高塩濃度と低移動相温度が、ジアステレオマーの選択性に有利に働くことが示されています3。 HILIC は、移動相温度に応じて変性条件でも非変性(ネイティブ)条件でも使用できるため、二本鎖型をその一本鎖成分から分離することも可能です4。 HILIC のもう 1 つの重要な利点として、イオン対を含まない移動相を使用できるため、MS アプリケーションに適していることが挙げられます5,6。 HILIC は、LC-MS セットアップおよび LC-MS/MS セットアップでの sgRNA のボトムアップ配列決定にも適用されています7。
HILIC で ON 用の頑健なメソッドを開発することができますが、その保持メカニズムは完全には理解されていません。一部の論文では、イオン性相互作用が保持メカニズムに関与している可能性があるとされていますが(この場合、反発効果のはずであることに注意が必要)、ON の HILIC は主に水素結合相互作用(吸着または分配)によってもたらされると結論付けている論文もあります1,3。 高塩濃度の移動相が、極性(アミド)固定相表面に固定化された水層を安定化して、イオン性相互作用を最小限に抑えるのであろうと考えられています3。 一方、一部のクロマトグラフィーモデルではその逆が提唱されています。つまり、水系移動相に塩が添加されると、水分子が主に塩イオンを溶媒和し(水の高次構造が失われ)、その結果、親水性表面と相互作用できる水分子の数が減少する(固定化された水層の厚みが減少することが予想される)という考えです。アセトニトリルの代わりにプロトン性共溶媒(つまりメタノール)を使用すると、HILIC カラムで良好なピーク形状と同様の選択性が得られるという結果も重要な観察結果です。この事実により、保持メカニズムの理解がさらに複雑になります。最後に、HILIC カラムから ON を溶出させるために必要な水の量はかなり多く(20 ~ 60% の水)、糖鎖や糖タンパク質の溶出に必要な量(15 ~ 35% の水)よりもはるかに多いことが特筆されます8。
実験方法
LC メソッドに関する検討事項
HILIC での ON 分離に関する体系的なメソッド開発についての言及はほとんどありません。ほとんどの場合、同じまたは類似の条件が使用されています(10 ~ 50 mM の酢酸アンモニウム、ギ酸、または重炭酸、および共溶媒としてのアセトニトリル)。移動相添加剤の塩濃度、温度効果および pH の試験が実施されていますが、ほとんどの場合、実験計画法(DoE)の組み合わせや多次元保持モデルではなく、個別の試験(一度に 1 つの要因、OFAT)が実施されています1,3,9。 今回、体系的アプローチを使用して、3 種類のアンモニウム塩を比較しました。まず、スクリーニングの目的で 3 要因 DoE を適用して、グラジエントの勾配(グラジエント時間、tG)、温度(T)、塩濃度(C)の影響を試験しました。tG と T の影響は 2 つのレベルで試験し、C の影響は 3 つのレベルで試験しました。次に、グラジエントのプログラムと温度を同時にさらに最適化して(DryLab™ 4.4 ソフトウェア(Molnar Institute、ドイツ、ベルリン)を使用)、さまざまなオリゴヌクレオチドラダーサンプルに全般的に適用できる 10 分間分離法を得ました。
HILIC ではブレークスルー効果(または部分的なピーク割れ)がしばしば発生します。これは、サンプル希釈液(タンパク質やオリゴヌクレオチドなどの生体分子は水系媒体中に溶解する必要がある)とほとんどの場合非プロトン性溶媒(アセトニトリル)からなる移動相の間の本質的な不適合によるものです10。 実践的なクロマトグラファーは、ブレークスルー効果を避けるために、アセトニトリル-水混合液でのサンプル希釈という妥協策を取ることが多くなります。これにより、サンプルの沈殿および/または変性を引き起こす可能性があります。今回、より洗練された解決策を提案します。つまり、弱溶媒(アセトニトリル)の注入前プラグおよび注入後プラグを追加すると同時に、サンプルを純粋な水系溶媒プラグ(シーケンス注入または「ブラケット」注入)の形で注入します。
オリゴヌクレオチド標準試料/ラダーの LC 分析
サンプル前処理:
オリゴ dT ラダーは、MassPREP™ オリゴヌクレオチド標準試料(製品番号:186004135)のバイアルの内容物を 100 µL の水に再溶解して調製しました。ssDNA ラダーは、ssDNA ラダー 20 ~ 100(製品番号:186009448)のバイアルの内容物を 100 µL の水に再溶解して調製しました。脂質結合 ASO オリゴヌクレオチドサンプルは、脂質結合 ASO LC-MS 標準試料(製品番号:186010747)の内容物を 20 µL のアセトンに再溶解し、80 µL の水で希釈することによって調製しました。siRNA LC-MS 標準試料(製品番号:186010598)は、100 µL 水に再溶解し、HILIC での融解温度の検討に使用しました。
移動相の調製:
HPLC グレードの水およびアセトニトリル(MeCN)は、Fisher Scientific(アイルランド、ダブリン)から購入しました。酢酸アンモニウム、ギ酸アンモニウム、重炭酸アンモニウムは、Sigma-Aldrich(スイス、ブフス)から購入しました。LC 試験では、沈殿および溶解度の問題を回避するために、事前混合した移動相を使用しました。移動相 A は水/MeCN 30/70、移動相 B は水/MeCN 90/10 を用いました。このような移動相組成により、0 ~ 100%B の直線的グラジエントを実行すると、すべてのオリゴヌクレオチド標準試料が適切に保持されて溶出できることがわかりました。移動相の添加剤の影響を検討するために、A および B の両方の 3 種類のアンモニウム塩すべてについて、3 種類の塩濃度(C1= 10 mM、C2 = 25 mM、C3 = 50 mM)を検討しました。
最終的に最適化された LC-UV メソッドでは、50 mM の塩を移動相 A と B の両方に添加しました。
その他の実験条件(LC-UV)はすべて、以下の表に記載しています。
|
LC システム: |
バイナリーソルベントマネージャー搭載 ACQUITY™ UPLC™ H-Class Bio システム(高 pH キットで構成した BSM FTN 装置搭載 ACQUITY Premier システムと同等) |
|
検出: |
ACQUITY TUV 検出器(チタン製フローセル、5 mm、1,500 nL) |
|
波長: |
260 nm |
|
データ取り込み: |
Empower™ Pro 3 ソフトウェア Feature Release 3 |
|
カラム: |
GTxResolve Premier BEH Amide カラム、300 Å、1.7 µm、2.1 × 50 mm カラム(製品番号:186011249) |
|
カラム温度: |
最適化したメソッドでは 50 ℃(温度効果をモデル化するために 30 ℃ および 70 ℃ に設定) |
|
サンプル温度: |
6 ℃ |
|
バイアル: |
MaxPeak™ HPS を採用した QuanRecovery™ 12 × 32 mm スクリューネックバイアル、300 µL(製品番号:186009186) |
|
注入量: |
0.5 ~ 1 µL |
|
流量: |
0.4 mL/分 |
|
移動相: |
A:50 mM 酢酸アンモニウム/ギ酸/重炭酸含有水/MeCN 30/70 B:50 mM 酢酸アンモニウム/ギ酸/重炭酸含有水/MeCN 90/10 |
|
シリンジ吸引速度: |
30 µL/分 |
|
ニードル配置: |
1.0 mm |
|
TUV サンプリングレート: |
20 Hz |
|
フィルタータイムコンスタント: |
なし |
脂質結合 ASO および CRISPR sgRNA の LC-MS 分析
Waters™ 脂質結合 ASO LC-MS 標準試料(製品番号:186010747)は、まず 20 µL のアセトンに再溶解し、次にヌクレアーゼフリーの水 80 µL で希釈しました。HPRT sgRNA を IDT から取得し、ヌクレアーゼフリー水に濃度 50 pmol/ µL になるように溶解しました。
|
LC システム: |
BioAccord™ LC-MS システム |
|
検出: |
ACQUITY TUV 検出器(分析用フローセル) |
|
波長: |
260 nm |
|
データ取り込み: |
Empower Pro 3 ソフトウェア Feature Release 3 |
|
カラム: |
GTxResolve Premier BEH Amide カラム、300 Å、1.7 µm、2.1 × 50 mm カラム(製品番号:186011249) |
|
カラム温度: |
75 ℃ |
|
サンプル温度: |
6 ℃ |
|
バイアル: |
MaxPeak HPS を採用した QuanRecovery 12 × 32 mm スクリューネックバイアル、300 μL(製品番号:186009186) |
|
注入量: |
1 µL |
|
流量: |
0.4 mL/分 |
|
移動相: |
A:100 mM 酢酸アンモニウム含有 18.2 MΩ 水/1% アセトニトリル(1 L 品質保証 LDPE リザーバー(製品番号:186009110)中の IonHance 酢酸アンモニウム濃縮液(製品番号:186009705)から調製) |
|
(sgRNA): |
B:1 L 品質保証ガラスリザーバー中の LC-MS グレードのアセトニトリル(製品番号:186007089) |
|
移動相: |
A:25 mM 酢酸アンモニウム含有 18.2 MΩ 水/0.25% アセトニトリル(1 L 品質保証 LDPE リザーバー(製品番号:186009110)中の IonHance™ 酢酸アンモニウム濃縮液(製品番号:186009705)から調製) |
|
(脂質 ASO): |
B:1 L 品質保証ガラスリザーバー中の LC-MS グレードのアセトニトリル(製品番号:186007089) |
|
シリンジ吸引速度: |
30 µL/分 |
|
ニードル配置: |
2.0 mm |
|
TUV サンプリングレート: |
20 Hz |
|
フィルタータイムコンスタント: |
なし |
|
MS モード: |
フルスキャン |
|
質量範囲: |
高(m/z 400 ~ 5000) |
|
スキャンレート: |
5 Hz |
|
コーン電圧: |
40V |
|
脱溶媒温度: |
550 ℃ |
LC-MS 実験のグラジエントプログラム
結果および考察
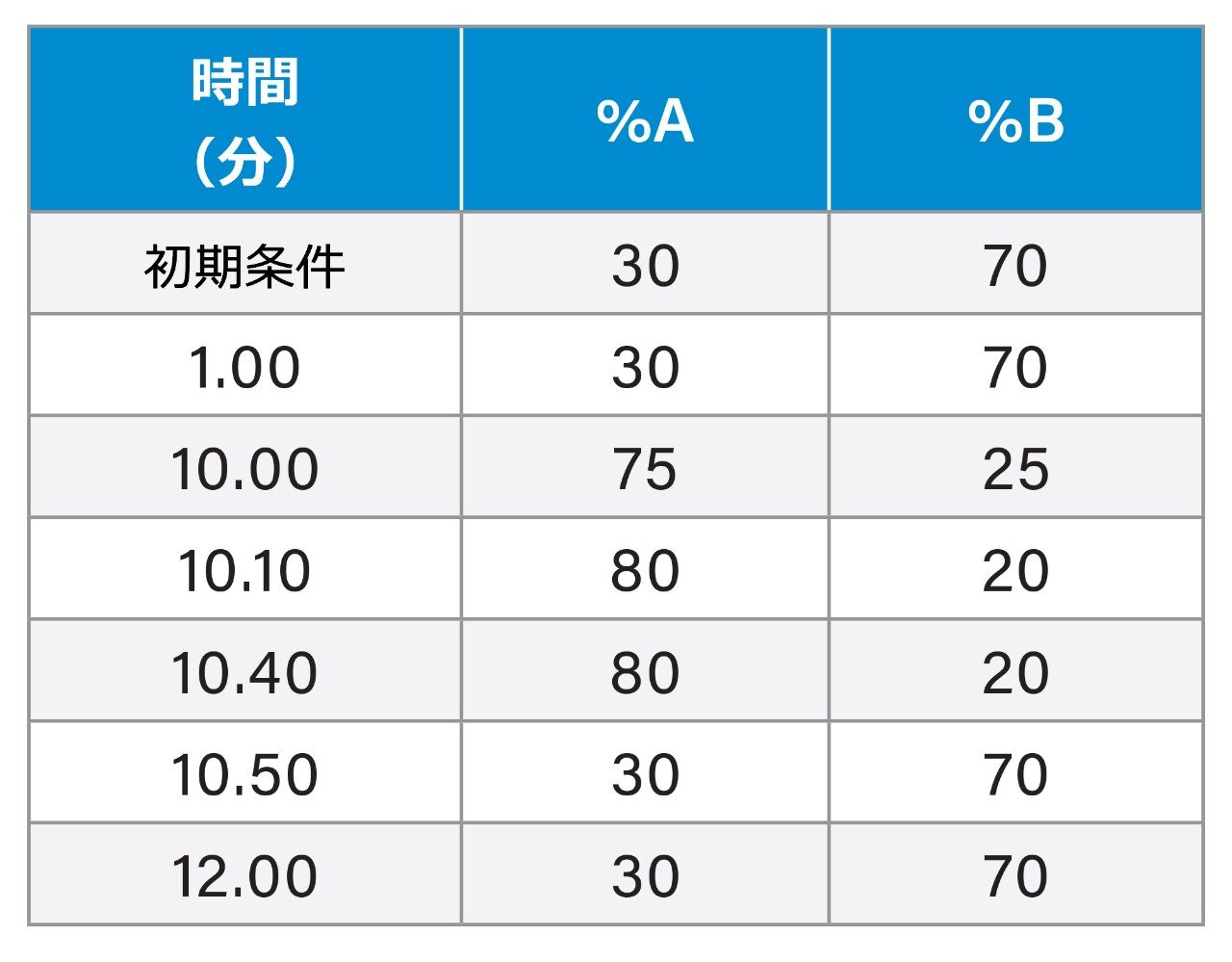
移動相添加剤としての 3 種類のアンモニウム塩の比較
C ≤ 40 mM の場合、塩濃度の影響が重要であることがわかりました。C ≤ 40 mM の場合、塩濃度が下がると保持が低下します。添加剤の濃度が低すぎる場合(C = 10 ~ 20 mM)、最初に溶出したピークの保持が見かけ上高く設定されていても(見かけ上の保持係数 kapp 約 5)、ブレークスルーおよび/またはピーク割れが大きくなることがあります。温度が高いと、ピーク割れやブレークスルーがよりひどくなります。
一般に、さまざまな ON 分子種に対する選択性や、さらには溶出順序の調整において、温度が非常に重要な変数であることがわかったことから、サンプルごとに温度を最適化することが推奨されます。
IP-RP の場合と同じ検討事項が HILIC のグラジエントプログラムにも当てはまります11。 ON(ラダー)が均一な挙動を示すため、直線的な移動相グラジエントを使用する場合、オリゴヌクレオチドの保持時間は、直線的ではなく対数関数的に増加します。したがって必然的に、対数または凹曲線型のグラジエントによって可能な限り高い選択性が得られます。図 1 に、最適化された条件下で 3 種類の塩(添加剤)を使用して得られたクロマトグラムを示します(同じグラジエント勾配を維持)。
クロマトグラフィー分離の観点からは、酢酸アンモニウムとギ酸アンモニウムは同等です。いずれの塩にも同じグラジエントプログラムを使用でき、ほとんど同じ保持および選択性が得られます。一方、重炭酸アンモニウムは酢酸やギ酸塩よりも保持が弱いため、同様の保持を得るには、グラジエントプログラムを調整する(-5%B シフトさせる)必要があります。重炭酸では選択性がわずかに異なることに注意が必要です。重炭酸では、一部の早く溶出するピークが(特に高温で)ピーク割れやブレークスルーしやすく、これが他の 2 種類の塩より劣る点です。ただし、ブラケット注入を使用することで、この影響は解消することができます(次のセクションを参照)。
ブレークスルーを回避するためのブラケット注入
HILIC におけるサンプル注入では、注入溶媒の溶離力が移動相と比較して高いため(移動相組成と注入溶媒の間の溶媒の不適合)、サンプルがカラムインレットで十分にフォーカスしないために問題が生じることがあります10。 注入希釈液を微調整する(サンプル希釈液中のアセトニトリルの割合を増やす)ことで、この不適合を最小限に抑えられる可能性があります。ただし、サンプルの性質により、この調整が必ずしも可能とは限りません。サンプルの性質によっては変性、翻訳後修飾の誘発、不溶性(沈殿)を引き起こす場合があります。通常、この溶媒の不適合により、わずかなピークの広がりからひどいピークの変形、さらにはピーク割れや分析種のブレークスルーに至るまで、クロマトグラフィー分離に望ましくない影響が生じます。ブレークスルーとは、サンプルの一部が固定相とほとんど相互作用せずにカラムを通過して、カラムのデッドタイム近くに溶出し、ピークの他の部分は正常な保持時間に溶出するという現象です。
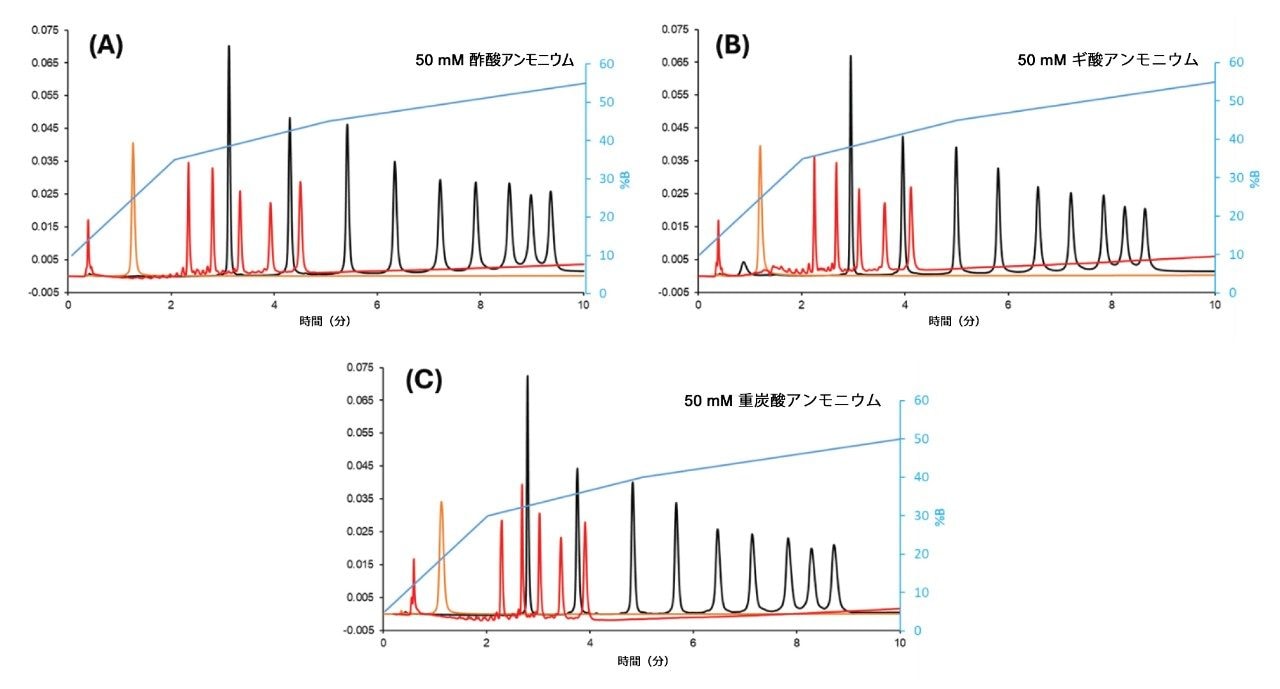
今回の例では、オリゴ dT ラダーが最もピーク割れしやすいように見えました。注入量を 0.5 µL に設定した場合、最初の化合物(15 nt)はピーク割れして溶出しました。注入量が 1.5 µL を超えると、5 種類の化合物(15、20、25、30、35 nt)すべてで大きなピーク割れが見られ、一部の溶質はカラムのボイドボリュームで溶出しました。
ブラケット注入シーケンスが、ブレークスルー効果を解消するのに役立ちます。ブラケット注入では、サンプルを、弱溶媒(この場合 MeCN)の注入前プラグと注入後プラグとともにカラムヘッドに注入します。このブラケット注入を使用すると、dT ラダーのサンプル注入量を、ピーク割れすることなく最大 2 µL まで増やすことが可能になります。図 2 に、流路に 1 µL のサンプルを導入する場合の通常の注入とブラケット注入の比較を示します。
分離を調整するための重要なパラメーターとしての温度
さまざまな分子種に対する選択性および溶出順序の調整とともに、温度も HILIC における非変性分離条件または変性分離条件を設定する際に重要です。
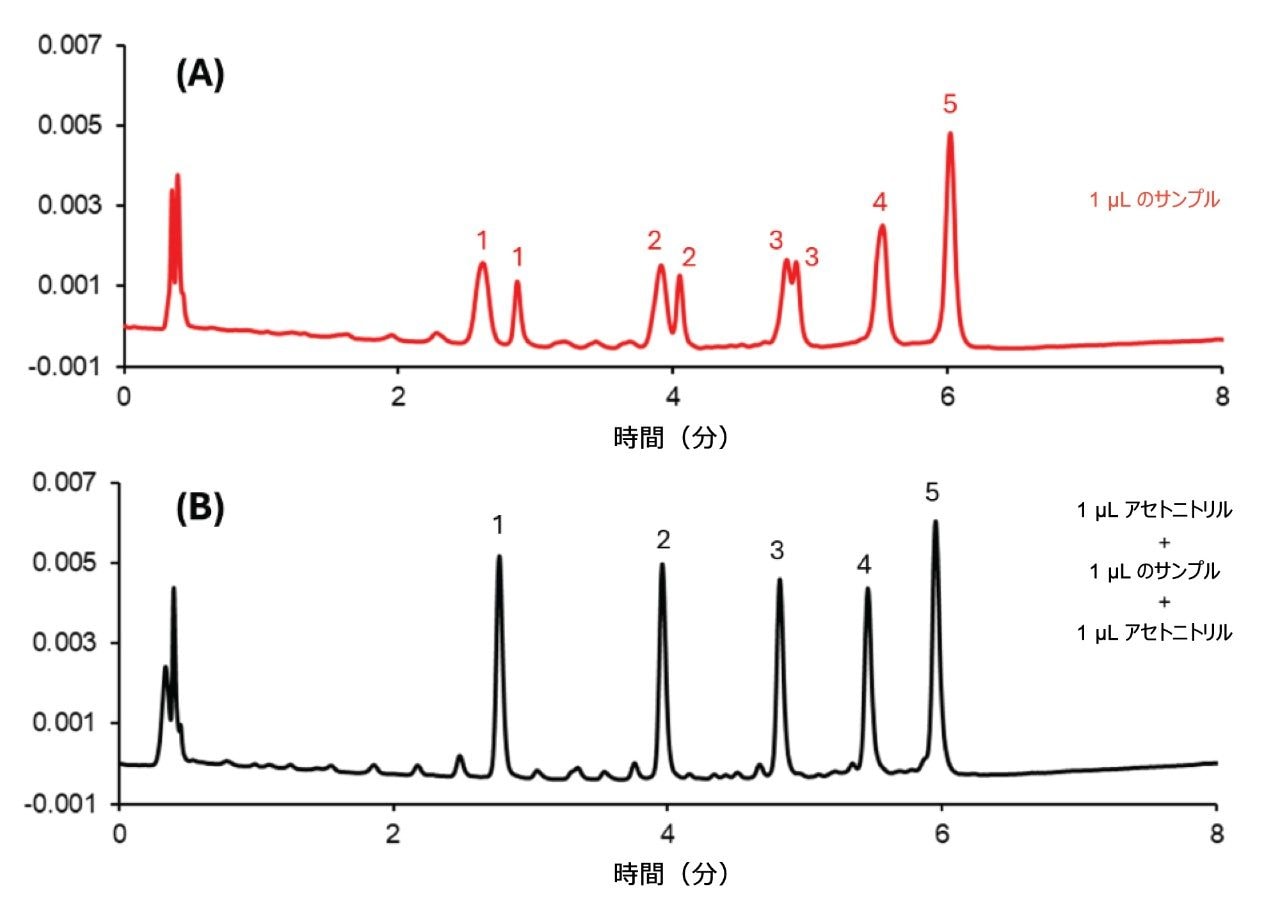
二本鎖 ON は、二本鎖融解温度(Tm)未満の非変性条件で安定化させることができます。融解温度は、二本鎖の安定性を評価するために使用する特性ですが、クロマトグラフィー条件が二本鎖の安定性にどのように影響するかは完全には理解されていません4。 圧力、温度、および共溶媒によって、見かけの融解温度が大きく変化することがあります。
siRNA 標準試料(アニール処理した 25 mer と 27 mer の RNA 鎖の混合物)を使用して、二本鎖の安定性に対する温度の影響を試験しました。55 ℃ 未満では、単一のピークが見られました。60 ℃ では、非常に幅の広いゾーンがクロマトグラムに現れ、その保持時間は、シャープな単一ピークと比較して大幅に減少していました(データは示していません)。温度が 65 ℃ を超えると、クロマトグラムに 2 つのシャープなピークが見られました。これらの結果は、この siRNA が 60 ℃ で一本鎖に解離し始めることを示唆しています。図 3 に、siRNA の二本鎖の安定性に対する温度の影響を示します。
CRISPR sgRNA および脂質結合アンチセンスオリゴヌクレオチドの HILIC MS
オリゴヌクレオチドの HILIC メソッドの調整可能性に関する情報が収集できたので、次にこのような分離の質量分析検出への直接のハイフネーションを検討しました。BioAccord LC-MS システムを、CRISPR sgRNA 分子および脂質結合 ASO(脂質結合 ASO LC-MS 標準試料)の分析に適用しました。2 種類の独自の移動相組成を適用しました。sgRNA の場合、100 mM 酢酸アンモニウムを加えた水系溶離液を適用しました。脂質結合 ASO には、より低濃度の 25 mM の酢酸アンモニウムを使用しました。いずれの場合も、モノアイソトピックピークまたは生質量スペクトルの MaxEnt デコンボリューションから導出された精密質量測定を使用して直接解釈できる、有効なレベルのイオンシグナルが生成しました。
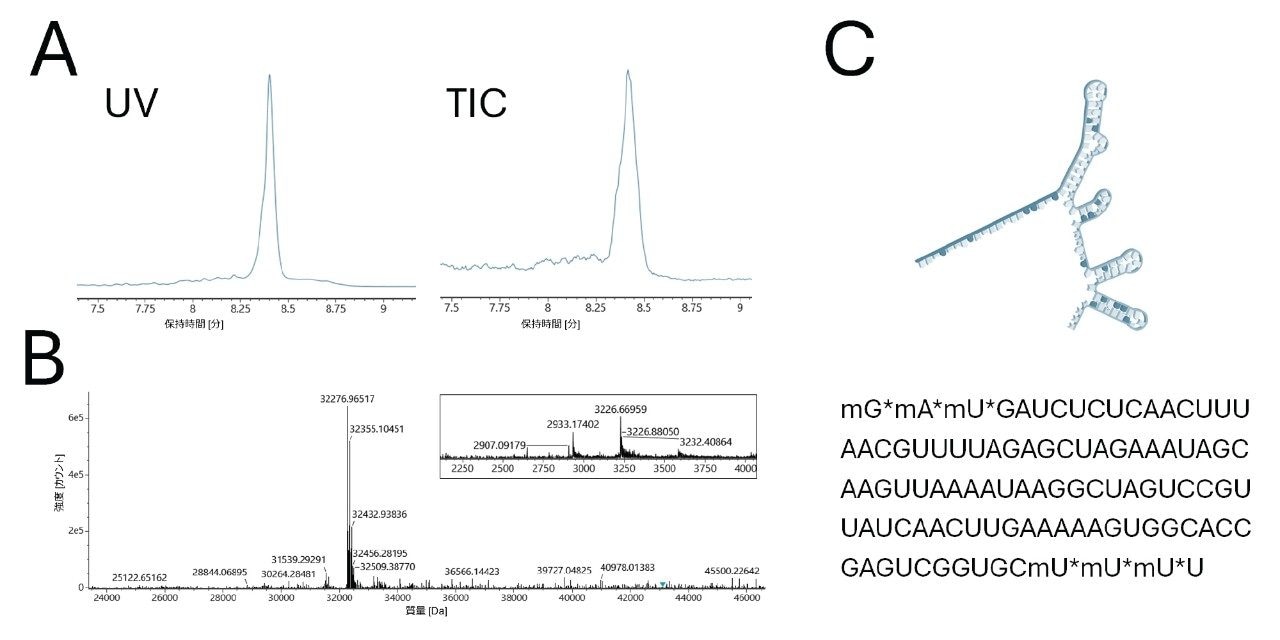
図 4 に、ヒト HPRT 配列と相補的な配列として合成された CRISPR sgRNA について収集された LC-MS データを示します。保持時間 8.4 分にピークが見られました。このピークについて取り込まれた MS 情報はパネル B に記載されています。挿入図には、合計生質量スペクトルと、チャージ状態が [M-11H+]11- および [M-10H+]10- のイオンのセットが示されています。このスペクトルから得られた MaxEnt デコンボリューション後の質量スペクトルから、sgRNA のインタクト分子量 32,276.97 Da が得られました。この値は 5' 末端および 3' 末端の修飾を含む sgRNA の配列とほぼ一致しています。
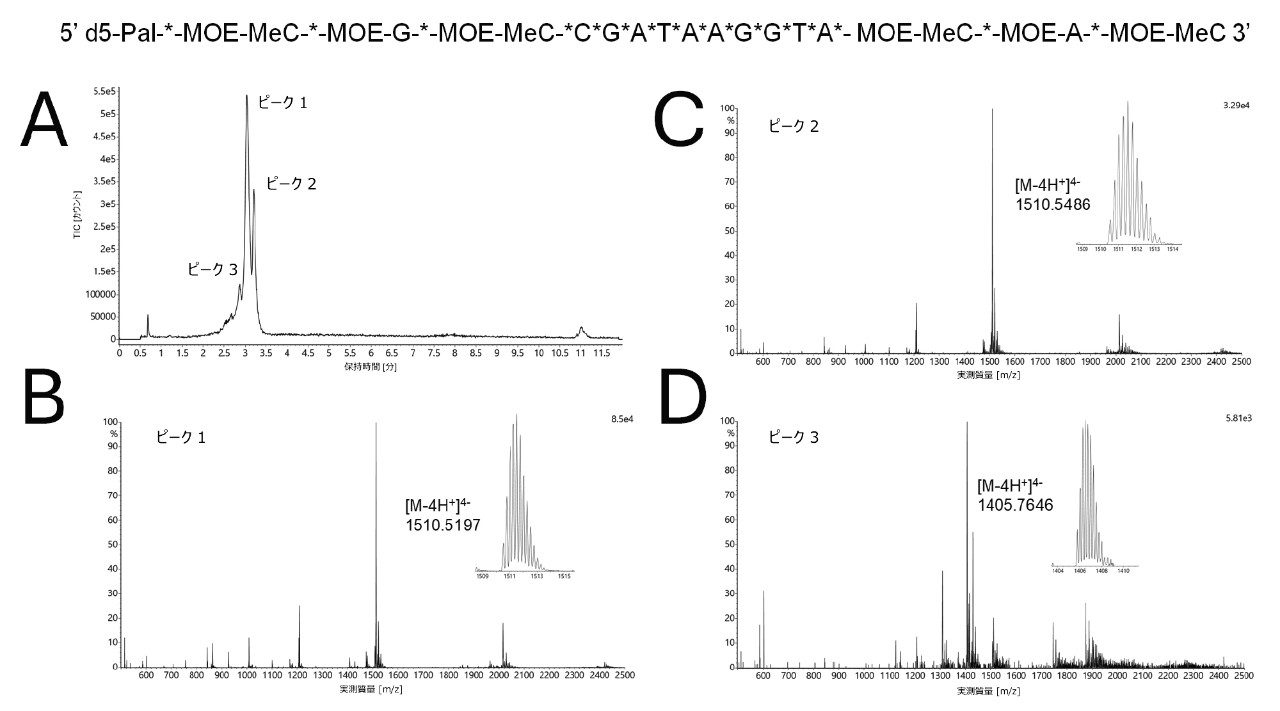
脂質結合 ASO の HILIC-MS 分析で得られた結果が図 5 に含まれています。脂質 ASO は、sgRNA とは対照的に、HILIC カラムでは強く保持されませんでした。これは、この分子が C16 脂質に結合した大幅に小さいオリゴヌクレオチドであることに起因すると考えられます。この脂質 ASO には、完全にホスホロチオ化された骨格と、その他の複数の修飾が含まれます。高温での分離により、サンプルのジアステレオマーに起因するピーク割れが減少することがわかりました。25 mM 酢酸アンモニウム溶離液を使用する最終的なメソッドでは、2 種類の同重体分子種が分離されました(ピーク 1 および 2)。さらに、質量が 419 Da 軽いサンプルの不純物が分離されました。
結論
この試験の結果から、オリゴヌクレオチドの分離および分析において、HILIC によって以下のメリットが得られることが示されました。HILIC メソッドには、オリゴヌクレオチド医薬品業界における分析法を進歩させる大きな可能性があることが実証されています。
- 分離条件における柔軟性:HILIC は変性条件と非変性条件の両方で使用でき、二本鎖オリゴヌクレオチドと一本鎖オリゴヌクレオチドの分離が可能になりました。このことは、複雑な RNA 構造および修飾の分析において重要です。
- 質量分析計との適合性:イオン対を含まない移動相を使用する HILIC の機能により MS との適合性が向上し、sgRNA のボトムアップの配列決定などのオリゴヌクレオチドの配列決定およびプロファイリングの感度と正確さが改善しました。
- 効率的な分析法開発およびブレークスルー効果への対処:実験計画法(DoE)の使用により、温度、塩濃度、グラジエントプログラムなどの重要なパラメーターの体系的な最適化が可能になり、頑健で再現性のある分離が得られました。ブラケット注入法によりサンプルのブレークスルーとピーク割れを効果的に低減することで、サンプルの完全性を損なうことなく、注入量を多くし、クロマトグラフィー性能を向上させることができました。
HILIC は、オリゴヌクレオチドの分離および分析のための、強力で汎用性の高いツールであり、オリゴヌクレオチド医薬品業界が直面している主要な課題に対処しています。より正確で高感度の分析法の需要が高まるにつれ、特にアンチセンスオリゴヌクレオチド(ASO)や RNA ベースの医薬品などの複雑な治療薬の台頭に伴い、HILIC は、高い分離能、MS 適合性、分析法開発における柔軟性を確保できる頑健なプラットフォームを提供します。HILIC の品質管理、配列決定、不純物プロファイリングへの応用が、次世代のオリゴヌクレオチド医薬品開発を加速する上で重要な役割を果たすと考えられます。
Waters、MassPREP、ACQUITY、UPLC、Empower、GTxResolve、BEH、QuanRecovery、MaxPeak、BioAccord、および IonHance は、Waters Technologies Corporation の商標です。DryLab は Molnar Institute の商標です。その他すべての商標はそれぞれの所有者に帰属します。
参考文献
- M. Gilar, B.M. Koshel , R.E. Birdsall, J. Chromatogr.A 1712 (2023) 464475.https://doi.org/10.1016/j.chroma.2023.464475
- A. Goyon, M.S. Blevins, J.G. Napolitano, D. Nguyen, M. Goel, B. Scott, J. Wang, S.G. Koenig, T. Chen, K. Zhang, J. Chromatogr.A 1708 (2023) 464327.https://doi.org/10.1016/j.chroma.2023.464327
- H. Lardeux, K. Stavenhagen, C. Paris, R. Dueholm, C. Kurek, L. De Maria, F. Gnerlich, T. Leek, W. Czechtizky, D. Guillarme, M. Jora, Anal.Chem.96 (2024) 9994–10002.https://doi.org/10.1021/acs.analchem.4c01384
- M. Gilar, S. Redstone, A. Gomes, J. Chromatogr.A 1733 (2024) 465285.https://doi.org/10.1016/j.chroma.2024.465285
- P.A. Lobue, M. Jora, B. Addepalli, P.A. Limbach, J. Chromatogr.A 1595 (2019) 39–48.https://doi.org/10.1016/j.chroma.2019.02.016
- P. Anand, M. Koleto, D.R. Kandula, L. Xiong, R. MacNeill, Bioanalysis 14 (2022) 47–62.https://doi.org/10.4155/bio-2021-0216
- A. Goyon, B. Scott, K. Kurita, C.M. Crittenden, D. Shaw, A. Lin, P. Yehl, K. Zhang, Anal.Chem.93 (2021) 14792−14801.https://doi.org/10.1021/acs.analchem.1c03533
- B. Bobaly, V. D’Atri, A. Goyon, O. Colas, A. Beck, S. Fekete, D. Guillarme, J. Chromatogr.B 1060 (2017) 325–335.http://dx.doi.org/10.1016/j.jchromb.2017.06.036
- H. Lardeux, V. D’Atri, D. Guillarme, TrACs 176 (2024) 117758.https://doi.org/10.1016/j.trac.2024.117758
- R. Perez-Robles, S. Fekete, Róbert Kormány, N. Navas, D. Guillarme, J. Chromatogr.A 1713 (2024) 464498.https://doi.org/10.1016/j.chroma.2023.464498
- S. Fekete, M. Imiolek, M. Lauber, Platform Ion Pairing RPLC Method for Oligonucleotides Using High Throughput 20 mm Length Columns, Waters Application Note, 720008460, August 2024.
720008602JA、2024 年 11 月