GTxResolve™ Premier SEC 1000 Å 3 µm カラムの速度論的効率および分取範囲
要約
この試験では、GTxResolve Premier SEC 1000 Å 3 µm カラムの速度論的効率および分取範囲について検討しました。このカラムは、細胞・遺伝子治療薬の原薬および製剤の分析用に設計された高度な分離ツールです。今回、DNA ラダーおよび RNA ラダーを使用して一連の基本的な比較試験を行っています。粒子径が 3 µm と小さくポアサイズが非常に大きい(1000 Å)充塡剤を使用したこのカラムでは、従来の 5 µm カラムと比較して、分離が大幅に改善しています。この新しいカラムテクノロジーのメリットを、同等のカラム効率を維持しつつより迅速かつよりハイスループットの分析を実現する、分離度を 30 ~ 40% 向上させる、という 2 つの方向性のうちのいずれかで適用することができます。新規治療薬の SEC 分析(特に mRNA やその他の核酸ベースの医薬品の凝集体および完全性分析)の必要性が増していることから、サイズ排除クロマトグラフィーのこのような進歩は、時宜を得ていると言えます。
アプリケーションのメリット
- 従来の 5 µm カラムと比較して、より迅速かつハイスループットな分析
- 二次的(非特異的)な静電相互作用が低減するため、高分子種(HMWS)の分析の信頼性が向上
- 分析時間または動作圧を固定した場合、新規の GTxResolve Premier SEC 1000 Å 3 µm カラムは、従来の 5 µm カラムよりも体系的に優位
はじめに
サイズ排除クロマトグラフィー(SEC)は、バイオ医薬品のサイズバリアントの特性解析に従来から用いられている分析法であり、新規治療薬の分析においてもますます重要になっています。高分子種の定量は、核酸などの多くの新規遺伝子治療薬における重要な品質特性の 1 つです1。 mRNA ワクチンに対して提案されている純度要件の 1 つとして、SEC による凝集体の測定が挙げられます。このクロマトグラフィーが、他のクラスの RNA ベースおよび DNA ベースの医薬品の品質評価において重要な役割を果たすことが期待されています2。
ペイロード遺伝物質のサイズは、100 ヌクレオチドの CRISPR ガイド RNA から 4700 bp の DNA ウイルスベクターゲノムまで、アプリケーションに応じてさまざまです。より一般的に使用される核酸の長さは 500 ~ 5000 ヌクレオチドです。例えば、ワクチンとして使用される長さ 4500 nt の COVID-19 mRNA は約 25 nm(流体力学的半径)です3。 このような分子種の SEC 分離には、ポアサイズが適度に大きいカラムを使用する必要があります。ただし、長さと溶液中での流体力学的半径の関係が不明瞭であることや、SEC カラムの公称ポアサイズの報告に関するカラムメーカー間の違いのため、適切な SEC カラムの選択は通常、実験によって決定する必要があります。バイオ医薬品の分析に通常使用されるカラム(ポア直径 125 Å および 250 Å の充塡剤)は、長さ 30 ~ 150 残基のオリゴヌクレオチドにしか適さない可能性があることがわかっています4。 ポアサイズが異なるカラムを比較した最近の試験では、1000 Å の充塡剤によってこのようなサイズの非複製 RNA に最適な分離が得られる可能性があると示唆されています5。
ワイドポアカラム(500 Å 超、超ワイドポアと呼ばれることもある)は通常、大きな粒子(5 µm 以上)としてしか入手できないため、分析のスループットが制限され、目的の分離を得るには長いカラム型式を使用することが必要になります。すると、分析に必要なサンプルの量が多くなり、多くの場合、これらの新規治療薬(特に新規の個別化 mRNA がんワクチン)のルーチン分析が制限されます6。
一方、核酸は、ポリ陰イオンという性質を有するため、吸着による二次的相互作用の影響を受けやすく、SEC 分析を成功させるにはこの相互作用を制限する必要があります。最良の結果および最も正確なサイジングの測定を実現するには、不活性な充塡剤のケミストリーに加えて、低吸着性カラムハードウェアを使用することが必要です。
このような問題に対処するため、MaxPeak™ HPS Premier ハードウェアに充塡した、ポアサイズ平均 1000 Å で粒子径がより小さい(3 µm)PEO 結合粒子の使用を検討し、高分子核酸ラダーサンプルで試験しました。このアプリケーションノートでは、新規の GTxResolve Premier SEC 1000 Å 3 µm カラムの分取範囲およびその核酸の分離における使用について説明します。さらに、このカラムの速度論的効率を検討し、これを同様のポアサイズの従来の 5 µm カラムと比較します。検量線、理論段数、分離インピーダンス、および速度論的性能の限界を体系的に比較したところ、新しい粒子および関連するカラムテクノロジーを使用した場合の方が優れていることがわかりました。
実験方法
サンプルおよび移動相の調製
dsDNA 50 ~ 1350 ラダー(Waters 製品番号:186010778)のアリコートを 1000 µL のヌクレアーゼフリー水に濃度が 0.1 mg/mL になるように再溶解し、100 bp dsDNA ラダー(New England Biolabs、N3231S)、低分子量範囲 ssRNA ラダー(New England Biolabs、N0364S)、および ssRNA ラダー(New England Biolabs、N0362S)をヌクレアーゼフリー水で 10 倍希釈して、これらをバイアルに移した後、SEC カラムに直接注入しました。
注:高度に凝集したサンプルを注入する場合は、注意が必要です。SEC カラムに注入する前に、遠心分離機で微粒子を除去する、および/またはサンプルをシリンジフィルターでろ過することを推奨します。
LC 条件
|
LC システム: |
ACQUITY™ H-Class Plus Bio システム、バイナリーソルベントマネージャー(BSM)搭載 |
|
検出: |
ACQUITY UPLC™ TUV 検出器(5 mm チタンフローセル装着)、260 nm および 230 nm、(2 ポイント/秒) |
|
バイアル: |
MaxPeak HPS を採用した QuanRecovery™ 12 × 32 mm スクリューネックバイアル、300 µL、100 本入り(製品番号:186009186) |
|
カラム: |
GTxResolve Premier SEC 1000 Å 3 µm カラム、150 × 4.6 mm(製品番号:186010735) |
|
カラム温度: |
25 ℃ |
|
サンプル温度: |
6 ℃ |
|
注入量: |
1.0 ~ 5.0 µL(流速に合わせてスケーリング) |
|
流速: |
0.05、0.1、0.175、0.25、0.40、0.50、0.60 mL/分 |
|
移動相: |
2x PBS バッファー、0.2 µm 滅菌ろ過済み (移動相の使用期限は 2 日間以内、あるいは細菌の増殖を防ぐアジドを使用)。 |
結果および考察
液体クロマトグラフィー分離の速度論的性能(速度および効率)を向上させるための一般的な戦略として、さらに小さな粒子を充塡したカラムを使用することが挙げられます。2 µm 以下の多孔性粒子を充塡した逆相(RP)カラムが 2004 年に発売され、2 µm 以下の SEC カラムが 2011 年に導入されました。このカラムにより、「超高速 SEC」(UHP-SEC)という新たな用語が導入されました。非常に微細な粒子を使用する背後にある概念としては、充塡済みカラムにおける渦分散(一般的な理論段高の式の A 項)と、SEC カラムのポア内に留まっている液相とカラム空隙容量内の移動相の間で溶質が分配される際の物質移動に対する抵抗(C 項)の両方を低減することにより、分離効率が向上されます。現在では、さまざまな 150 ~ 300 Å の SEC カラムが、2 µm 以下および 3 µm 以下の型式で市販されています7。 しかし、小さい粒子の場合、高圧が発生します。圧力は、粒子直径の二乗の逆数に比例して、カラムに沿って発生します。ポアの直径が非常に大きい(超ワイドポア)SEC カラムは、ポアがより小さいカラムよりも、機械的安定性が低いことが知られています。そのため、高い圧力機能を有する超ワイドポア粒子を製造するのは困難です。したがって、市販の超ワイドポア SEC カラム(500 Å または 1000 Å)のほとんどは 5 µm の粒子を用いており、その結果、速度論的性能が低下しています。GTxResolve Premier SEC カラムは、高い圧力機能と優れた速度論的性能を併せ持つ初めての 3 µm、1000 Å カラムです。このカラムは、粒子径とポアサイズのバランスが取れているため、さまざまな種類のサンプルに対して幅広い動作条件が有効になります。
検量線および分取範囲
新規の GTxResolve Premier SEC 1000 Å、3 µm カラムのサイジング範囲を、市販の 5 µm シリカ 1000 Å 粒子のカラムと比較しました。50 bp および 100 bp の DNA ラダー、および ssRNA と低分子量範囲の ssRNA のラダーを注入して、検量線を作成しました。その結果を、ベースペア数(対数スケール)対分子種のノーマライズした溶出ボリューム(Ve,n)としてプロットしました。SEC の検量線から基本的に、ポアサイズの分布(PSD)およびカラムのアクセス可能なポアの平均ポアサイズがわかります8。 したがって、この方法は、異なる SEC カラムのポア構造(形態)を比較するのに適しています。
2 本のカラムで得られた検量線を比較すると、DNA ホモログと ssRNA ホモログの両方について、高い類似性が見られました。図 1 に、DNA ラダーで得られた検量線を示します。
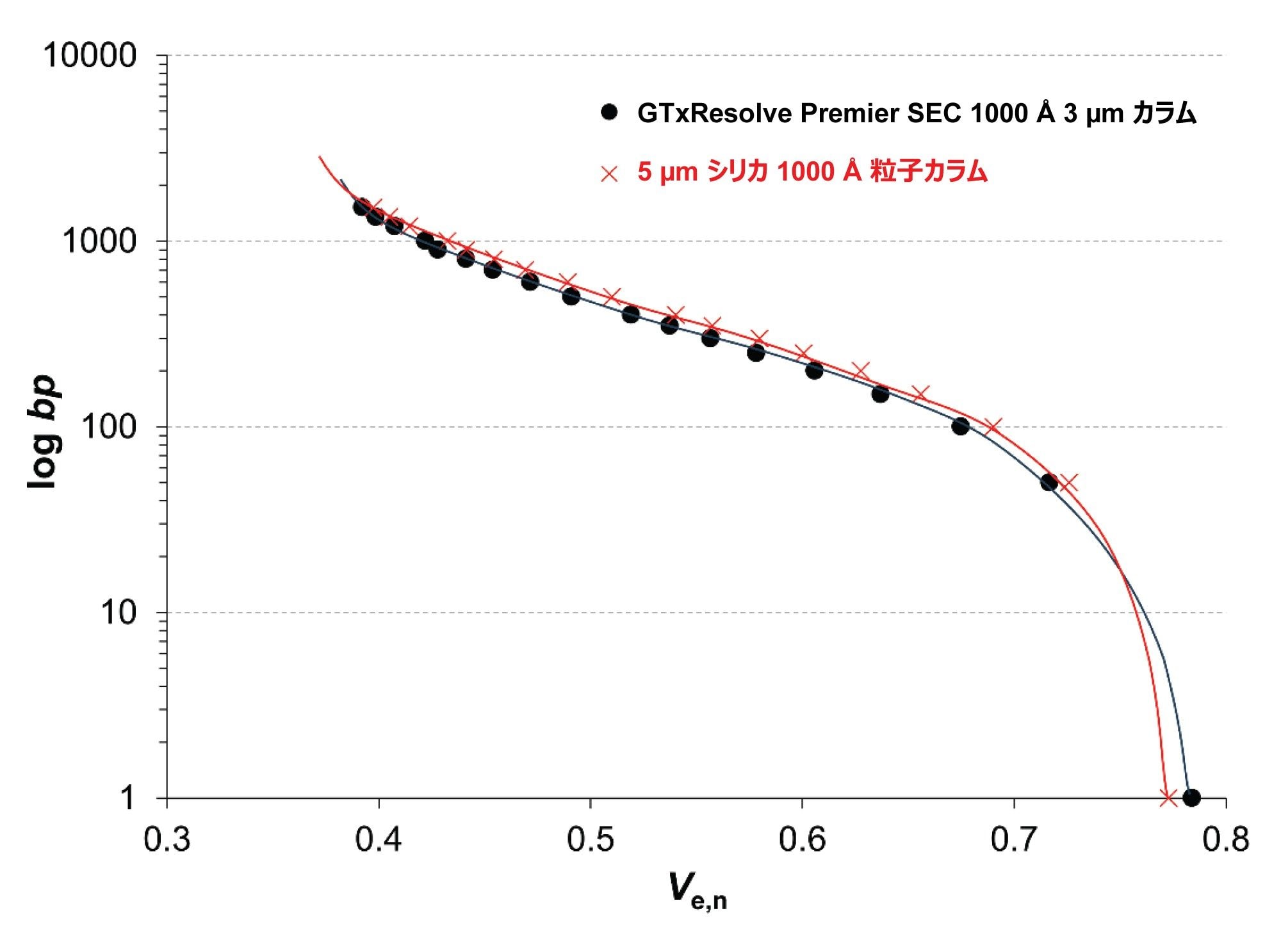
検量線の右端は、充塡剤の総多孔性(または総浸透性)によって決まります。GTxResolve Premier SEC カラムの方が多孔性がわずかに高いことがわかりました(Ve,n = 0.773 対 0.784)。
排除限界(検量線の左端)と浸透限界の間の溶出ウィンドウは多くの場合、カラムの「分取範囲」または「サイジング範囲」と呼ばれます。この比較により、2 本のカラムの分取範囲はほぼ同一であることが示唆されました。検量線の傾きは PSD の結果です。傾きが非常に類似していることから、この 2 つの材料の PSD も同様であると考えられます。曲線の垂直位置は、ポア直径の平均によって決まります。ここでもその位置が非常に類似しているため、2 つの充塡剤の平均ポア直径は非常に類似していることになります。
結論として、検量線の比較により、2 本のカラムのサイジング範囲、アクセス可能なポア直径の平均、および予想される選択性が非常に類似していることが示唆されました。変曲点は、DNA のサイズ約 350 ~ 400 bp または ssRNA のサイズ約 3 kb にあります。これらのサイズは、溶出ウィンドウの中央に対応します(ここで分配係数 KSEC は約 0.5)。
達成可能な理論段高と理論段数
カラムの速度論的効率を、まず理論段高曲線を使用して評価しました。流速の範囲を設定し(0.05 ~ 0.60 mL/分)、得られた理論段高(H)値を移動相の線速度(u)の関数としてプロットしました。幅広い溶質のサイズをカバーするため、100、300、500、1350 bp DNA について H - u 曲線をプロットしました。GTxResolve Premier SEC 1000Å、3 µm カラムでは、所定の流速で、5 µm シリカ 1000 Å カラムよりも平均して 1.5 ~ 2 倍低い H 値になりました。つまり、同じ長さのカラムを比較すると、3 µm カラムは、理論段数値(N)が 1.5 ~ 2 倍高く、したがって分離度が約 1.2 ~ 1.4 倍高くなります(Rs は √N に比例するため)。つまり、5 µm カラムより長さが 1.5 ~ 2 分の 1 の 3 µm カラムでも、同じ効率と分離度が維持されています。したがって、分析時間を 1.5 ~ 2 倍短縮することができます。
カラムの浸透性および分離インピーダンス
カラムの性能を比較する場合は、理論段数だけでなく、所定の理論段数(N)を得るのに必要な圧力(Δp)も考慮することが重要です。特に SEC にはこのことが当てはまり、圧力および摩擦に関連するアーティファクトを避けるために、低圧力条件が推奨されます。一般的な理論段高の曲線では浸透性が考慮されていないため、他の性能特性も試験する必要があります。
流速試験においては、カラム圧を記録し、システム圧に対して補正しました。次に、カラムの浸透性を計算しました。GTxResolve Premier SEC 1000 Å、3 µm カラムでは K = 1.22 × 10-10 cm2、5 µm シリカカラムでは K = 3.72 × 10-10 cm2 になりました。
全体的なカラムの性能は、達成可能な理論段高、カラムの浸透性、分析時間によって決まり、分析時間はカラムのデッドタイム(t0)に関連します。したがって、H-u 曲線のみでは、達成可能な分離時間に関する情報は得られず、到達可能なピーク幅に関する情報しか得られません。分離インピーダンス(E、Knox によって導入され、有効カラム抵抗と呼ばれることがある)は、ピーク幅、分析時間、カラム浸透性を同時に考慮するための複合的な評価尺度です9。 以下の計算式が多くの場合使用されます。
E = (t0∙∆p)/(N2∙η) = H2/K
(式 1)
ここで、η は移動相の粘度です。E は無次元であり、その値が低いほど分離が改善されます。図 2 に、低分子(100 bp)DNAおよび高分子(1350 bp)DNA について得られた分離インピーダンスのプロットを線速度の関数として示します。
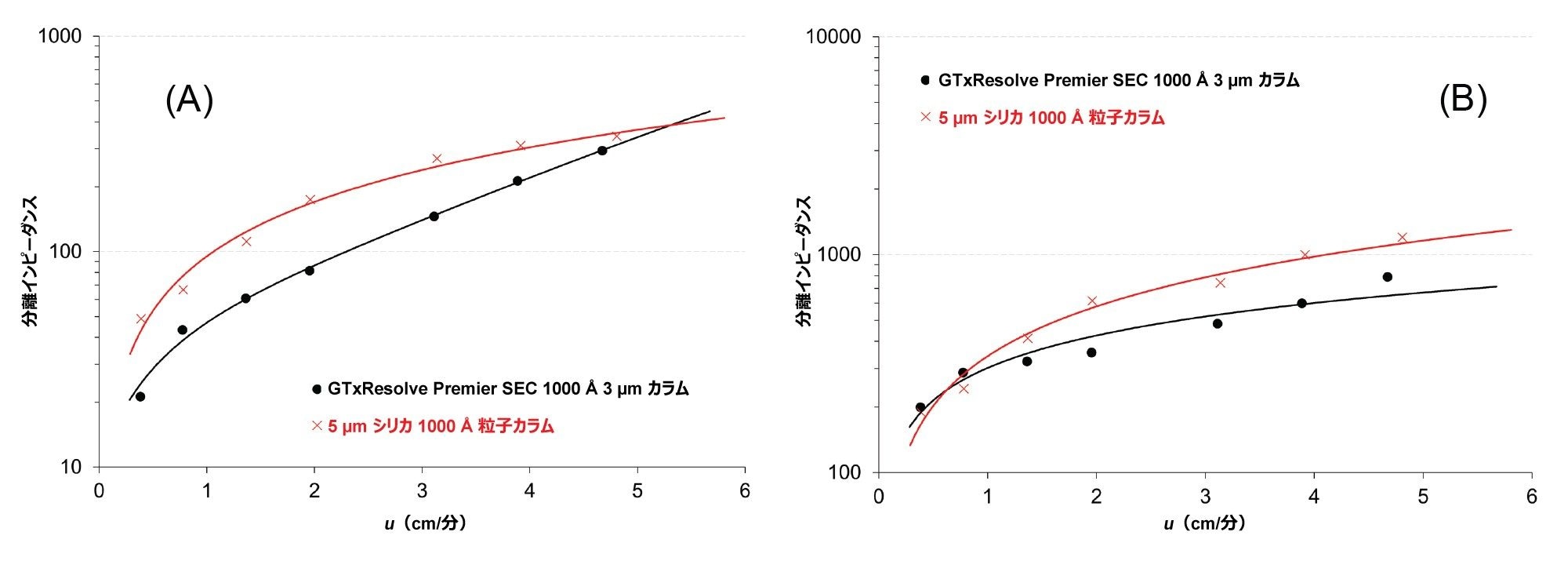
100 bp DNA の場合、3 µm カラムでは E = 20 ~ 300、5 µm カラムでは E = 50 ~ 350 になりました。3 µm カラムは、特に低流速領域で最大のメリットが認められました。
高分子 DNA の場合、3 µm カラムおよび 5 µm カラムのインピーダンス値はそれぞれ、E = 200 ~ 800 および E = 200 ~ 1200 でした。この高分子 DNA の場合、3 µm カラムは、高流速で特に有利と思われます。分取範囲全体にわたり、すべての分析種について同じ傾向が認められました。
速度論的プロットの比較
最後に、カラム圧が一定の場合に何が起こるかを検討することも重要です。この種の分析により、特定の分析時間内に目的の結果を得るためにどの種類のカラムを使用すべきかについてのヒントが得られます。
Poppe は、理論段数(N)に対して Plate time(1 理論段を与えるのに必要な時間、t0/N)をプロットする「Poppe プロット」を提案しました10。 これは、分離速度と分離効率の間の兼ね合いを視覚化できる優れたツールです。後に Desmet は、カラム圧が一定の場合(Δpfixed)のさまざまな充塡剤の速度論的性能をマッピングするための一般的なツールとして、「速度論的プロット」理論を導入しました11。 達成可能な理論段数および対応する分析時間(t0)を推定するために以下の式が提案されています。
N = (∆pfixed)/η (K/(u∙H))
(式 2)
t0 = (∆pfixed)/η (K/u2)
(式 3)
代表的な例を図 3 に示します。カラムを Δp = 100 bar で使用した場合の、100 bp および 1350 bp の DNA 分子種についての速度論的性能限界のプロットを示しています。
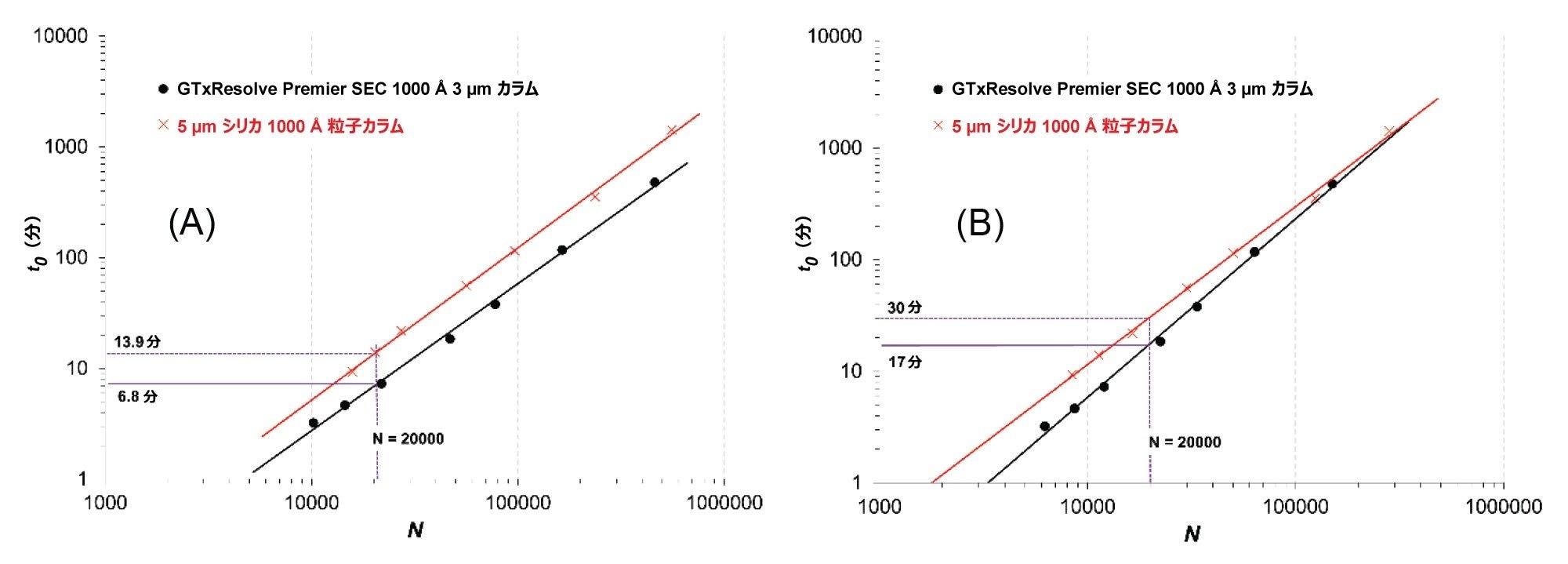
このように、3 µm のカラムは、分析時間の面で常に 5 µm シリカ 1000 Å 粒子カラムよりも優れています。このことは、黒色の曲線がどのように赤色の曲線の下を通っているかを見ると最も明確にわかります。
低分子 DNA(100 bp)の場合、3 µm カラムを使用すると分析時間 6.8 分以内に N = 20000 を達成でき、5 µm カラムを使用した場合は 13.9 分でこれと同じ効率が得られます。
高分子 DNA(1350 bp)の場合、20000 段が、3 µm カラムおよび 5 µm カラムでそれぞれ 17 分および 30 分で得られます。
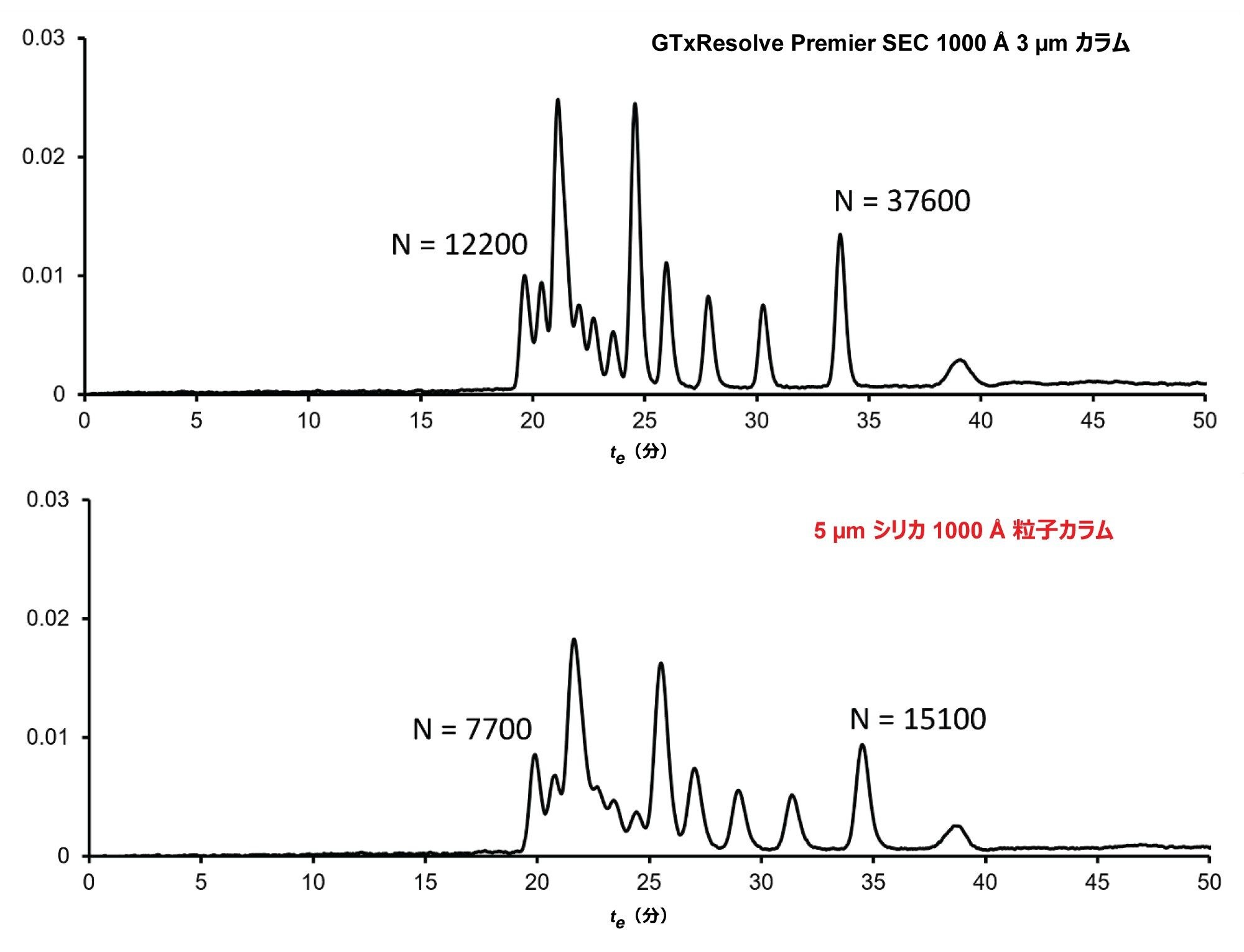
例:DNA ラダーの分離
図 4 に、カラムを F = 0.05 mL/分で使用した場合に DNA ラダーについて得られたクロマトグラムの例を示します。最初に溶出したピーク(1500 bp DNA)と最後に溶出したピーク(100 bp DNA)について理論段数を示しています。GTxResolve Premier SEC 1000 Å、3 µm カラムでは、5 µm シリカ 1000 Å 粒子カラムと比較して、約 2 倍高い理論段数が得られました。3 µm 粒子カラムを使用したことで、すべてのピーク対の間の分離が向上していることは明らかです。
結論
インタクト mRNA、LNP 凝集体、プラスミド DNA のトポロジーなどの細胞・遺伝子治療薬の製剤および原薬の迅速かつ効率的な分離用に設計された新規の超ワイドポア 1000 Å、3 µm カラムが導入されました。GTxResolve Premier SEC 1000Å 3 µm カラムの独自の小さい粒子径と高い機械的安定性により、新たなレベルの分離効率が得られます。理論段高、分離インピーダンス、速度論的プロットの分析により、この新規のカラムは、他の従来の 5 µm の充塡剤と比較して、カラム効率が全体的に優れていることが示されました。
参考文献
- Fekete S., Doneanu C., Addepalli B., Gaye M., Nguyen J., Alden B., Birdsall R., Han D., Isaac G., Lauber M., Challenges and Emerging Trends in Liquid Chromatography-Based Analyses of mRNA Pharmaceuticals, J. Pharm.Biomed.Anal. 224 (2023) 115174, https://doi.org/10.1016/j.jpba.2022.115174.
- Analytical Procedures for mRNA Vaccine Quality (Draft Guidelines) - 2nd Edition, Posting Date: 28-Apr-2023, Access Date: 28-Feb-2024.https://www.uspnf.com/notices/analytical-procedures-mrna-vaccines-20230428.
- Tan Y.Z., Qiao L.Z., Wang S.S., Zhang J., Qian J., Zhu M., Yao S.J., Lin D.Q., Enhanced Adsorption Performance of Varying-Length mRNA on Oligo dT Affinity Resins Through Optimal Pore Size and Grafting, Biochem. Eng.J. 203 (2024) 109213, https://doi.org/10.1016/j.bej.2023.109213.
- Gilar M., Doneanu C., Gaye M.M., Liquid Chromatography Methods for Analysis of mRNA Poly(A) Tail Length and Heterogeneity, Anal.Chem. 95 (2023) 14308–14316.https://doi.org/10.1021/acs.analchem.3c02552.
- De Vos J., Morreel K., Alvarez P., Vanluchene H., Vankeirsbilck R., Sandra P., Sandra K., Evaluation of Size-Exclusion Chromatography, Multi-Angle Light Scattering Detection and Mass Photometry for the Characterization of mRNA, J. Chromatogr. A 1719 (2024) 464756.https://doi.org/10.1016/j.chroma.2024.464756.
- Weber J. S., Carlino M.S., Khattak A., Meniawy T., Ansstas G., Taylor M.H., (...) & Zaks T., Individualised Neoantigen Therapy Mrna-4157 (V940) Plus Pembrolizumab Versus Pembrolizumab Monotherapy in Resected Melanoma (Keynote-942): A Randomised, Phase 2B Study.The Lancet, 403 (2024) 632–644.https://doi.org/10.1016/S0140-6736(23)02268-7.
- Goyon A., Beck A., Colas O., Sandra K., Guillarme D., Fekete S., Evaluation of Size Exclusion Chromatography Columns Packed With Sub-3 μm Particles for the Analysis of Biopharmaceutical Proteins, J. Chromatogr. A 1498 (2017) 80–89.https://doi.org/10.1016/j.chroma.2016.11.056.
- Harlan J.E., Picot D., Loll P.J., Garavito R.M., Calibration of Size-Exclusion Chromatography: Use of a Double Gaussian Distribution Function to Describe Pore Sizes, Anal.Biochem.224 (1995) 557-563.https://doi.org/10.1006/abio.1995.1087.
- Bristow P.A., Knox J.H., Standardization of Test Conditions for High Performance Liquid Chromatography Columns, Chromatographia 10 (1977) 279–289.https://doi.org/10.1007/BF02263001.
- Poppe H., Some Reflections on Speed and Efficiency of Modern Chromatographic Methods, J. Chromatogr.A 778 (1997) 3–21.https://doi.org/10.1016/S0021-9673(97)00376-2.
- Desmet G., Clicq D., Gzil P., Geometry-Independent Plate Height Representation Methods for the Direct Comparison of the Kinetic Performance of LC Supports With a Different Size or Morphology, Anal.Chem. 77 (2005) 4058–4070.https://doi.org/10.1021/ac050160z.
720008284JA、2024 年 3 月