Confirm Sequence でターゲットを絞ったデータ非依存的フラグメンテーションデータを用いた、合成オリゴヌクレオチドの迅速で効率的かつシンプルな配列確認
要約
このアプリケーションノートでは、合成オリゴヌクレオチドの配列確認に waters_connect™ CONFIRM Sequence アプリケーションソフトウェアを使用して得られる効率を、QTOF(Vion™ 質量分析計)MS/MS およびベンチトップ型 TOF(BioAccord™ 質量分析計)MSE(DIA)データを用いる手動アプローチと比較して実証します。
アプリケーションのメリット
はじめに
近年、低分子医薬品およびタンパク質医薬品の有力な代替物として、合成オリゴヌクレオチドが登場してきました1。 市場に投入されるオリゴヌクレオチド医薬品の量は増加しており、信頼性が高く頑健な品質管理を行える分析法が求められています2,3。 上市されている合成オリゴヌクレオチドの主要なカテゴリーは、アンチセンスオリゴヌクレオチド(ASO)とサイレンシング RNA(siRNA)の 2 つです4,5。 いずれの場合も、通常は長さが 50 モノマーより短いオリゴヌクレオチドを使用します。
質量分析は、このような分子(特に修飾されたヌクレオシドおよびバックボーンが使用されている場合)を特性解析するための究極のアプローチという定評を得ています6-8。 ターゲット MS/MS フラグメンテーションによる合成オリゴヌクレオチドの配列の特性解析は、十分に理解されて注釈付けされているフラグメンテーション経路を使用する推奨アプローチです9。 オリゴヌクレオチドのサイズ、プリカーサーの荷電状態、コリジョンエネルギーの関係に関する最近の調査により、規制申請に必要な配列確認を行うための最適な条件が得られています10。
このワークフローの重要な限界として、得られた複雑なデータセットを手動で解析すること、およびディスカバリーおよび初期開発に通常使用する質量分析計における質の高い MS/MS フラグメンテーションの生成に必要な経験豊富な分析者の可用性が限られていること、の 2 つがあります。これらの限界により、製造ラボおよび品質管理ラボにおける MS/MS ベースの配列確認のダウンストリームへの活用が制限されます。
今回、waters_connect™ CONFIRM Sequence アプリケーションソフトウェアを使用して、修飾合成オリゴヌクレオチド医薬品である CpG7909 の質の高い配列確認データが、短時間で得られることを実証しました11。 さらに、MS の専門知識が限られているユーザー向けに設計されたベンチトップ型 BioAccord™ システム(UPLC™-TOF-MS)を使用して、合成オリゴヌクレオチド製造をルーチンに品質管理できる可能性を実証しました。これらの結果を、QTOF MS(Vion™ MS、通常オリゴヌクレオチドの特性解析に使用される装置)で得られた MS/MS データと比較しています。
合成オリゴヌクレオチドから生じるフラグメントイオンを手動で予測することは困難です。CONFIRM Sequence アプリケーション内のライブラリー機能により、構造情報を下位成分レベル(塩基、糖、リンカー)まで使用して、カスタムオリゴヌクレオチド配列を作成、管理、保存することができます。アプリケーションに事前にインストールされている既定のモノマーのエントリーで CpG7909 を十分作成できますが、このライブラリーは、モノマー作成およびカスタムフラグメンテーション規則の設定において、必要に応じて高度にカスタマイズすることができます。
手作業では数時間かかる解析が、数分の自動データ解析に短縮されています。グラフ表示、問題のあるデータ領域の自動フラグ付け、結果の効率的なキュレーションにより、結果のレビューが容易になりました。CONFIRM Sequence のカスタマイズされたターゲット同位体クラスタリングアルゴリズムを使用することで、詳細なフラグメントイオンマッチが生データに直接注釈付けされ、高い信頼性が得られます。
実験方法
サンプルの説明
CpG7909(配列 dT*dC*dG* dT*dC*dG* dT*dT*dT* dT*dG*dT* dC*dG*dT* dT*dT*dT* dG*dT*dC* dG*dT*dT、d はデオキシリボ核酸、* はホスホロチオエートバックボーンの修飾を示す)は、BioSprings 社内で固相合成により合成しました。CpG7909 を水で最終濃度 0.5 mg/mL に希釈しました。
LC 条件
|
LC システム: |
ACQUITY™ UPLC™ バイナリーソルベントマネージャー |
|
光学検出器: |
TUV |
|
カラム: |
ACQUITY UPLC Oligonucleotide BEH™、130Å、C18 1.7 µm 2.1 × 100 mm |
|
カラム温度: |
60 ℃ |
|
注入量: |
2 µL |
|
流速: |
0.3 mL/分 |
|
移動相 A: |
7 mM TEA、80 mM HFIP 水溶液 |
|
移動相 B: |
3.5 mM TEA および 40 mM HFIP 含有 50:50 メタノール:水 |
|
グラジエント: |
使用した LC グラジエントは、初期条件の 10%B バッファーで 0.2 分間、0.2 分~ 2.0 分で 10%B ~ 100%B、2.00 ~ 2.20 分は 100%B、2.20 ~ 2.30 分で 100%B ~ 10%B、2.30 分 ~ 3.00 分は 10%B でした。カラム温度は 60 ℃、流速は 0.3 mL/分でした。 |
MS 条件
BioAccord™ システム RDa™ を、m/z 50 ~ 2000 という低質量の取り込み範囲において、フラグメンテーションモードによるフルスキャンで使用しました。ESI の極性はネガティブ、スキャンレートは 1 Hz に設定しました。コーン電圧は 50 V に調整し、フラグメンテーションコーン電圧は、60 V または 70 V から 100 ~ 120 V まで急上昇させました。キャピラリー電圧は 0.8 kV、脱溶媒温度は 550 ℃、脱溶媒ガスの流量は最大 800 L/時に設定しました。
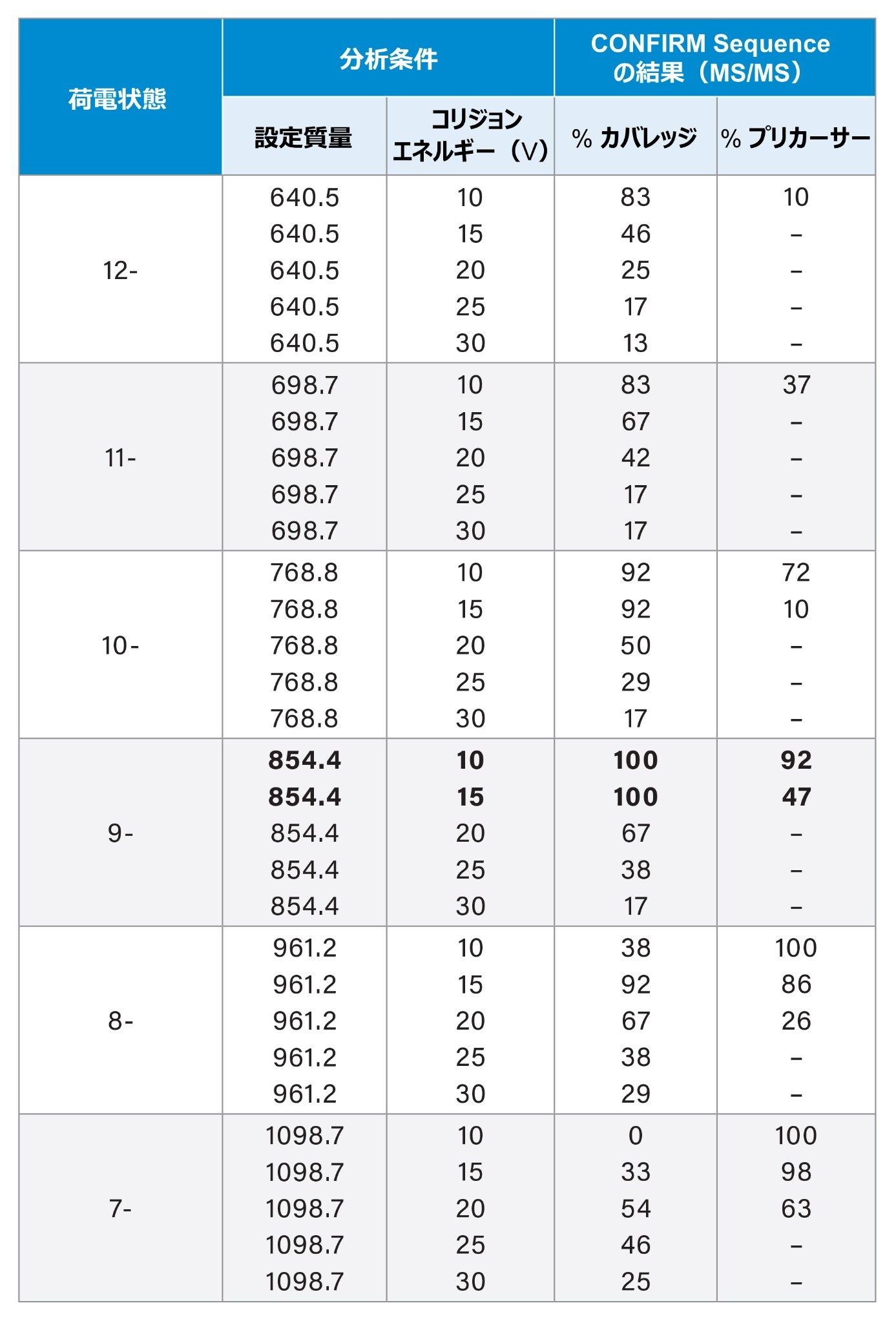
Vion IMS QTOF は、感度モードで標準的な透過が得られる ESI ネガティブ極性で使用しました。イオン源の種類は ESI に設定し、イオン源温度 120 ℃、脱溶媒温度 400 ℃、脱溶媒ガス流量 800 L/時間、コーンガス流量 50 L/時間、キャピラリー電圧 2.0 kV、サンプルコーン電圧 60 V、イオン源オフセット電圧 100 V に設定しました。MS/MS 取り込みは m/z 50 ~ 2000 の範囲、スキャン時間は 0.5 秒で、低プリカーサー分解能に設定しました。以下のプリカーサーイオン(m/z 単位の値)を選択しました:640.5(荷電状態:12-)、698.7(荷電状態:11-)、768.8(荷電状態:10-)、854.4(荷電状態:9-)、961.2(荷電状態:8-)、1098.7(荷電状態:7-)。コリジョンエネルギーは、各プリカーサーイオンについて、10、15、20、25、30 V とさまざまでした。
解析の設定
|
CONFIRM Sequence のメソッド設定(MS/MS) |
|
|
PPM 誤差: |
20 |
|
強度カットオフ(カウント): |
20 |
|
同位体類似性しきい値: |
65 |
|
同位体強度しきい値: |
65 |
|
CONFIRM Sequence のメソッド設定(MSe) |
|
|
PPM 誤差: |
30 |
|
強度カットオフ(カウント): |
200 |
|
同位体類似性しきい値: |
65 |
|
同位体強度しきい値: |
55 |
結果および考察
QTOF MS を使用したオリゴヌクレオチドの配列決定では、最適な結果を得るために、荷電状態の選択およびコリジョンエネルギーのスクリーニングを行います。荷電状態に応じてコリジョンエネルギーを最適化して、低エネルギーでの部分的フラグメンテーションおよび高エネルギーでの過剰フラグメンテーション(配列の内部切断およびフラグメントのフラグメンテーション)を回避する必要があります。
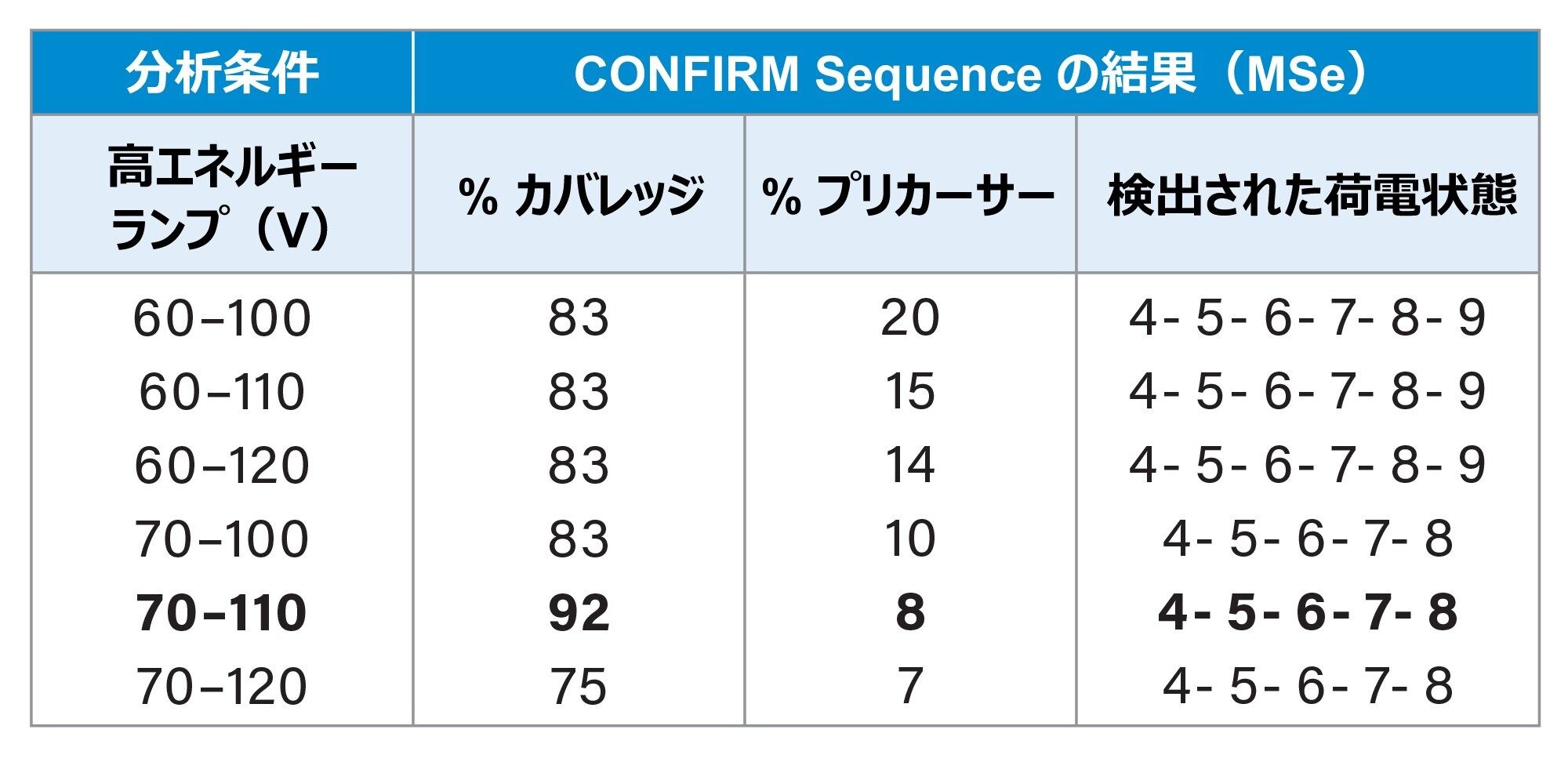
今回の被験分子 CpG7909(免疫賦活作用のあるアジュバント合成オリゴヌクレオチド医薬品)では、Gawlig および Rühlが報告している、低荷電状態には高エネルギーが必要であるという結果(2023)も、MS/MS(表 1)および MSe(表 2)の両方で見られます10。
フラグメンテーションの調整を追加するという戦略では、フラグメンテーションデータに存在する残留プリカーサーの量をターゲットにします。残留プリカーサーのシグナルが存在しない場合は、分子が過剰にフラグメンテーションされているか、あるいはその条件では荷電状態が不安定である可能性があります。CONFIRM Sequence に表示されるプリカーサーの割合の減少は、いずれの場合もコリジョンエネルギーの増加と相関しており、コリジョンエネルギーの増加が過剰なフラグメンテーションの原因であることを示している可能性もあります。これらを考慮すると、特定の荷電状態に対して最も高いコリジョンエネルギーを示すプリカーサーの存在は、最適の設定であることを示しており、プリカーサーの割合の指標を使用してこの最適化された結果を得ることができると考えられます。
MSe フラグメンテーションでは、最小量の残留プリカーサー(5 ~ 10%)が生じる最も高いコリジョンエネルギーが、ここに示すデータでは最適です。MS/MS フラグメンテーションでは、最適条件は荷電状態によって異なります。同じ荷電状態での低エネルギーと比較して低いシーケンスカバレッジとプリカーサーの割合を示すコリジョンエネルギーは、過剰フラグメンテーションの兆候です。MS/MS 取り込み条件の最適化では、プリカーサーの割合が 60% 未満になるまでコリジョンエネルギーを高くすることを試み、より低エネルギーよりもシーケンスカバレッジが高い最低のプリカーサーの割合が得られるまで、エネルギーを増大させ続ける必要があります。
手動解析と CONFIRM Sequence の比較
手動ワークフローで得られるデータのレビューには時間がかかり、ユーザーミスが発生しやすいという問題があります。CONFIRM Sequence のレビューステップは、シーケンスカバレッジを分かりやすく提示するように設計されており、生データに直接注釈付けされた各フラグメントイオンマッチを手動でレビューする機能があります。
CONFIRM Sequence でのデータ解析は迅速であり(今回示したデータはすべて 5 分以内で解析)、専門家のユーザーが事前に設定することで、より経験の浅いユーザーでも専門家の監督下で大きなデータセットを解析することができます。さらに、waters_connect™ ソフトウェアプラットホームは、品質保証ラボの業務における規制遵守に必要なレポート作成、データインテグリティーの管理、メソッドのバージョン管理なでのコンプライアンスに対応しています。
CONFIRM Sequence アプリにより、再現性の高いフラグメントイオンマッチングを行うことができ、同重体フラグメントの割り当てがカバレッジの計算から除かれます。これにより、偽陽性の割り当てが減り、ユーザーによる手動でのデータ検証の負担が軽減します。
マッチングの忠実性の向上
高質量範囲にわたる修飾合成オリゴヌクレオチドの複雑なフラグメンテーションデータは、デコンボリューションにかなりの時間がかかる可能性があります(1 スペクトルあたり最大 20 分)。また、デコンボリューションした結果を手動で調査し、質量に基づいて予測フラグメントを割り当てるには、ある程度の専門知識が必要になります。
CONFIRM Sequence アプリでは、インテリジェントなターゲット同位体クラスタリングアルゴリズムを使用することで、手動アプローチおよびノンターゲットアプローチの問題が軽減されます。CONFIRM Sequence ではターゲットを絞ってデータを解析します。つまり、各フラグメントイオンの予測同位体クラスターを生データに対して直接検索し、さまざまなスコアリング指標に基づいてマッチングします。生データに対するマッチをアプリケーションでレビューして、フラグメントイオンの割り当てを裏付けることができます。
さらに、一連のフラグメントイオンをモノマーの割り当ての確認に使用する方法を管理する一連の論理規則が適用されます。その一例として、塩基喪失フラグメンテーションイオン(例:a-b-イオン)の使用の場合が挙げられます。この場合、イオンが衝突誘起解離フラグメンテーションの後に気相イオン/イオン転位を受けて、塩基成分が失われます。この喪失のため、フラグメンテーション部位の 5' に直接存在するモノマーの存在を確認するために、塩基喪失イオンのみを使用することは妥当ではありません。CONFIRM Sequence のロジックを考えると、代わりに n-1 シリーズのフラグメントイオンの内から使用するべきです(例:3 番目のモノマーの存在を確認するのに a-b3 イオンを使用するべきではありません。[x-]2 イオンシリーズの内から使用するべきです)。CONFIRM Sequence で使用されるこの割り当てのロジックは、アプリケーション内で確認されたモノマーが、予測可能なフラグメンテーションモデリングおよび実測可能なデータに基づくように設計されています。このソフトウェアには、それぞれのモノマー確認規則を詳細に説明しているヘルプファイルがあります。
非特異的なマッチ
CpG7909 には同重体であるさまざまな予測フラグメントイオンがあり、これらを配列の複数のセクションの確認に使用することは不誠実です。CONFIRM Sequence は、これらの非特異的なマッチにフラグ付けし、最初の配列確認のセットから除外しますが、ユーザーには、データをレビューするときに再度追加するという選択肢があります。例として、w1- イオンを戻して加えました。このイオンは、5' 末端と 3' 末端のモノマーが同一であるため、d1- と同重体です。この場合、これらのモノマーはいずれも配列の末端にあるため、この非特異的なマッチは許容可能と考えられます。ただし、CpG7909 の配列内には、配列の確認に合理的に使用できない複数の同重体フラグメントイオンがあり、一部の配列をカバーすることが困難になっています。しかし、このような非特異的なマッチは容易には確認できないため、手動で配列確認作業を行うことの危険性が浮き彫りになります。
ターゲット MS/MS とデータ非依存的取り込み(MSe)の比較
CONFIRM Sequence は、単一の MS/MS フラグメンテーションスペクトルでは 100% のシーケンスカバレッジ、BioAccord システムでデータ非依存的フラグメンテーションに基づく取り込み(DIA)を使用すると 92% のシーケンスカバレッジを示すことができます。MSe(DIA)取り込みのメリットは、設定が簡素化されていることです。これは、最適化された CE 設定にマッチするように設計されたコリジョンエネルギーランプで、すべてのプリカーサーイオンをフラグメント化するもので、ターゲット MS/MS 取り込みを使用して事前に決定する必要があります。これにより、ターゲットプリカーサーの荷電状態および実験計画を事前に選択して、コリジョンエネルギーを最適化する必要がなくなりました。
CONFIRM Sequence アプリでは、MS/MS 実験の最適化を支援するために、各 MS/MS 取り込みの結果が概要テーブルにまとめて表示されています(図 1)。ここには、組み合わせたシーケンスカバーレッジが、各スペクトルからの個々の寄与を表示するテーブルと並べて表示されます。これにより、すべてのデータにわたって最適なフラグメンテーション条件を直ちに決定することができ、最適な条件を得るには、どの荷電状態でより多くのエネルギーあるいはより少ないエネルギーが必要かに関する情報が得られます。
CONFIRM Sequence アプリでの個々の分析のフラグメンテーション結果のレビューは、各フラグメントイオン割り当ての注釈が付けられたスペクトルによって合理化されており、必要に応じて検索することも却下することもできます(図 2)。非特異的なフラグメントイオンの割り当ても、レビュー用にフラグ付けされます。
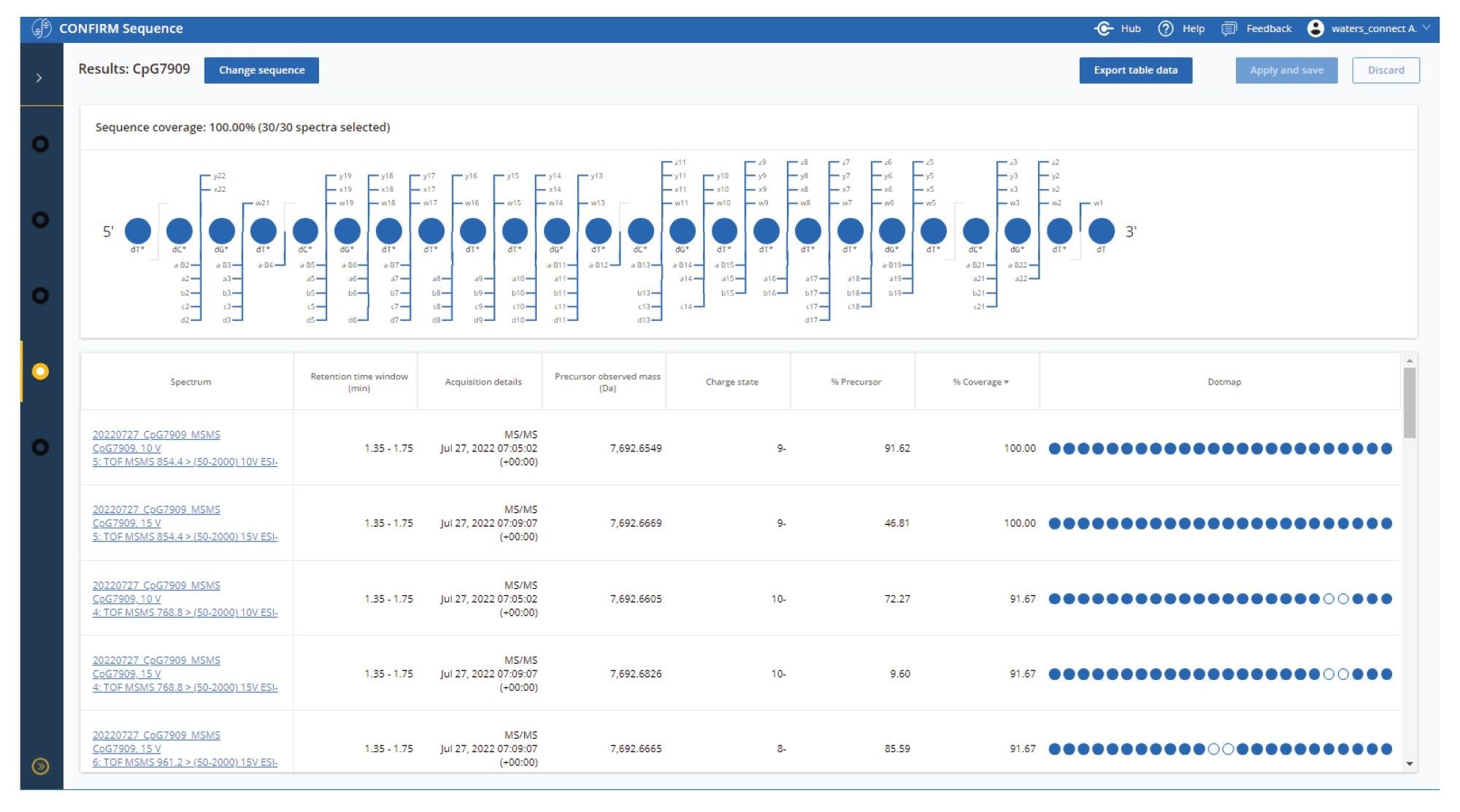
図 1. 結果を示す CONFIRM Sequence アプリケーションの結果の概要ページのスクリーンショット。ここに示すデータは、ターゲットプリカーサー(m/z)(640.5(-12)、698.7(-11)、768.8(-10)、854.4(-9)、961.2(-8)、1098.7(-7))を使用した CpG7909 に関する MS/MS 実験の結果です。コリジョンエネルギーは、各プリカーサーイオンについて、10、15、20、25、30 V とさまざまでした。組み合わせたシーケンスカバレッジの概要が Dotmap™ ビューに表示されています。ここでは、配列が色付きの丸印およびフラグメントイオンの「脚」で表されており、生データ中でマッチしたフラグメントイオンに基づいてモノマーが確認されていること(青色の丸印)を示しています。確認されていないモノマーは白色の丸印で表わされています。
テーブルには、個々の MS/MS スペクトルの結果が示されており、注入/チャンネル名、保持時間、取り込みの詳細、プリカーサーの実測質量、同定されたプリカーサーの荷電状態、検出された残留プリカーサーイオンの割合、確認されたモノマーの簡素化した Dotmap™ 表示で補足されたカバレッジに関する情報が記載されています。この表をシーケンスカバレッジで並べ替えると、2 種類の個別の MS/MS 条件でシーケンスカバレッジ 100%(コリジョンエネルギー 10V および 15V での荷電状態 -9)が得られることがわかりました。プリカーサーの割合は、10V での 91.62% から、15V での 46.81% に低下しています。20V を使用した場合、この荷電状態ではプリカーサーが検出されませんでした。このことは、このエネルギーで過剰なフラグメンテーションが発生したことを示します。
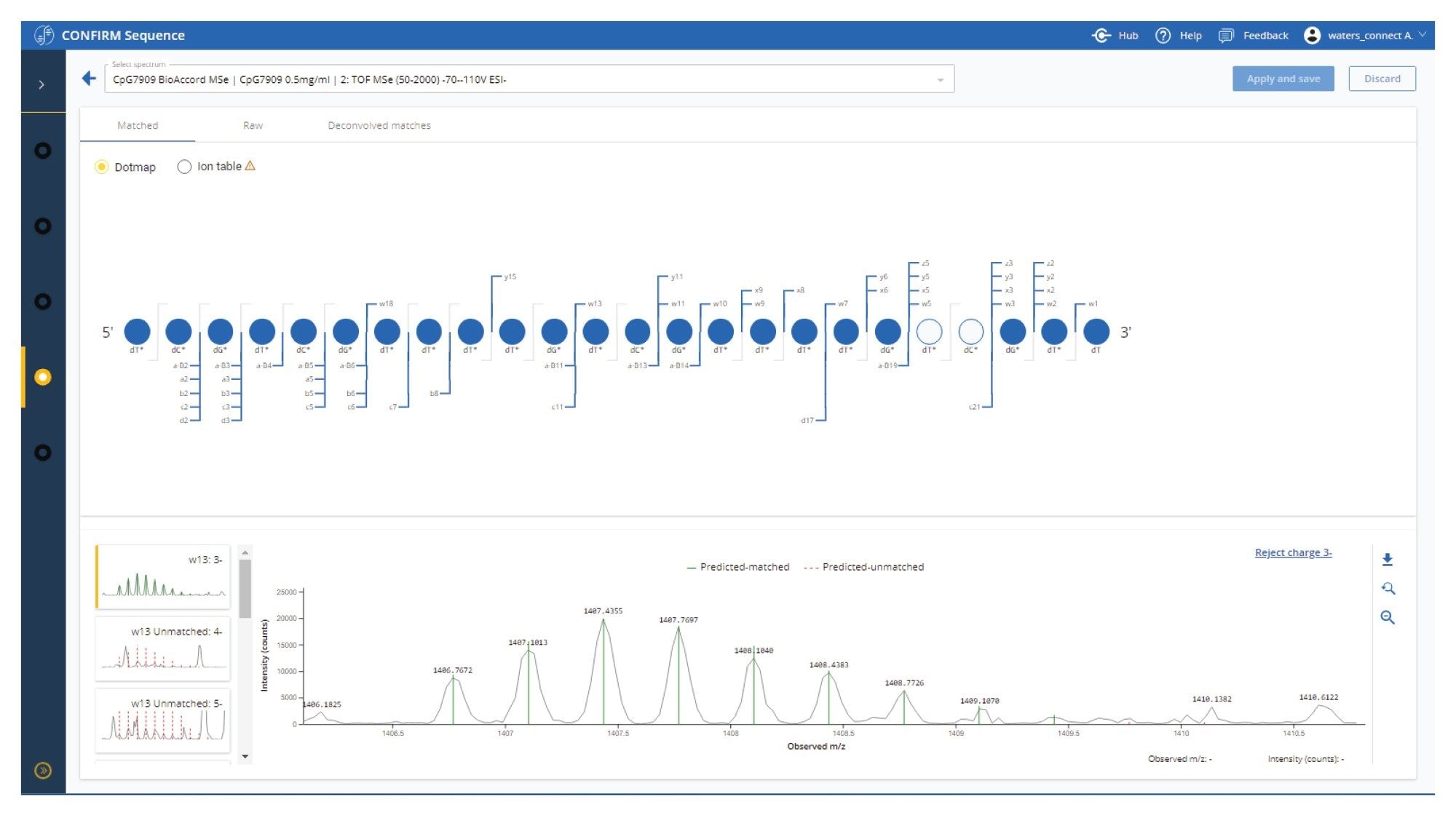
CpG7909(配列 dT*dC*dG* dT*dC*dG* dT*dT*dT* dT*dG*dT* dC*dG*dT* dT*dT*dT* dG*dT*dC* dG*dT*dT、d はデオキシリボ核酸、* はホスホロチオエートバックボーンの修飾を示す)。
結論
CONFIRM Sequence waters_connect™ アプリケーションは、ターゲット MS/MS データおよびデータ非依存的フラグメンテーションデータの両方に対して良好に機能します。
合成オリゴヌクレオチド配列確認のための手動デコンボリューションおよびフラグメント割り当ては、時間がかかり、ユーザーミスが発生しやすく、ユーザーエクスペリエンスに大きく依存します。対照的に、自動 CONFIRM Sequence ワークフローでは、手動ワークフローと同等の結果が得られるにもかかわらず、自動フラグメントマッチ、生データでの直接マッチング、解析速度の向上、シーケンスカバレッジの結果において同重体による非特異的なマッチが考慮されている、などのメリットがあります。
BioAccord は、コンプライアンス対応のプラットホームで使用する MS の専門家でないユーザー向けに設計された、設置面積が小さく、費用対効果の高い LC-MS システムであり、規制対象の製造ラボおよび品質ラボにおける自動データ解析の有用性がさらに拡がります。CONFIRM Sequence アプリは、BioAccord システムで得られた DIA フラグメンテーションデータを解析して注釈付けすることができるため、自動ワークフローにおける信頼性の高い割り当てが可能になります。この TOF MS システムによって得られる質の高い高エネルギーデータは、QTOF やその他のハイエンドの質量分析法の代替として使用できます。
分子の最初の特性解析において、優れたフラグメンテーション制御および感度を有する QTOF システムはこのようなルーチンの配列確認の課題には必要以上である可能性があります。両方のワークフローをサポートする CONFIRM Sequence アプリの有用性により、トレーニングの負担が軽減し、開発組織から製造組織および QC 組織への分子および分析法のよりスムーズな移行が可能になります。
参考文献
- Sharma, V.K., Watts, J.K.: Oligonucleotide Therapeutics: Chemistry, Delivery, and Clinical Progress.http://dx.doi.org/10.4155/fmc.15.144.7, 2221–2242 (2015).https://doi.org/10.4155/FMC.15.144
- Al Shaer, D., Al Musaimi, O., Albericio, F., de la Torre, B.G.: 2021 FDA TIDES (Peptides and Oligonucleotides) Harvest.Pharmaceuticals.15, (2022).https://doi.org/10.3390/PH15020222
- Musaimi, O. Al, Shaer, D. Al, Albericio, F., de la Torre, B.G.: 2020 FDA TIDES (Peptides and Oligonucleotides) Harvest. Pharmaceuticals.14, 1–14 (2021).https://doi.org/10.3390/PH14020145
- Dhuri, K., Bechtold, C., Quijano, E., Pham, H., Gupta, A., Vikram, A., Bahal, R.: Antisense Oligonucleotides: An Emerging Area in Drug Discovery and Development. J Clin Med. 9, 1–24 (2020).https://doi.org/10.3390/JCM9062004
- Rossi, J.J., Rossi, D.J.: siRNA Drugs: Here to Stay.Molecular Therapy.29, 431–432 (2021).https://doi.org/10.1016/J.YMTHE.2021.01.015
- McLuckey, S.A., Habibi-Goudarzi, S.: Ion Trap Tandem Mass Spectrometry Applied to Small Multiply Charged Oligonucleotides with a Modified Base.J Am Soc Mass Spectrom.5, 740–747 (1994).https://doi.org/10.1016/1044-0305(94)80006-5
- Mcluckey, S.A., Van Berkel, G.J., Glish, G.L.: Tandem Mass Spectrometry of Small, Multiply Charged Oligonucleotides.J Am Soc Mass Spectrom.3, 60–70 (1992).https://doi.org/10.1016/1044-0305(92)85019-G
- Abdullah, A.M., Sommers, C., Hawes, J., Rodriguez, J.D., Yang, K.: Tandem Mass Spectrometric Sequence Characterization of Synthetic Thymidine-Rich Oligonucleotides.Journal of Mass Spectrometry.57, (2022).https://doi.org/10.1002/JMS.4819
- Wu, J., McLuckey, S.A.: Gas-Phase Fragmentation of Oligonucleotide Ions.Int J Mass Spectrom.237, 197–241 (2004).https://doi.org/10.1016/J.IJMS.2004.06.014
- Gawlig, C., Rühl, M.: Investigation of the Influence of Charge State and Collision Energy on Oligonucleotide Fragmentation by Tandem Mass Spectrometry.Molecules 2023, Vol.28, Page 1169.28, 1169 (2023).https://doi.org/10.3390/MOLECULES28031169
- Cooper, C.L., Davis, H.L., Morris, M.L., Efler, S.M., Al Adhami, M., Krieg, A.M., Cameron, D.W., Heathcote, J.: CPG 7909, An Immunostimulatory TLR9 Agonist Oligodeoxynucleotide, as Adjuvant to Engerix-B® HBV Vaccine in Healthy Adults: A double-blind phase I/II study.J Clin Immunol.24, 693–701 (2004).https://doi.org/10.1007/S10875-004-6244-3/METRICS
謝辞
データ生成においてご支援頂いた BioSpring GmbH の Alexander Litz さんに感謝いたします。
waters_connect、Vion、BioAccord UPLC、ACQUITY および RDa は、Waters Technologies Corporation の商標です。
今回紹介した試験の BioSpring GmbH での実施を可能にしてくださった Hüseyin Aygün さんに感謝いたします。
Michael Rühl および Güngör Hanci は、BioSpring GmbH(ドイツ、フランクフルト Alt-Fechenheim 34、60386)の社員です。
720008079JA、2023 年 11 月