高感度イオン対 LC-MS を使用した PCR プライマーおよびプローブ試薬の質量確認および純度分析
体外診断(IVD)目的です。一部の国/地域では提供されていません。
要約
SARS-CoV-2 パンデミックにより、RT-qPCR キットを使用した感染検査が定期的に行われるようになりました。認定済みの高品質の qPCR 試薬を使用することで、この種の疾病モニタリングの取り組みにおいてメリットが得られます。このアプリケーションノートでは、最適化した IP-RPLC およびコンプライアンス対応のベンチトップ型 Tof 装置を用いた、SARS-CoV-2 CDC アッセイで使用される PCR 検査試薬の品質評価について説明しています。この感度の高いアプローチにより、わずか 10 pmol のヌクレオカプシドプライマー N1 と N2(フォワード配列とリバース配列の両方)および N1 プローブと N2 プローブから、純度について多くの情報が収集できました。すべての PCR 試薬についてシャープで対称的なピークが得られました。ネガティブイオンマススペクトルを取り込むことで、多重荷電分子種(最大 [M-10H]10-)が容易に観察され、RT-qPCR キット試薬の分子量を明確に決定できました。UV 検出および MS 検出を組み合わせて、試薬および未精製の粗製品の純度を評価しました。これに伴い、N2 プローブの質量が 42.9 シフトした不純物の興味深い例について検討しました。まとめると、高分解能で高感度の LC-UV-MS を組み合わせた手法により、掘り下げた情報が得られ、RT-qPCR 試薬を認定する上でのメリットがもたらされました。
アプリケーションのメリット
- ジイソプロピルエチルアミンのイオン対を PCR 試薬分析に適用して優れたクロマトグラフィー分離を実現し、高感度 UV-MS BioAccord システムを用いてオンライン MS 検出および精密質量ピーク同定を実現
- ACQUITY Premier クロマトグラフィーシステムを PCR 試薬の特性解析に適用
はじめに
世界保健機関(WHO)によると、現在の SARS-CoV-2 パンデミックによる死者数は既に 400 万人を超えています(2021 年 9 月現在)1。 世界中で 50 億回を超えるワクチン投与が行われましたが1、残念ながら、入手可能性や利用可能なワクチンの接種率に大きなばらつきが見られます。長期間にわたるウイルスのヒト間での伝播と、それに続く変異の蓄積により、病原体のゲノムが大幅に変化する可能性が増大し、伝播性および感染性が増大する可能性が高まっています2。現時点では、SARS-CoV-2 の蔓延を低減するための重要な要素として、定量的逆転写-ポリメラーゼ連鎖反応(RT-qPCR)による定期的な検査が挙げられます3,4。米国食品医薬品局(FDA)は、米国疾病予防管理センター(CDC)の 2019-nCoV RT-qPCR キットの緊急使用を許可しました。この検査はヌクレオカプシド RNA 遺伝子の 2 つの異なる場所(N1 および N2)の検出に基づいており、アッセイの検出限界は 1 µL あたり約 65 ウイルスコピーです4。重要な点として、アッセイの感度は、プライマー/プローブ結合領域のヌクレオチド置換3、RNA 抽出キットの試薬の品質4、およびオリゴヌクレオチド合成時のエラーに左右されます5。そのため、PCR 検査試薬の調査および確認のための分析法が重要になります。今回、イオン対逆相クロマトグラフィーに、飛行時間型(Tof)質量分析計を組み合わせて使用し、RT-qPCR プライマー/プローブキットの高純度評価を行いました。
実験方法
サンプル情報
SARS-CoV-2 アッセイでの直接使用向けの化学的に合成したオリゴヌクレオチド、および脱塩のみの粗製品をベンダーから入手しました。これらのサンプルを、ヌクレアーゼフリー 10 mM Tris、0.1 mM EDTA、pH 7.5 バッファー中に 10 pmol/μL になるように調製し、ポリプロピレン製 300 μL オートサンプラーバイアル(製品番号:186002639)に移しました。
LC 条件
|
LC システム: |
ACQUITY UPLC I-Class システム |
|
検出器: |
ACQUITY UPLC TUV 検出器 |
|
波長: |
260 nm |
|
カラム: |
ACQUITY Premier BEH C18、2.1 × 50 mm、130 Å、1.7 μm |
|
カラム温度: |
60 ℃ |
|
サンプル温度: |
4 ℃ |
|
注入: |
1 µL(10 pmol) |
|
流速: |
0.3 mL/分 |
|
移動相 A: |
0.1% N,N-ジイソプロピルエチルアミン(DIPEA)を IP 試薬とし、1% 1,1,1,3,3,3-ヘキサフルオロイソプロパノール(HFIP)を脱イオン水中に調製 |
|
移動相 B: |
0.0375% DIPEA、0.75% HFIP 含有 65:35 アセトニトリル:水 |
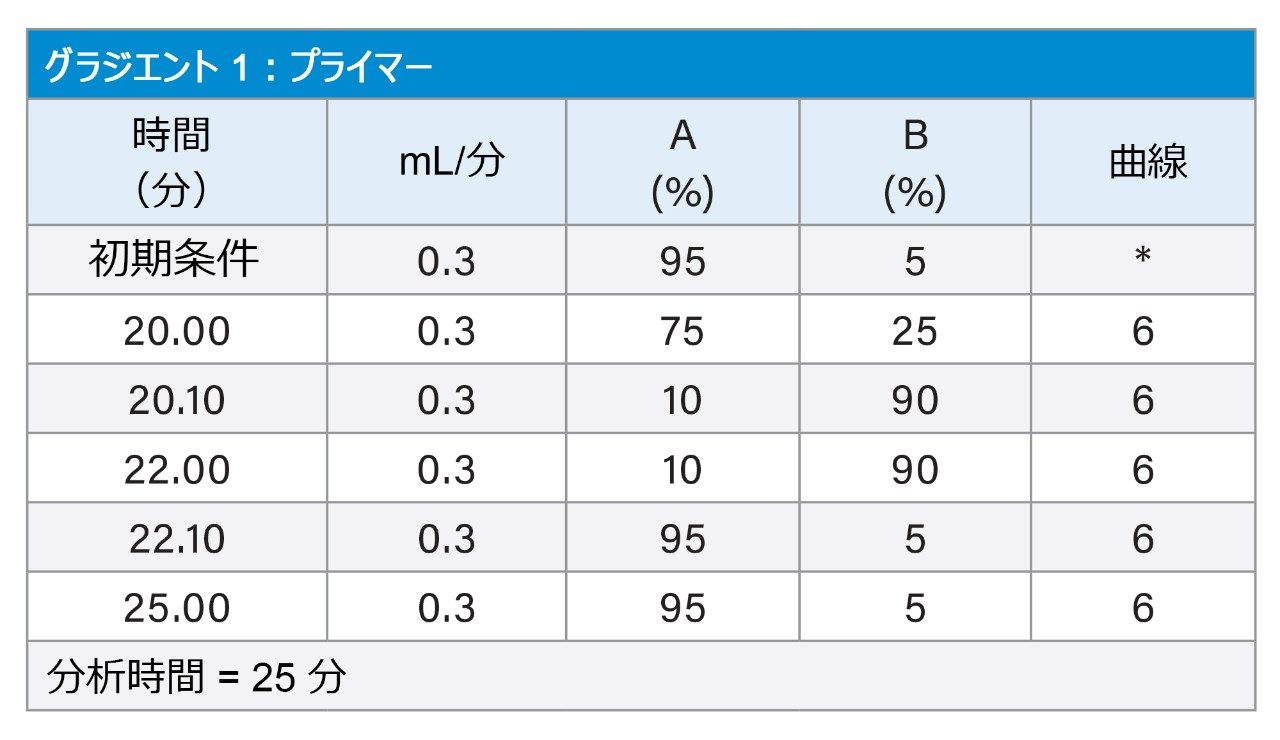

MS 条件
|
MS システム: |
BioAccord |
|
検出器: |
ACQUITY RDa 検出器 |
|
モード: |
フルスキャン |
|
極性: |
ネガティブ |
|
コーン電圧: |
40 V |
|
質量範囲: |
高(m/z 400 ~ 5000) |
|
スキャンレート: |
2 Hz |
|
キャピラリー電圧: |
0.80 kV |
|
脱溶媒温度: |
400 ℃ |
結果および考察
イオン対逆相クロマトグラフィー(IP-RPLC)では、移動相にイオン対試薬(通常はアルキルアミン)が含まれており、これが C18 固定相に吸着するため6-8、オリゴヌクレオチドの荷電と蛍光物質の疎水性がいずれも IP-RPLC における保持に寄与する、ミックスモード様の保持メカニズムが導入されます6-8。このアプリケーションで使用した移動相は、光学 UV 検出およびネガティブイオンモード質量分析のいずれにも対応しています8,9。
この試験では、ジイソプロピルエチルアミン(DIPEA)を IP 試薬とし、1,1,1-ヘキサフルオロイソプロパノール(HFIP)を添加した MS 対応の移動相システムを用いました。HFIP は、負に荷電した分析種の C18 固定相による保持と、エレクトロスプレーイオン化の間の荷電の濃縮プロセスを改善することで、IP-RPLC-MS 分析の感度を高めることが示されています6。このアプローチを実践するため、水性移動相およびアセトニトリル含有移動相を使用して、ACQUITY UPLC TUV 検出器を搭載した ACQUITY UPLC I-Class で用いるグラジエントを開発しました。オリゴヌクレオチドサンプル 10 pmol を ACQUITY Premier Oligonucleotide C18(2.1 × 50 mm、130 Å、1.7 µm)カラムに注入しました。UV 検出の後、溶出したオリゴヌクレオチド PCR 試薬のアイデンティティーを、ベンチトップ型 BioAccord LC-MS システムの ACQUITY RDa 検出器でネガティブイオンモード質量分析を用いて確認しました。
CDC RT-qPCR キットに含まれる 2019 SARS-CoV-2 のヌクレオカプシド N1 および N2 プライマー(フォワード配列とリバース配列の両方)は、20 分の 5 ~ 25% B グラジエントを用いて、11 分以内にカラムから溶出しました。UV および MS クロマトグラムの両方で、シャープで対称的なピーク(図 1)および再現性の高い保持時間(RT)が見られました。MassPREP オリゴヌクレオチド標準試料(製品番号:186004135)を 4 ヶ月の間隔を置いて同様の条件で分析したところ、保持時間 0.1 分以内の再現性が得られました。N1 プライマーおよび N2 プライマーのいずれにおいても、フォワードとリバースのオリゴヌクレオチドの RT の差は約 1 分でした。
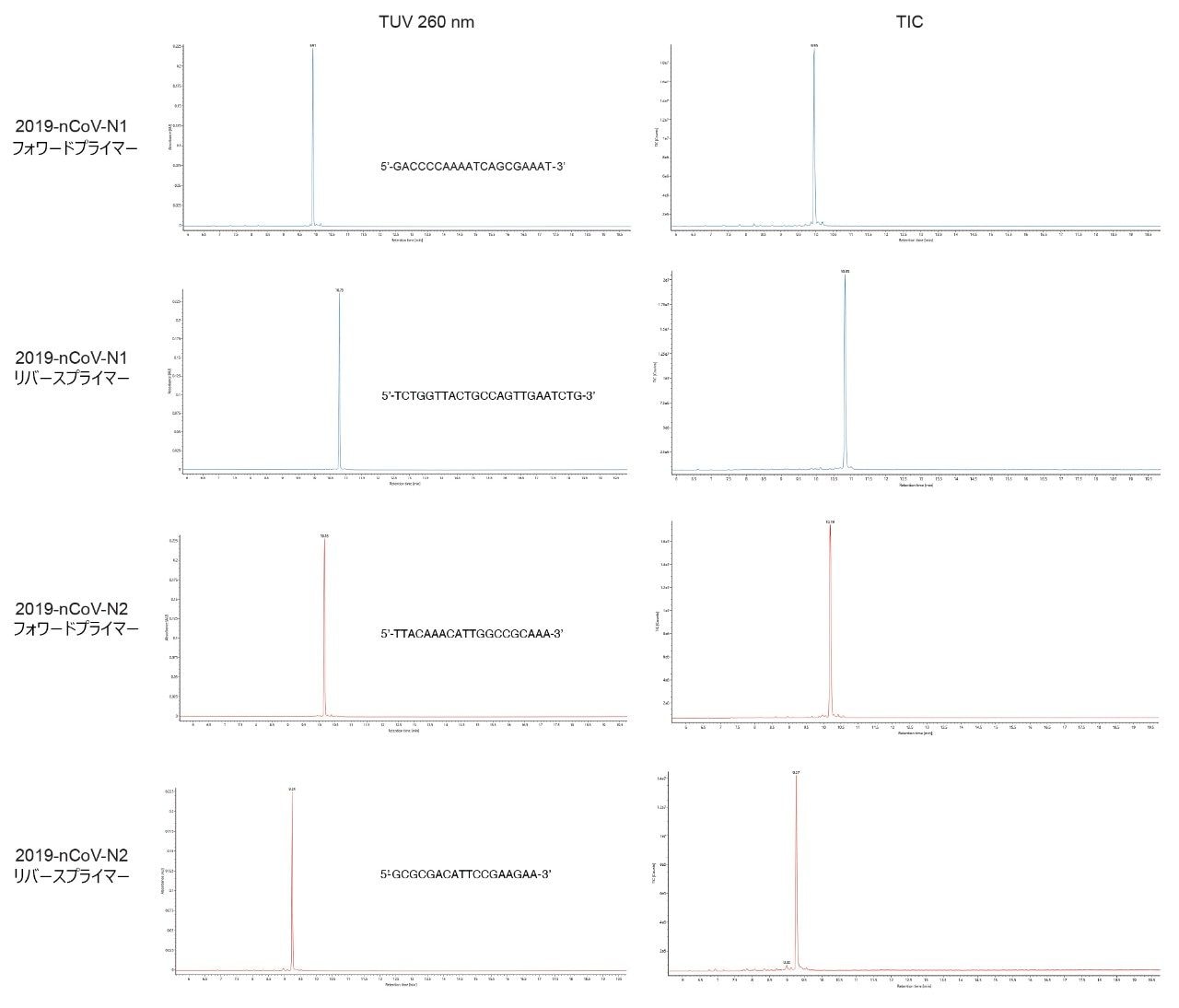
CDC RT-qPCR アッセイで使用したプローブは、オリゴヌクレオチド配列の 5’ 末端が蛍光物質、3’ 末端がクエンチャーで標識されています。qPCR アッセイの感度は、蛍光物質の適切な合成およびそのリンカーによるオリゴヌクレオチド配列への適切な付加により大きく左右されます8。 N1 プローブおよび N2 プローブでもシャープなピークが得られました(図 2)。グラジエントを 30% B までに修正し、より強く保持されるプローブがより確実に溶出されるようにしました。この最終溶出強度の変更により、プローブ溶出の保持時間が 20 分から 16 分に変わりました。これらの結果から、シングルクエンチャープローブの保持の挙動がわかります。3’ のクエンチャーに加えて、内部クエンチャーで標識したプローブも分析しました。これらのいわゆるダブルクエンチャープローブについて得られた分離を、図 2 の下のパネルに示します。別の内部クエンチャーで標識したプローブは、約 15 分という短い保持時間で溶出しました。
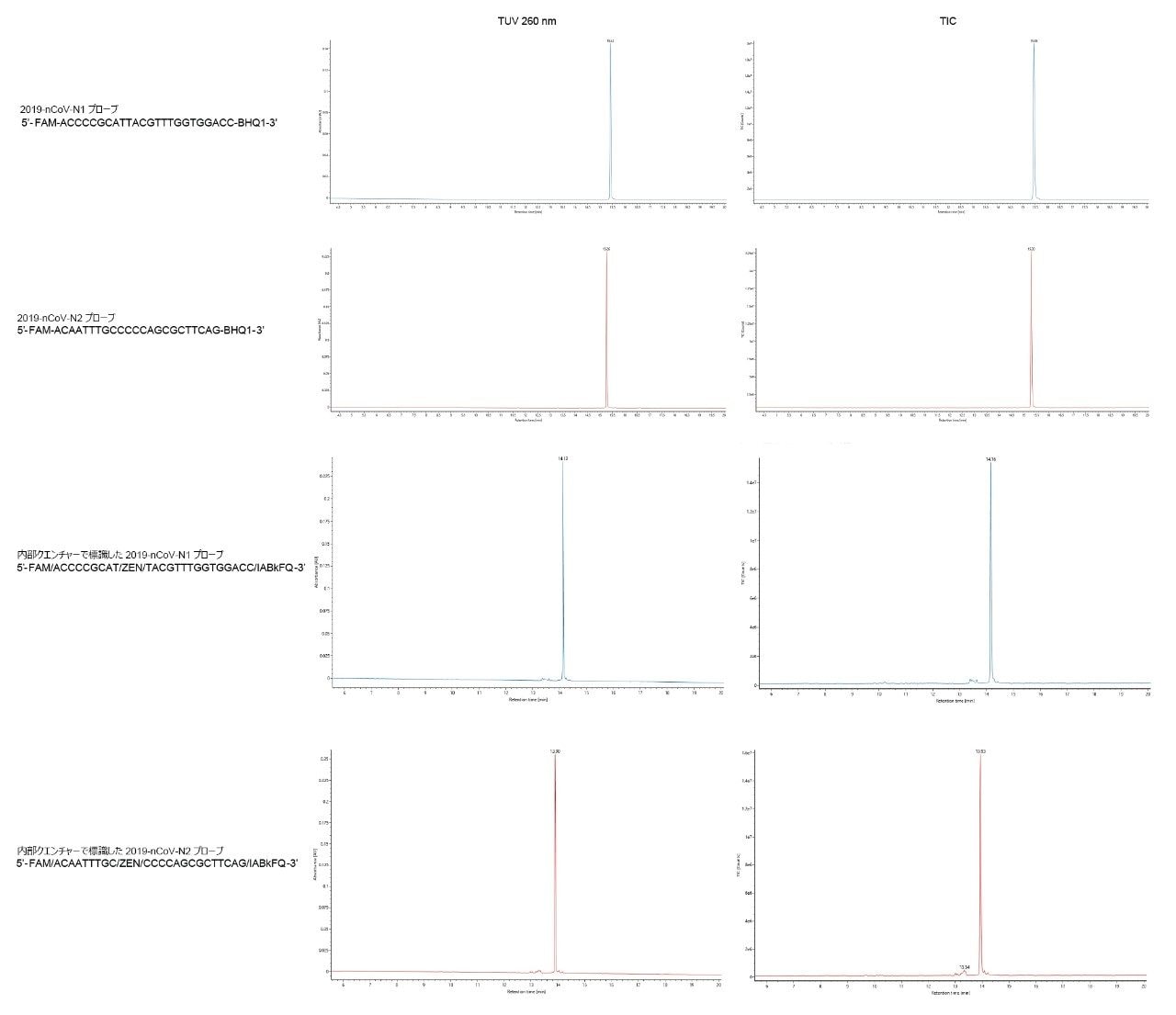
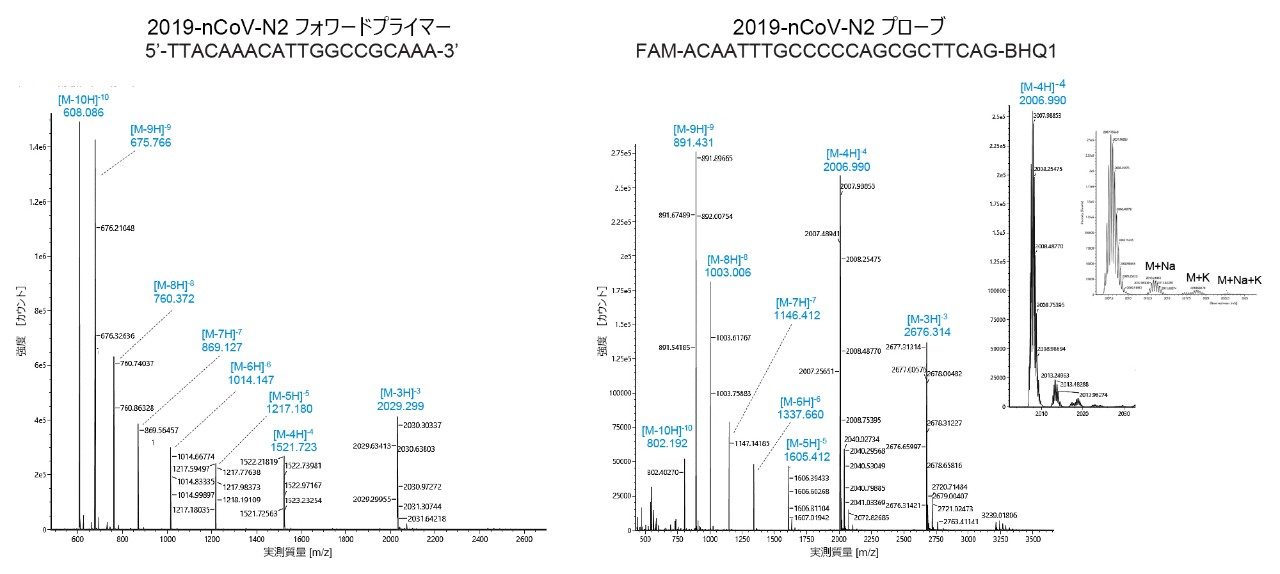
BioAccord ACQUITY RDa 検出器を用いた質量分析により、質量確認が可能になりました。260 nm での UV 検出の後、スキャンレート 2 Hz で m/z 400 ~ 5000 のネガティブモードのマススペクトルを取り込みました。コーン電圧およびキャピラリー電圧には既定値を使用しました(それぞれ 40 V および 0.80 kV)。脱溶媒温度に 400 ℃ を使用しました。ダイバートバルブは、1.5 分後に MS に設定し、22 分に再び廃液に戻しました。N2 フォワードプライマーおよび N2 プローブについてのマススペクトルの例を図 3 に示します。プライマーおよびプローブのマススペクトルはいずれも、多重荷電分子種(最大 [M-10H]10-)を示し、RT-qPCR キット試薬の精密質量が明確に測定できました(表 2)。図 3 の右パネルの挿入図は N2 プローブの [M-4H]4- イオンに対応しており、このスペクトルに見られるナトリウムおよびカリウムの付加イオンは、ベースピークシグナルの 10% を下回っています。EPFL オンラインカリキュレーターを用い、オリゴヌクレオチド配列に基づいて、プライマーの元素組成およびモノアイソトピック質量が決定されています10。HRMS を用いてサンプルを詳細に分析し、モノアイソトピックピークの精密質量分析で質量誤差が 0.7 ~ 7 ppm でした。次に BioAccord を、対応する質量確認測定に適用しました。実際には、ここではモノアイソトピックピークの手動解析に重点を置きました。ただし、UNIFI/waters_connect ソフトウェアを使用することで、MaxEnt1 または BayesSpray のデコンボリューションアルゴリズムのいずれかにより、荷電ゼロのマススペクトルを得ることができます。このデータ解析アプローチも、質量分析および質量確認と同じく魅力的な手段です11,12。PCR プローブの分子の詳細は公開されていません。そこで、ウォーターズ社内の Tof HRMS 装置を用いた精密質量測定と、BioAccord LC-MS システムを用いた質量確認に基づいて、最も可能性の高い分子式を決定しました。すべての質量測定値を表 1 に示します。
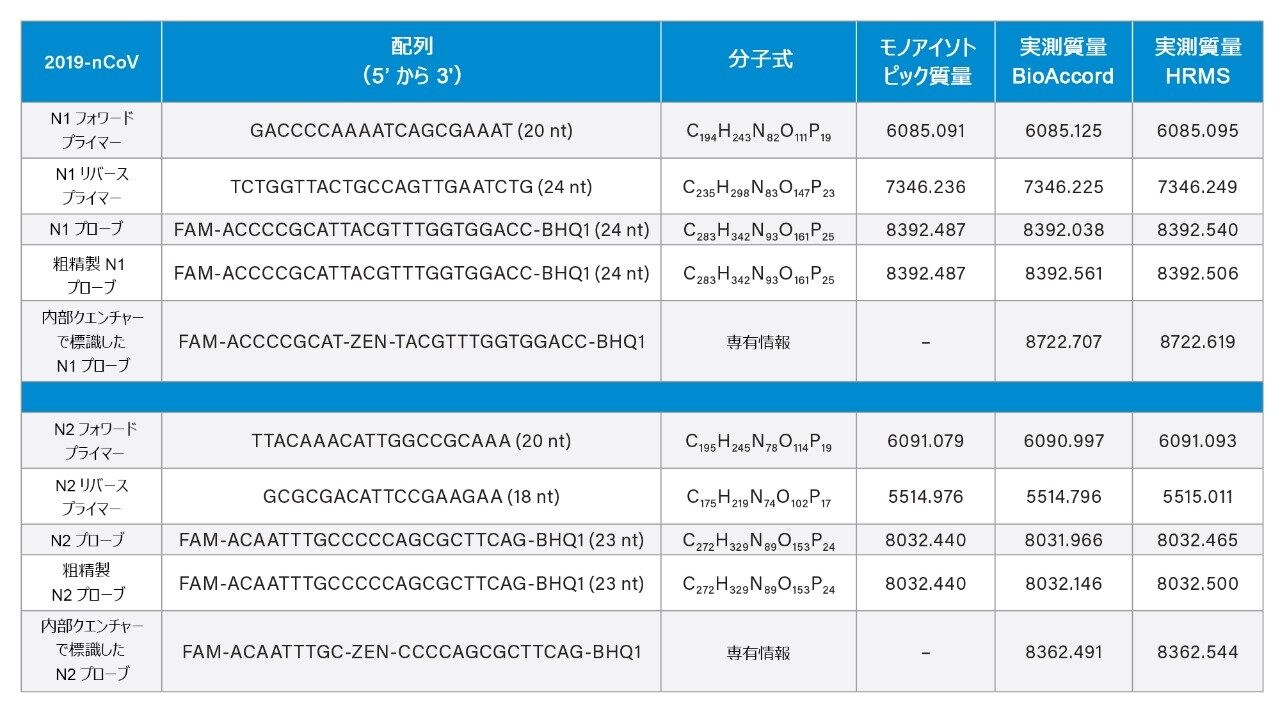
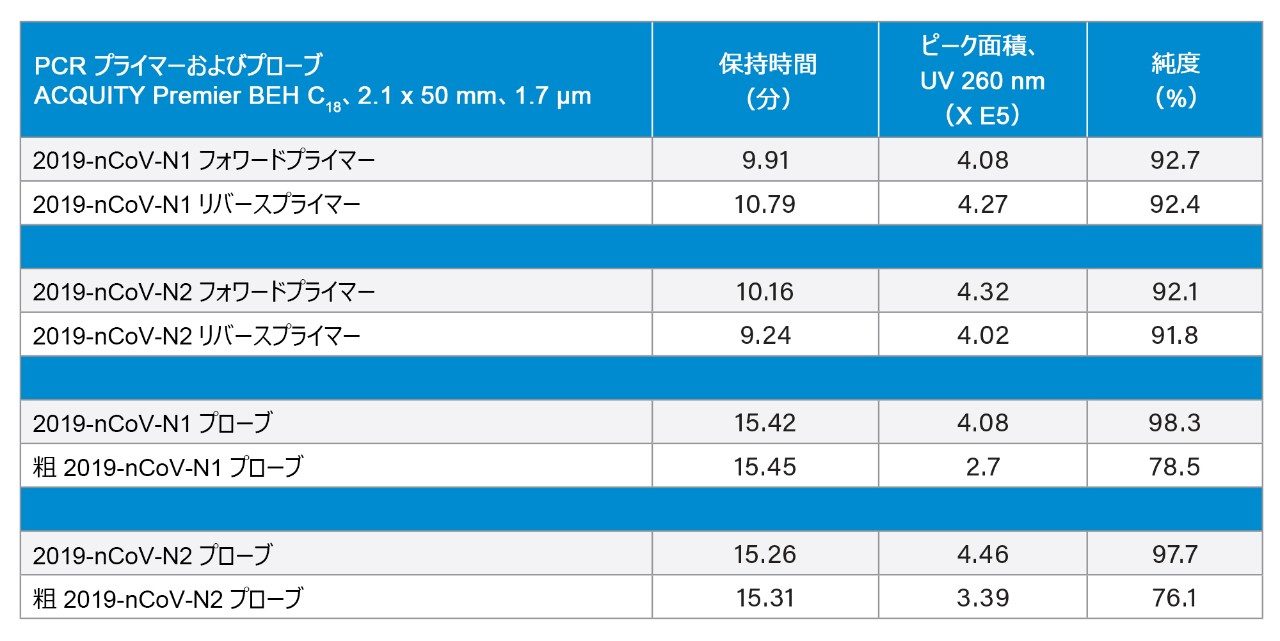
精密質量測定に加え、UV および MS と組み合わせて IP-RPLC を使用し、試薬の純度および化学合成後に脱塩しただけの未精製の粗製品を評価しました。そのため、収集した UV クロマトグラムを使用してクロマトグラフィーピークを波形解析しました。図 4 に波形解析したクロマトグラムを示します。ここで粗 N2 プローブの純度割合を計算するのは興味深いことでした。UNIFI を使用して自動ピーク解析を行いました。保持時間、クロマトグラフィーピーク面積および純度が表 2 に報告されています。CDC アッセイ用に適格性評価したプライマーの純度は 92%、対応する粗製品の純度は約 90% であることがわかりました。CDC アッセイ用に適格性評価したプローブは高純度(約 98%)でしたが、対応する粗製品(脱塩のみ)の純度はわずか約 76% でした。
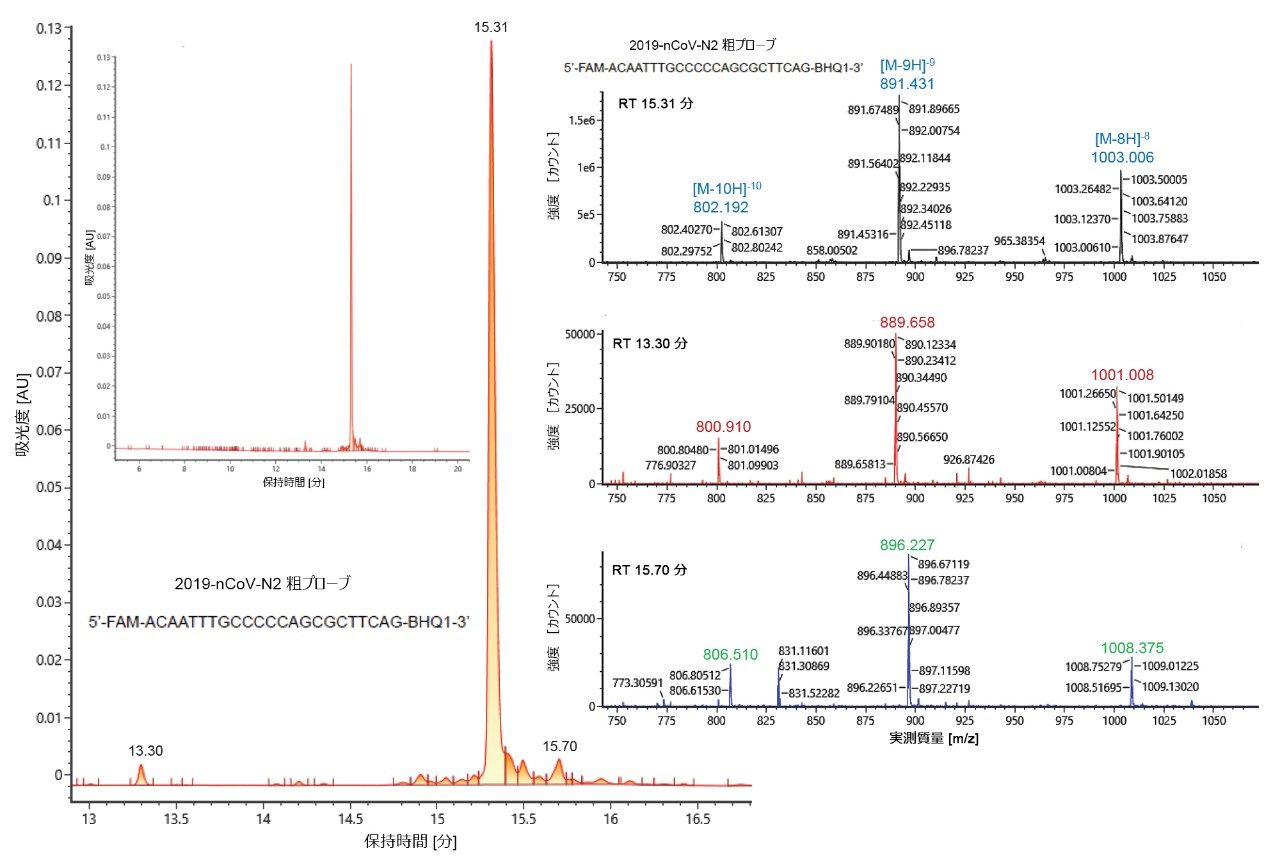
粗プローブ中の不純物を更に詳細に調査しました。主要クロマトグラフィーピークが 15.31 分に見られたのに加えて、粗 N2 プローブの UV トレースには 13.30 分と 15.70 分に小さなピークが見られました(図 4)。これら 3 つのピークについて得られた合計マススペクトル[15.31 分(上)、13.30 分(中央)、15.70 分(下)]を図 4 の右のパネルに示します。13.30 分のピークは、主要クロマトグラフィーピークと比較して質量が -16 Da シフトした分子種に対応しています。これは、プローブ構造中の酸素原子の損失を示しています。その後 15.70 分に溶出するピークは、正の質量シフト 42.9 Da に対応する推定不純物です。まとめると、表 2 に報告し図 4 に例示したデータから、ACQUITY UPLC TUV 検出器と BioAccord RDa 検出器を用いた直列検出により、PCR 試薬の純度評価ができることが分かりました。
結論
このアプリケーションノートでは、最適化した IP-RPLC およびコンプライアンス対応のベンチトップ型 Tof 装置を用いた、SARS-CoV-2 CDC アッセイで使用される PCR 検査試薬の品質評価について説明しました。この感度の高いアプローチにより、わずか 10 pmol のヌクレオカプシドプライマー N1 と N2(フォワード配列とリバース配列の両方)および N1 プローブと N2 プローブから、純度について多くの情報が収集できました。すべての PCR 試薬についてシャープで対称的なピークが得られました。260 nm での UV 検出の後、ネガティブイオンモードでマススペクトルを取り込むことで、多重荷電分子種(最大 [M-10H]10-)が容易に観察されました。ナトリウムおよびカリウムの付加イオンは、ベースピークシグナルの 10% を下回っており、RT-qPCR キット試薬の分子量を明確に決定できました。UV 検出および MS 検出を組み合わせて、試薬の純度および未精製の粗製品を評価しました。これらの純度測定の例では、UNIFI を使用して UV クロマトグラムの自動ピーク解析を行いました。更に、このアプリケーションノートで説明した分析アプローチにより、シングルクエンチャー N2 プローブの未精製の粗合成品の調査で実証されたように、不純物のより詳しい調査が可能になりました。まとめると、高分解能で高感度の LC-UV-MS を組み合わせた手法により、掘り下げた情報が得られ、RT-qPCR 試薬を認定する上でのメリットがもたらされました。
参考文献
- WHO Coronavirus (COVID-19) Dashboard [Internet].[cited 2021 Sept 02] https://covid19.who.int/.
- Akkiz, H., Implications of the Novel Mutations in the SARS-CoV-2 Genome for Transmission, Disease Severity, and the Vaccine Development.Front.Med.2021, 8, 10.
- Vogels, C. B. F.; Brito, A. F.; Wyllie, A. L.; Fauver, J. R.; Ott, I. M.; Kalinich, C. C.; Petrone, M. E.; Casanovas-Massana, A.; Catherine Muenker, M.; Moore, A. J.; Klein, J.; Lu, P.; Lu-Culligan, A.; Jiang, X.; Kim, D. J.; Kudo, E.; Mao, T.; Moriyama, M.; Oh, J. E.; Park, A.; Silva, J.; Song, E.; Takahashi, T.; Taura, M.; Tokuyama, M.; Venkataraman, A.; Weizman, O.-E.; Wong, P.; Yang, Y.; Cheemarla, N. R.; White, E. B.; Lapidus, S.; Earnest, R.; Geng, B.; Vijayakumar, P.; Odio, C.; Fournier, J.; Bermejo, S.; Farhadian, S.; Dela Cruz, C. S.; Iwasaki, A.; Ko, A. I.; Landry, M. L.; Foxman, E. F.; Grubaugh, N. D., Analytical Sensitivity and Efficiency Comparisons of Sars-Cov-2 RT–qPCR Primer–Probe Sets.Nature Microbiology 2020, 5 (10), 1299–1305.
- Pearson, J. D.; Trcka, D.; Lu, S. Y.; Hyduk, S. J.; Jen, M.; Aynaud, M. M.; Hernandez, J. J.; Peidis, P.; Barrios-Rodiles, M.; Chan, K.; Woodgett, J.; Mazzulli, T.; Attisano, L.; Pelletier, L.; Cybulsky, M. I.; Wrana, J. L.; Bremner, R., Comparison of SARS-CoV-2 Indirect and Direct RT-qPCR Detection Methods.Virol.J. 2021, 18 (1), 12.
- Filges, S.; Mouhanna, P.; Ståhlberg, A., Digital Quantification of Chemical Oligonucleotide Synthesis Errors.Clinical Chemistry 2021.
- Guo, L.; Worth, A. J.; Mesaros, C.; Snyder, N. W.; Glickson, J. D.; Blair, I. A., Diisopropylethylamine/Hexafluoroisopropanol-Mediated Ion-Pairing Ultra-High-Performance Liquid Chromatography/Mass Spectrometry for Phosphate and Carboxylate Metabolite Analysis: Utility for Studying Cellular Metabolism.Rapid Commun Mass Spectrom 2016, 30 (16), 1835–1845.
- Birdsall, R. E.; Gilar, M.; Shion, H.; Yu, Y. Q.; Chen, W., Reduction of Metal Adducts in Oligonucleotide Mass Spectra in Ion-Pair Reversed-Phase Chromatography/Mass Spectrometry Analysis.Rapid Commun Mass Spectrom 2016, 30 (14), 1667–1679.
- Fountain, K.; Gilar, M.; Budman, Y.; Gebler, J., Purification of Dye-Labeled Oligonucleotides by Ion-Pair Reversed-Phase High-Performance Liquid Chromatography.Journal of Chromatography.B, Analytical Technologies in the Biomedical and Life Sciences 2003, 783, 61–72.
- Jiang, T.; Yu, N.; Kim, J.; Murgo, J.-R.; Kissai, M.; Ravichandran, K.; Miracco, E. J.; Presnyak, V.; Hua, S., Oligonucleotide Sequence Mapping of Large Therapeutic mRNAs via Parallel Ribonuclease Digestions and LC-MS/MS.Anal.Chem.2019, 91 (13), 8500–8506.
- Ortiz, D.; Gasilova, N.; Sepulveda, F.; Patiny, L.; Dyson, P. J.; Menin, L., Aom2S: A New Web-Based Application for DNA/RNA Tandem Mass Spectrometry Data Interpretation.Rapid Communications in Mass Spectrometry 2020, 34 (23), e8927.
720007429JA、2021 年 12 月