COVID-19 を理解する:SARS-CoV-2 LC-MS キット(RUO)を用いた SARS-CoV-2 デルタ変異株の明確な同定
研究目的のみに使用してください。診断用には使用できません。
要約
2021 年 7 月現在、SARS-CoV-2 デルタ(B.1.617.2)変異株が、世界中で急速に優勢なウイルスになりつつあります。SARS-CoV-2 LC-MS キット(RUO)は、野生型 SARS-CoV-2 NCAP タンパク質に含まれるシグネチャーペプチドを検出するために開発されました。GISAID データベースを使用して進行中の変異株を追跡することで、デルタ変異株から生じたトリプシン消化ペプチドを、SARS-CoV-2 LC-MS キット(RUO)の SISCAPA 抗体を用いて捕捉・定量できることが確認されました。さらに、デルタ変異株は、クロマトグラフィー分離とマルチプルリアクションモニタリング(MRM)を用いて、他の懸念される変異株に含まれる野生型ペプチドから選択的に同定・区別できます。
アプリケーションのメリット
- SARS-CoV-2 デルタ変異株を SARS-CoV-2 LC-MS キット(RUO)を使用して同定できます。
- SARS-CoV-2 デルタ変異株を、SARS-CoV-2 LC-MS キット(RUO)を使用して、他の懸念される変異株と区別できます。
はじめに
急速に変化しつつある新型コロナウイルス感染症パンデミックの中で、SARS-CoV-2 ウイルスの変化に適応し、対応することが、かつてないほど重要になっています。2021 年 7 月現在、SARS-CoV-2 デルタ(B.1.617.2)変異株が、世界中で急速に優勢なウイルスになりつつあります。SARS-CoV-2 の mRNA および翻訳されたアミノ酸配列が、ウイルスの有無を特定するための多くの分析技術のターゲットとして使用されています。デルタ変異株に含まれている変異は、これらの標的部位に影響を及ぼすため、SARS-CoV-2 の同定へのこれらのプラットホームの適合性に影響を及ぼします。
SARS-CoV-2 LC-MS キット(RUO)は、臨床研究の試験における SARS-CoV-2 ヌクレオカプシド(NCAP)ペプチド、AYNVTQAFGR(AYN)、ADETQALPQR(ADE)、および NPANNAAIVLQLPQGTTLPK(NPA)の直接分析を補助するために発売されたものです。安定同位体標準および抗ペプチド抗体による捕捉(SISCAPA)および ACQUITY UPLC I-Class と Xevo TQ-XS 質量分析計を用いて、ウイルス輸送培地中の 3 amol/μL の各ペプチドを検出できることが実証されています1。
キットに含まれるこれらのターゲットペプチドに対する変異の影響が、GISAID データベースを使用してモニターされています2。 以前に懸念された変異株であるアルファ、ベータ、ガンマのデータを抽出して検討したところ、ターゲットのペプチド配列に影響を与える変異は確認されませんでした。一方、デルタ変異株を検証したところ、ADE ペプチドが影響を受けており、その結果、ペプチド(AYETQALPQK)の N 末端近傍のアミノ酸置換(D377Y)およびそれほど高頻度に存在しない C 末端(R385K)の置換があることが確認されました。
このアプリケーションブリーフでは、SARS-CoV-2 LC-MS キット(RUO)の SISCAPA 抗体を使用してデルタ変異株の捕捉も可能であり、クロマトグラフィー分離とマルチプルリアクションモニタリング(MRM)を用いて、他の懸念される変異株に含まれる野生型ペプチドから選択的に同定・区別できることを示しています。
結果および考察
このアプリケーションノートに詳述した試験は、SARS-CoV-2 LC-MS キット(RUO)の取扱説明書(IFU)に従って実施しました。
キットのワークフローでは、3 種のペプチド(SARS-CoV-2 NCAP タンパク質に由来する AYNVTQAFGR(AYN)、NPANNAAIVLQLPQGTTLPK(NPA)、ADETQALPQR(ADE))の変性、消化、濃縮、および UPLC-MS/MS 分析について説明しています。
ペプチド AYETQALPQR、ADETQALPQK および AYETQALPQK を合成し(Lifetein、NJ、USA)、ADETQALPQR(ADE)、AYNVTQAFGR(AYN)、および NPANNAAIVLQLPQGTTLPK(NPA)を含む溶液に添加しました。これらのペプチドをウイルス輸送培地に添加し、SARS-CoV-2 LC-MS キット(RUO)を用いて繰り返しで抽出し、1 回の実験で分析しました。
Skyline(MacCoss Lab Software、WA、USA)を用いて変異ペプチドの MRM トランジションを特定し、ACQUITY UPLC I-Class と Xevo TQ-XS 質量分析計を使用して分析しました。
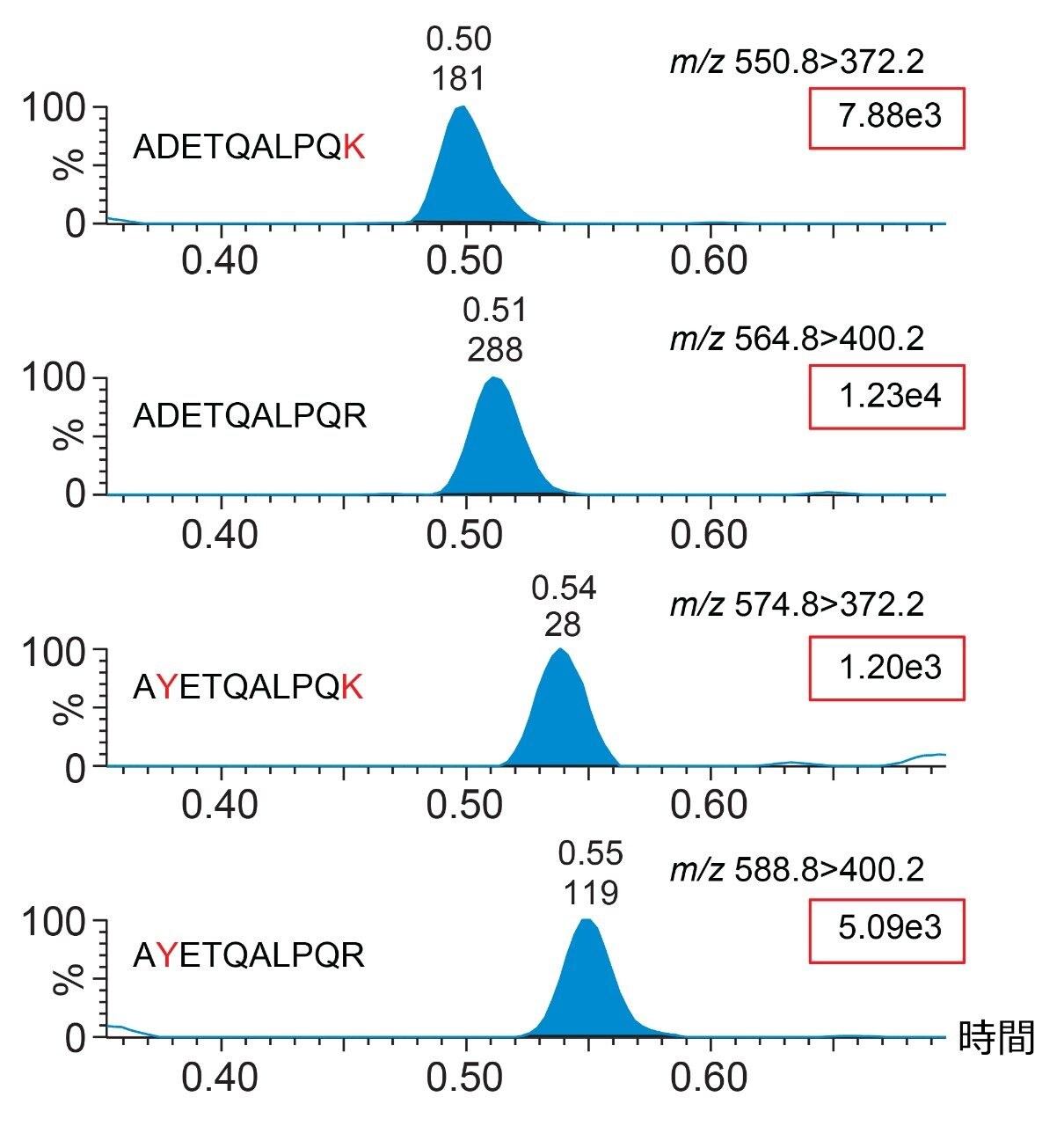
変異型ペプチドは、特定の MRM トランジションを使用して、ペプチド間の保持時間のわずかなずれで同定できます(図 1A)。これらの新しいペプチドターゲットを同定するのに、この分析法に大きな変更を加える必要はありません。
SISCAPA を用いると、すべての変異型ペプチドが、野生型 ADE のモノクローナル抗体によって1 回の実験で捕捉されます。ただし、二重変異(D377Y/R385K)がある場合、他のペプチドと比較して分析法の感度が大幅に低下することが確認されています(図 1B)が、スパイクしたすべてのペプチドについて、5 回の繰り返しにわたって依然として検出が再現可能です(≤8.9% RSD)。D377Y/R385K 変異型ペプチドは、濃度が 10 amol/μL を超えていれば同定できます(図 2)。
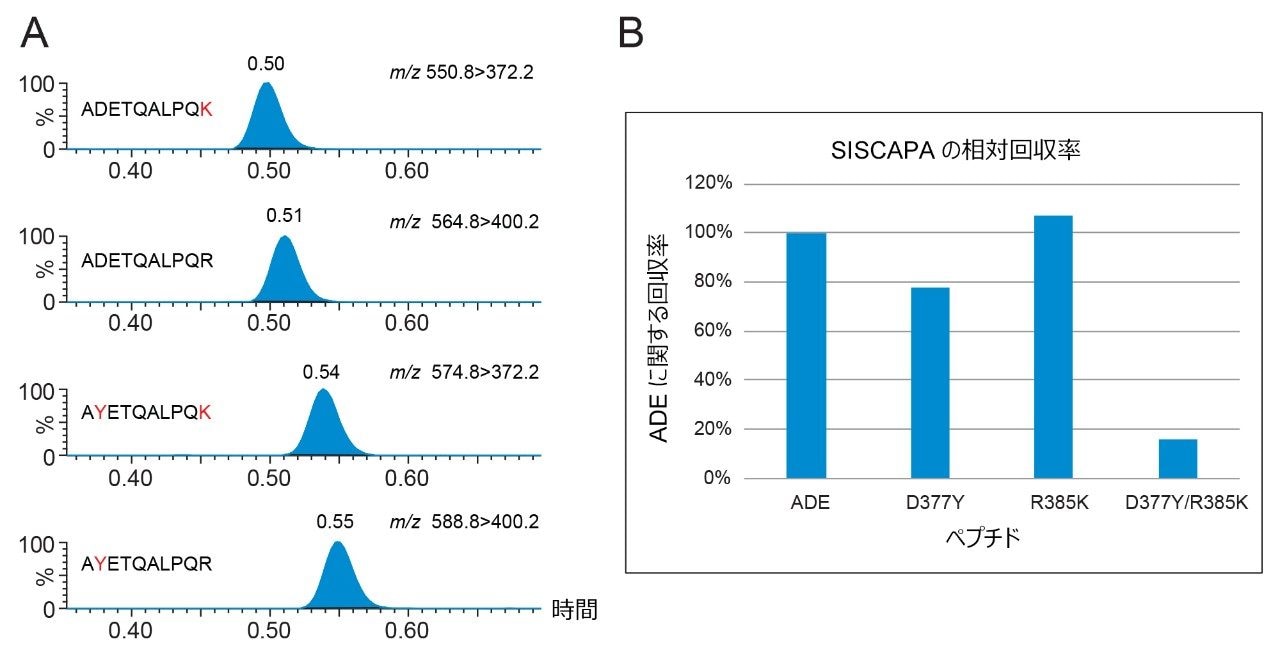
図 1B. ADETQALPQR、AYETQALPQR(D377Y)、ADETQALPQK(R385K)、および AYETQALPQK(D377Y/R385K)の SISCAPA での相対回収率(各ペプチドにつき n=5)
結論
Waters SARS-CoV-2 LC-MS キット(RUO)を使用することで、SARS-CoV-2 デルタ(B.1.617.2)変異株に関連する変異型ペプチドを捕捉し、同定することに成功しました。
D377Y 置換は、GISAID データベースを 2020 年 3 月から 2021 年 7 月まで分析した際に最も多く見られた変異であり(55%)、R385K および D377Y/R385K 二重置換の頻度は 0.045% 未満と推定されています。デルタ変異株ペプチドとその他の懸念される変異株(アルファ、ベータ、ガンマ)に関連するペプチドが、クロマトグラフィー分離と MRM トランジションの同時モニタリングによって、1 回の実験に基づいて区別できます。したがって、SARS-CoV-2 LC-MS キット(RUO)は、主要な SARS-CoV-2 デルタ変異株の NCAP ペプチドの臨床研究のための LC-MS/MS 分析に適していることが確認できました。
参考文献
- Foley D, Wardle R, Ferries S, Pattison R, Warren J, Calton L. Advancing Research with the SARS-CoV-2 LC-MS Kit (RUO).Waters Application Note 720007266, May 2021.
- GISAID database.https://www.gisaid.org/hcov19-variants/.
720007353JA、2021 年 8 月