本アプリケーションノートでは、イオンモビリティー機能を搭載した高分解能質量分析を用いて、いくつかの血清サンプルを再分析しました。Mass-MetaSite および WebMetabase ソフトウェアパッケージを使用してデータを解析し、ペプチド分解代謝物を自動的に検出し、可視化しました。HDMSE で取得したデータ(イオンモビリティーを用いた HRMS データ非依存的測定)の正確度と選択性と組み合わせることで、分解代謝物の正確な同定と追跡が可能になります。
ペプチドは、新薬の候補化合物として多くの魅力的な特性を備えていますが、透過性や安定性などの薬理学的特性に欠けることが多く、開発は困難です。このような制限を克服するために、ペプチドは、複雑な構造および非天然型の修飾を導入することによって、ますます複雑なエンジニアリングが施され、強化されています。
これらのペプチドの分解代謝物の同定は、医薬品開発プロセスにおける重要な一環であり、特に特殊なヒト分解代謝物の場合に、クリアランスおよび代謝物の結末を理解するために重要になります。信頼性の高い同定を行うことは、分析上および計算上も困難です。簡単な配列であっても、何百もの潜在的な代謝物をもたらす多くの加水分解可能な結合が含まれています。in vitro または in vivo の研究では、様々なアミノ酸やエンジニアリングされたペプチドセグメントが、酸化、脱アミド化、結合、脱結合などの更なる形質転換経路に直面することもあります。エレクトロスプレーベースの LC-MS 分析を使用して取得したペプチドの多価の性質により、多くの m/z 値が得られ、すべての分解代謝物が確実に同定されるように、それらすべてをスクリーニングする必要があります。これらの要因すべてにより、ペプチドの分解代謝物の同定は複雑なものになっています。
ソマトスタチンは天然の成長ホルモン抑制ペプチドホルモンであり、数多くの研究が行われています。Riera 他は、ソマトスタチンについて、およびヒト血清中の安定性に対する配列修飾の影響について、詳細な説明を発表してます3。 本アプリケーションノートでは、イオンモビリティー機能を搭載した高分解能質量分析を用いて、これらの血清サンプルの一部を再分析します。
Mass-MetaSite および WebMetabase(Molecular Discovery Ltd.)ソフトウェアパッケージを使用してデータを解析し、ペプチド分解代謝物を自動的に検出し、可視化しました。HDMSE で取得したデータ(イオンモビリティーを用いた HRMS データ非依存的測定)の正確度と選択性を組み合わせることで、分解代謝物の正確な同定と追跡が可能になります4。
試験結果は、英国にある集中型サーバー上で WebMetabase を用いて分析しました。このサーバーでは、英国および米国にある多数のグローバルユーザーおよびラボが、データを同時にアップロード、解析、確認することができ、データセットの単一リポジトリーとして使用できます。さらに、WebMetabase で HELM 表記を有効にする機能(Pistoia Alliance)は、複雑なペプチドの分解代謝物の可視化およびフラグメントの同定に便利であることが証明されました。
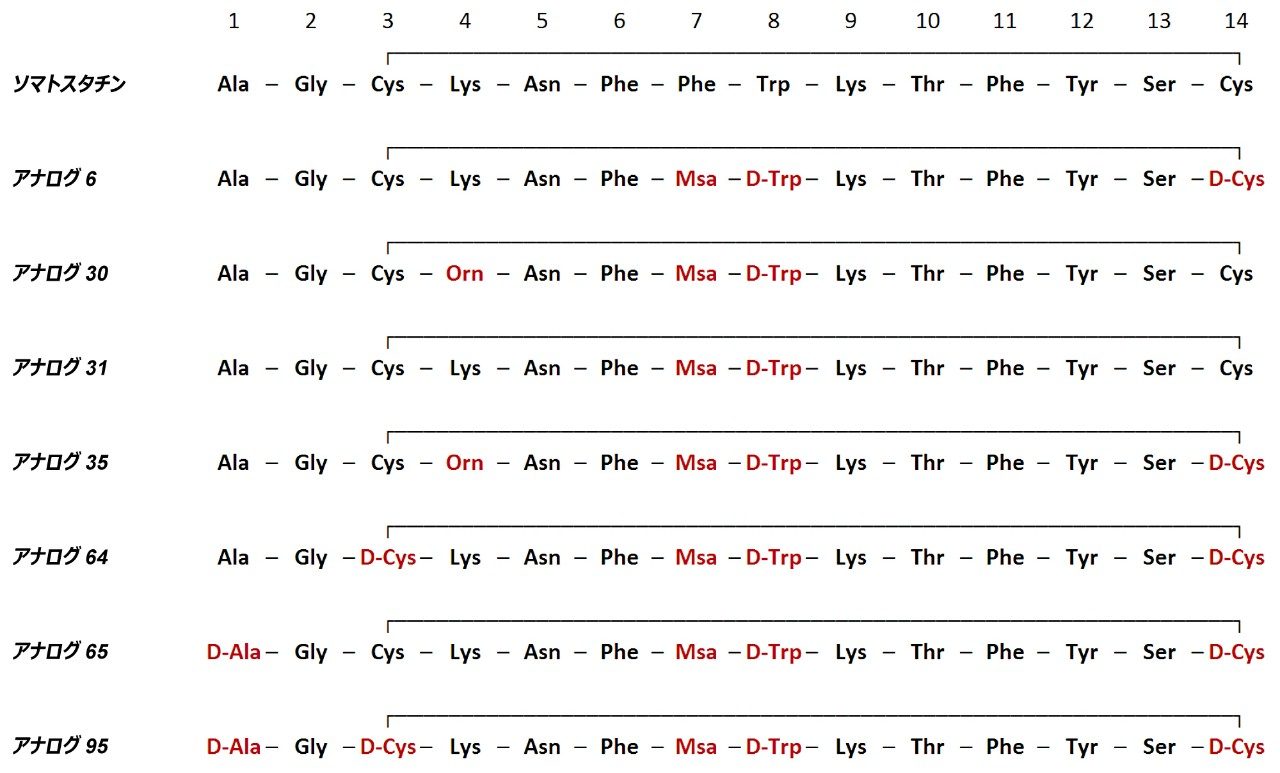
ソマトスタチンホルモンの 14-アミノ酸アナログ 8 種を検討しました(図 1)。ヒト血清で化合物を 11 の時間(0 分、5 分、10 分、30 分、1 時間、2 時間、4 時間、8 時間、24 時間、30 時間、48 時間)でインキュベートし、安定性と主要な代謝物の形成について評価しました。サンプルは、スペイン、バルセロナ科学技術研究所・生物医学研究室の Antoni Riera 教授の研究室からご提供いただきました。
|
システム: |
ACQUITY UPLC I-Class |
|
バイアル: |
ポリプロピレンバイアル、300 µL [製品番号:186002628] |
|
カラム: |
ACQUITY CSH C18、1.7 µm、2.1 mm × 100 mm [製品番号:186005297] |
|
カラム温度: |
45 ℃ |
|
サンプル温度: |
8℃ |
|
注入量: |
1 µL |
|
流量: |
0.4 mL/分 |
|
移動相 A: |
0.1% ギ酸水溶液 |
|
移動相 B: |
0.1% ギ酸含有アセトニトリル |
|
グラジエント: |
7 分間で 2%~30%、8 分間で 100% まで、10 分まで保持 |
|
システム: |
Vion IMS QTof 質量分析計 |
|
イオン化モード: |
ESI+ イオンモード |
|
測定モード: |
HDMSE(CCS を使用) |
|
取り込み範囲: |
m/z 50~2000 |
|
キャピラリー電圧: |
1.0 kV |
|
コリジョンエネルギー: |
低エネルギー 6 eV、高エネルギー 15〜55 eV まで上昇 |
|
スキャン時間: |
0.2 秒 |
API(アプリケーションプログラミングインターフェース)を搭載した UNIFI 1.9.4
Mass-MetaSite 3.4.2、WebMetabase 4.0(Molecular Discovery Ltd.)
一般データ、TEC(2 つのエネルギーの衝突)モードが有効
代謝物の生成、モニターされる反応:アミド加水分解、酸化的脱アミド、ジスルフィド還元
質量、MS ピーク、パターンフィルタリング、許容範囲(%):30
質量、Met ID、代謝物の世代:2
質量、Met ID、基質結合破壊限界:1
質量、Met ID、破壊代謝物:True、1
TEC、質量分析計:Waters QTof 質量分析計
TEC、アルゴリズムしきい値:クロマトグラム/MS/MSMS フィルタリング:0.95/0.95/0.95
TEC、シグナルしきい値:40
外部 LC-MS ファイルコンバーター:バージョン 2.1.9 UNIFI msvc64
HDMSE データは、自動 CCS レポート機能を完全に統合した UNIFI を使用して、Vion IMS QTof 質量分析計から収集されました。データは Mass-MetaSite を使用して解析し、MetaSite Batch Processor で WebMetabase にアップロードし、可視化しました1。
調査対象となったすべてのアナログは、Phe[7] が Msa に、Trp[8] が D-Trp に置換されています。Ala[1]、Cys[3]、Cys[14] の様々な順列からそれらの D-アミノ酸同等物への置換、および Lys[4] からオルニチンへの置換の安定性を評価しました。すべてのアナログに対して、WebMetabase を使用して、基質の消失をモニターし、主要代謝物形成のモジュレーション(特に -Ala および -AlaGly)を評価し、開環を含む他の主要な代謝経路のモニタリングを行いました。以前に報告されたように、天然ソマトスタチンの代謝は、主は分子の尾部からの Ala(-71 Da)および -AlaGly(-128 Da)の喪失であると認められています2,3。
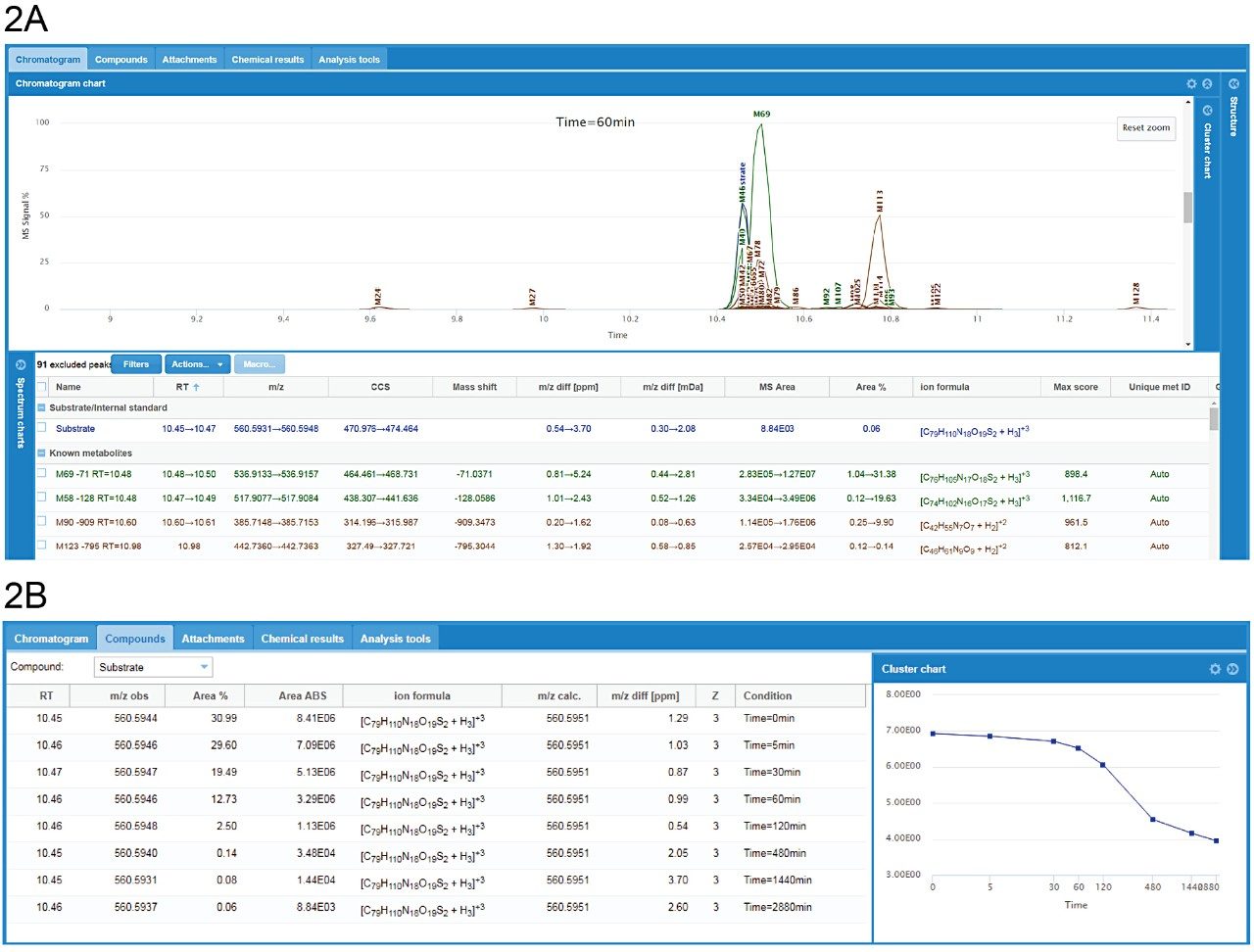
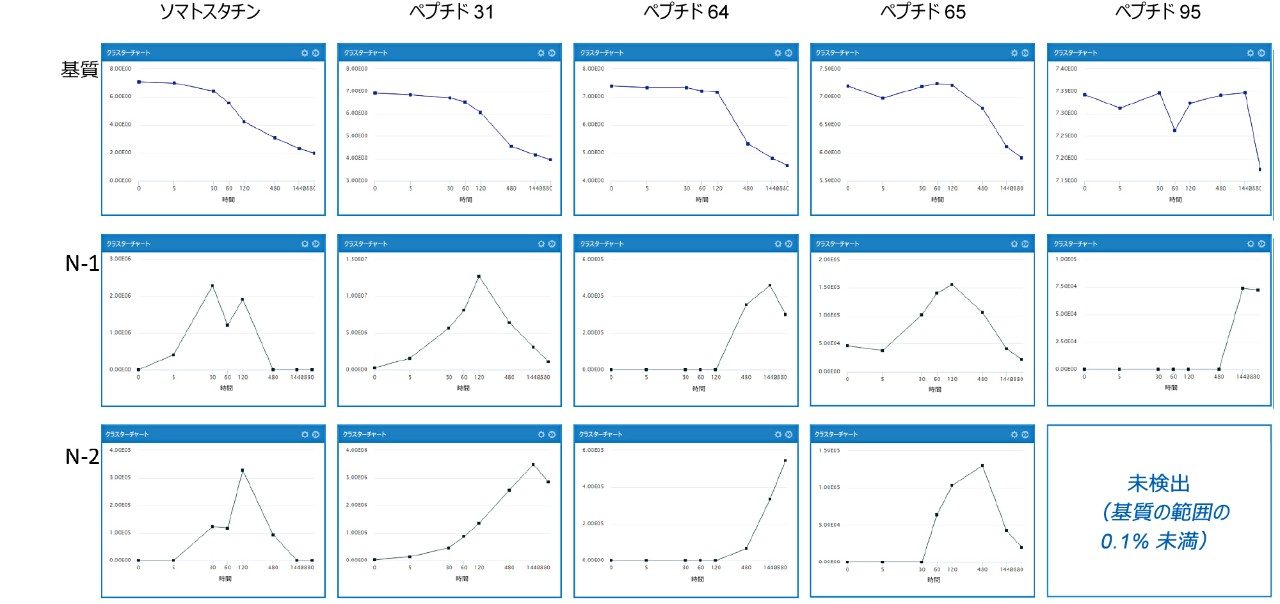
WebMetabase での代表例として、ペプチド 31 は、48 時間のインキュベーション後に大半が消えました。クロマトグラムの概要(図 2A)と化合物の概要(図 2B)を切り替えることにより、ユーザーは合計クロマトグラムの重ね書き、RT 値、m/z 値、親基質との質量差、MS 面積/レスポンス、およびサンプルセット全体または全成分の個々の化合物の基質測定範囲の比率を確認できます。CCS 値も、Mass-MetaSite と WebMetabase の両方に直接統合されます。イオンモビリティーにより、スペクトルのクリーンアップや無関係なイオンの除去が可能になり、m/z だけでなく、CCS も同一であるピークを追跡できることが保証されます。これは、同重体のピークがシフトしたり、近接して共溶出する場合に特に有益です。
WebMetabase は、検出されたすべての代謝物の視覚表示と、同定されたすべての代謝物を評価するスコアリングアルゴリズムを提供します。主要代謝物とその構造を図 3A に示しています。代謝物予測のスコアは各構造式の左上に示されており(すべて 800 以上)、に示されており、スペクトル同定の一致および不一致の数もそれぞれ示されています。図 3B は、これらの化合物のレスポンスの経時的変化を示しています。-71(-Ala)および -128(-AlaGly)代謝物の形成は 10 分後に見られ、30~60 分で最大レベルに達します。後続の開環生成物 Asn-Phe-Msa-D-Trp-Lys-Thr および Phe-Msa-D-Trp-Lys-Thr(それぞれ NF-Msa-dWKT および F-MSa-dWKT と略す)の形成は、60 分後に見られています。
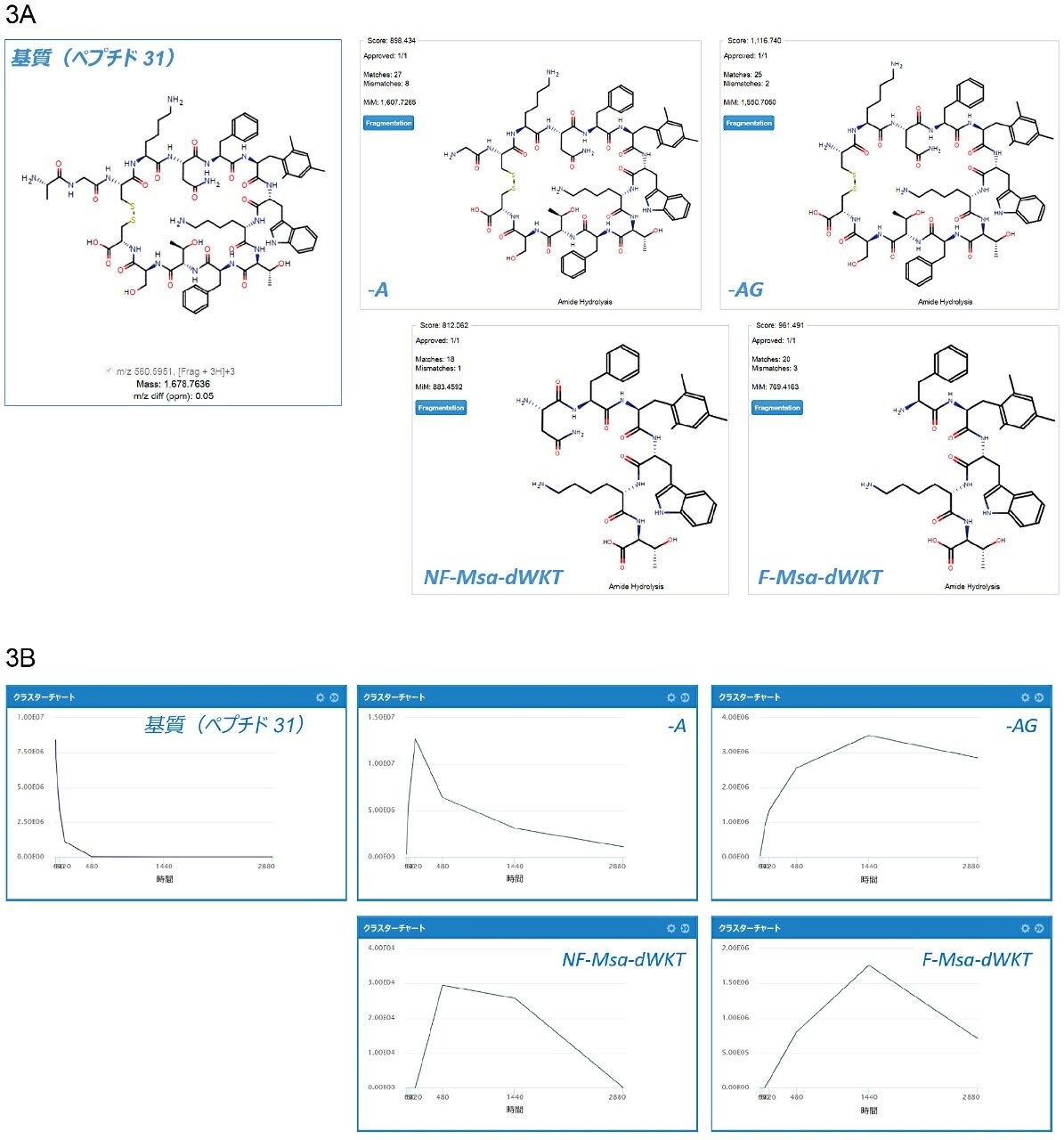
図 4 では、ソマトスタチンおよび 4 つの Msa/D-Trp 修飾ペプチド(31、64、65、95)のレスポンスをプロットし、基質および N-1、N-2 の分解代謝物の喪失をすべて詳細に比較しました。代謝の大部分は、N-1 および N-2 のアミノ酸喪失(-Ala および -AlaGly)に起因すると考えられます。末端アラニンまたは架橋システイン分子が修飾されている場合、クリアランスに大きな差異が見られました。基質と主要な N-1 および N-2 代謝物の時間対レスポンスプロファイルは、薬物の消失と、主要代謝物の出現(または不在)との関係を示しています(図 4)。
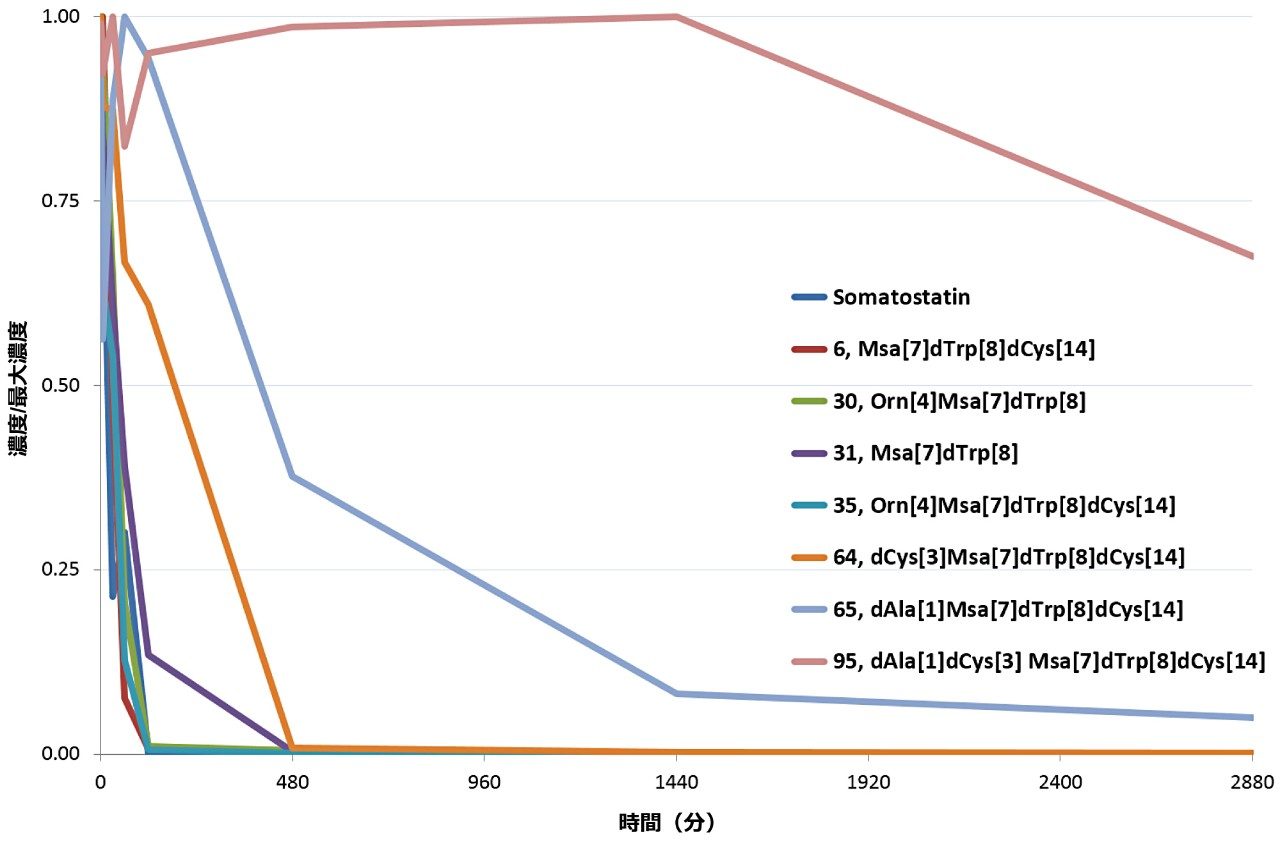
すべてのペプチドアナログを WebMetabase で評価した後、安定性をプロットし(Excel)、2 日間の相対比較を示しました(図 5)。WebMetabase によって解析された分解代謝物の情報からは、Ala[1]、Cys[3]、Cys[14] の D-アミノ酸置換により、それぞれの安定性が向上するだけでなく、相乗効果がもたらされるようであることがわかります。ペプチド 31 からペプチド 6 のように、一部の修飾(特に Cys[14] から D-Cys[14])自体には顕著な影響はありませんでしたが、D-Cys[3] との組み合わせでは、ペプチド 6 からペプチド 64、またはペプチド 65 からペプチド 95 のように、非常に有効でした。Lys[4] から Orn[4] および Phe[7]Trp[8] から Msa[7]D-Trp[8] への置換は、全体的な安定性に関して、天然ソマトスタチンと比較してわずかな違いを示しました(詳細データは表示されていません)。全体的に、ペプチド 95 は非常に安定しています。これは、他のアナログと比較して、代謝の遅延および減少によってさらに証明されます。
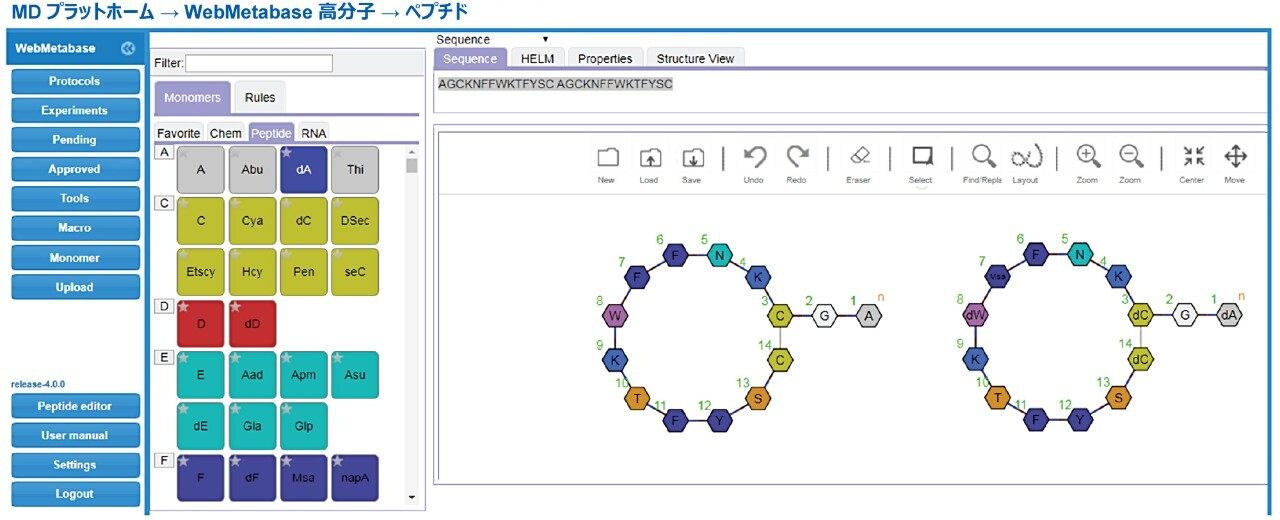
HELM 表記により、複雑な高分子構造をモノマー形式で表示できます(Pistoia Alliance、https://www.pistoiaalliance.org/projects/current-projects/helm/)。この機能により、モノマーの化学構造情報が保持され、高分子構造のシーケンスベースの構造表記と複雑な分子描画(例:.mol または .SDF 形式)の間のギャップが埋められます。この例を図 6 に示しています。ここでは、ソマトスタチンとペプチド 95 の分子構造を HELM 表記を用いて示しています。
WebMetabase を使用して、ソマトスタチンアナログ 8 種すべてのデータを解析および可視化しました。分解は主に、ジスルフィド結合した環部分の外側の末端アミノ酸で発生しました。主要な代謝経路および比率をモニターするために、親の安定性を観察し、主要な開裂の出現をモニターしました。L-システインと末端 L-アラニン残基の置換により、安定性が最大限に向上しました。D-Cys[3] と D-Cys[14] の結合などによる相乗効果が認められ、安定性が大幅に向上しました。WebMetabase で Vion データを解析することで、このような修飾とソマトスタチンの分解を低減する効果の効率的な同定が可能になり、特定の開裂が置換によって増強または阻害されたことが明確になりました。
イオンモビリティーの使用および生成された CCS 値により、各ペプチドに追加の追跡特性が提供され、モビリティーによりフィルタリングされた XIC が向上し、特定の置換または開裂において、CCS の分子サイズに与える効果を更に掘り下げることが可能になり、データ品質が向上しました。Mass-MetaSite および WebMetabase を使用して解析されたイオンモビリティーデータにより、複雑なペプチド分解データを簡単な方法で効率的にレビューすることができました。
Molecular Discovery などのサードパーティベンダーの機能を統合することで、IMS 拡張ワークフロー、HELM 統合、クラウドベースの視覚化パッケージなどの機能に迅速にアクセスすることができました。便利な HELM 統合により、生物学者は(.mol または .SDF 形式よりも)身近な視点を容易に得るようになり、難しい構造を持つ分子およびハイブリッドが開発されるにつれて、今後も有用性は高いと考えられます。データをクラウドで保存、解析、共有する機能により、世界各国にいる多数のプロジェクトユーザーが(英国にある)集中型サーバーにアクセスできるようになっています。施設間でデータを日常的に共有することで、観測結果について議論し、複数の研究者の知識を活用できるようになりました。WebMetabase はブラウザーベースであるため、複数の PC や様々な場所から簡単にアクセス可能で、クライアント PC 用の高価なハードウェアを揃える必要性や、ソフトウェアのローカルコピーをインストールするための要件が少なくなります。
720006586JA、2019 年 8 月