
高分解能質量分析(HRMS)の手法は、高分子化合物のリストを扱う(ノンターゲット取り込み)のに適しており、より多くの分子情報(フラグメント経路、同位体情報、付加イオンなど)が得られます。
食品の安全性分析に最近導入されたイオンモビリティーと HRMS の組み合わせにより、サイズ、形状、電荷に基づいたイオンの更なる分離が可能になります。Vion IMS QTof システムでは、イオンがイオン源に導入された後にイオンモビリティー分離(IMS)が行われます。その結果、化合物のコリジョン断面積(CCS、単位は Å2)値が得られ、モビリティーセル中を通過するときのイオンの平均回転値を反映するユニークなパラメーターになります。イオンモビリティーデータのユニークな点として、スペクトルのクリーンアップが挙げられます。これは、未知化合物のスペクトルを解析して、既知化合物を確認するのに役立ちます。
このアプリケーションノートでは、親イオンとフラグメントイオンの両方の精密質量データ、同位体分布、イオンモビリティー分離、質の高い UPLC分離を組み合わせて使用し、大規模なスクリーニングライブラリーを用いて、インゲン、イチゴ、ハラペーニョ、ミニパプリカの抽出物中のターゲット農薬化合物を同定する方法を実証します。また、このアプローチを利用して、野菜抽出物中に存在する新しい殺虫剤を同定しました。
「農薬スクリーニング」という一般用語には多くの場合、複雑なサンプルと大規模なターゲットのリストが関わってきます。前者は、適用化合物の範囲を最大化するための一般化された抽出メソッドと、食品の生化学におけるバリエーションの結果です。後者は、何千もの化合物が使用登録されている結果です。タンデム四重極 MS/MS 法の使用に加え、高分解能質量分析(HRMS)手法は、高分子化合物のリストを扱う(ノンターゲット取り込み)のに適しており、より多くの分子情報(フラグメント経路、同位体情報、付加イオンなど)が得られます。食品の安全性分析に最近導入されたイオンモビリティーと HRMS の組み合わせにより、サイズ、形状、電荷に基づいたイオンの更なる分離が可能になります1。 Waters Vion IMS QTof システムでは、イオンがイオン源に導入された後にイオンモビリティー分離(IMS)が行われます。その結果、化合物のコリジョン断面積(CCS、単位は Å2)値が得られ、イオンがモビリティーセル中を通過するときのイオンの平均回転値を反映するユニークなパラメーターになります2。イオンモビリティーデータのユニークな点として、スペクトルのクリーンアップが挙げられます3。これは、未知化合物のスペクトルを解析して、既知化合物を確認するのに役立ちます。
このアプリケーションノートでは、親イオンとフラグメントイオンの両方の精密質量データ、同位体分布、イオンモビリティー分離、質の高い UPLC 分離を組み合わせて使用し、大規模なスクリーニングライブラリーを用いて、インゲン、イチゴ、ハラペーニョ、ミニパプリカの抽出物中のターゲット農薬化合物を同定しました。データインディペンデント取得を使用して、ミニパプリカ抽出液中に、スクリーニングライブラリーに存在しない比較的新しい代替農薬を同定することができました。すべての結果をクロスチェックしたところ、正確であることがわかり、以前のタンデム四重極 MS/MS 分析で予想された化合物が同定されました。
最適なシステム性能を確保するため、サンプル分析の前に自動セットアップを行いました。ボタンを押すだけで、検出器、分解能、ロックスプレー、キャリブレーションがすべてセットアップされます。インゲン、イチゴ、ハラペーニョ、ミニパプリカのサンプル抽出液を、(3 回繰り返しで)1 µL および 5 µL 注入して分析しました。「UPLC 条件」セクションに記載しているように、2 種類のクロマトグラフィーメソッドを使用しました。サンプルの分析前と分析後に、繰り返し QC 注入を実行して、質量精度の安定性、CCS 値、予想フラグメントイオンの生成、レスポンス、保持時間(RT)を評価しました。イオンモビリティー分離によるフルスペクトル取り込みを使用し、高コリジョンエネルギー状態と低コリジョンエネルギー状態を交互に使用して(HDMSE)データを取り込みました。取り込み後、データは自動的にコンポーネント化され、化合物名、構造、保持時間、予想フラグメント質量、CCS 値(場合によっては +H イオンおよび +Na/+ 付加イオンについて)が含まれるスクリーニングライブラリーに照らして解析されました。ライブラリーの値は、メソッド A の LC 条件(メソッド条件で説明)および溶媒標準試料を使用して生成されています。CCS および RT の場合、十分に特性解析された値になるように、複数の入力の平均値を求めました。データの取り込み、解析、レビューは UNIFI ソフトウェア v.1.8 を使用して行いました。
サンプル抽出物は、共同研究者から提供され、改変された QuEChERS メソッドを使用して前処理しました。抽出物は 100% アセトニトリル中で保存し、注入する前に水で 1:1 希釈しました。
|
メソッド A: |
|
||
|---|---|---|---|
|
LC システム: |
ACQUITY UPLC I-Class |
||
|
カラム: |
ACQUITY UPLC BEH C18、1.7 µm、2.1 × 100 mm |
||
|
カラム温度: |
45 ℃ |
||
|
サンプル温度: |
4 ℃ |
||
|
流速: |
0.450 mL/分 |
||
|
注入量: |
1 µL および 5 µL |
||
|
移動相 A: |
10 mM 酢酸アンモニウム(pH 5.0)水溶液 |
||
|
移動相 B: |
10 mM 酢酸アンモニウム(pH 5.0)メタノール溶液 |
||
|
サイクルタイム: |
17 分 |
|
分 |
流速(mL/分) |
%A |
%B |
|---|---|---|---|
|
初期条件 |
0.45 |
98 |
2 |
|
0.25 |
0.45 |
98 |
2 |
|
12.25 |
0.45 |
1 |
99 |
|
13.00 |
0.45 |
1 |
99 |
|
13.01 |
0.45 |
98 |
2 |
|
17.00 |
0.45 |
98 |
2 |
|
メソッド B |
|
|---|---|
|
UPLC システム |
ACQUITY UPLC I-Class |
|
カラム: |
ACQUITY UPLC HSS T3 1.8 µm 、2.1 × 100 mm |
|
カラム温度: |
40 ℃ |
|
サンプル温度: |
4 ℃ |
|
流速: |
0.450 mL/分 |
|
注入量: |
1 µL および 5 µL |
|
移動相 A: |
0.1% ギ酸含有 4 mM ギ酸アンモニウム水溶液 |
|
移動相 B: |
0.1% ギ酸含有 4 mM ギ酸アンモニウムメタノール溶液 |
|
サイクルタイム: |
18.5 分 |
|
分 |
流速(mL/分) |
%A |
%B |
|---|---|---|---|
|
初期条件 |
0.4 |
95 |
5 |
|
0.50 |
0.4 |
95 |
5 |
|
3.00 |
0.4 |
57 |
43 |
|
13.50 |
0.4 |
19 |
81 |
|
14.50 |
0.4 |
1 |
99 |
|
16.50 |
0.4 |
1 |
99 |
|
16.51 |
0.4 |
95 |
5 |
|
18.50 |
0.4 |
95 |
5 |
|
MS システム: |
Vion IMS QTof |
||
|
イオン化モード: |
ESI+ |
||
|
コリジョンエネルギー(LE): |
3 eV |
||
|
コリジョンエネルギー(HE ランプ): |
20 ~ 55 eV |
||
|
スキャン時間: |
0.25 秒 |
||
|
取り込み範囲: |
m/z 50 ~ 1200 |
||
|
ドリフトガス: |
N2 |
||
|
IMS 波速: |
250 m/秒 |
||
|
IMS 波高(ランプ): |
20 ~ 50 V |
||
|
キャピラリー電圧: |
0.8 kV |
||
|
サンプルコーン電圧: |
20.0 V |
||
|
イオン源温度: |
120 ℃ |
||
|
イオン源オフセット: |
80 |
||
|
脱溶媒温度: |
550 ℃ |
||
|
コーンガス流量: |
50 L/時間 |
||
|
脱溶媒ガス流量: |
1,000 L/時間 |
||
|
ロックマス: |
ロイシンエンケファリン(m/z 556.2766) |
||
|
データ管理: |
UNIFI 科学情報システム |
非常に複雑なデータセットに対してライブラリースクリーニングアプローチを使用して、すべての抽出について、500 種類以上のターゲット化合物のリストに対して、フルスペクトルのサンプルデータを調査しました。すべてのサンプルのレビューに続いて、以前に実行した LC タンデム四重極 MS/MS 分析および同定に対してクロスチェックしたところ、予想された化合物がすべて見つかりました。これらの同定は、図 1 のミニパプリカの成分プロットに表示されています。338 ng/g ~ 10 ng/g 未満までの広い濃度範囲(「微量」レベルと記載)も、タンデム四重極 MS/MS 分析から得られたものです。
場合によっては、LC タンデム四重極 MS/MS データには報告されていませんが、ターゲット農薬の代謝物がライブラリーに存在し、同定されていたものもありました(図 1)。UNIFI ソフトウェアの[レビュー]部分内で、フィルターを使用して同定にたどり着きました。フィルターは、ユーザーが定義する柔軟な自動基準であり、解析済みデータに対して適用されます。フィルターのデータレビューへの完全な統合は、ソフトウェアの個々のワークフローのステップを通して得られます。これには、サンプルの種類(未知試料、QC、標準試料など)や前述の特定の視点での基準が含まれます。UNIFI フィルターとワークフローのさらに詳細な説明は、ウォーターズの 720005436en に記載されています。偽陽性を減らし、提案される同定に確信が持てるように、このデータセットに適用するフィルターには UNIFI スクリーニングライブラリーの情報を利用しました。具体的には、表示されているデータは、図 2 に一覧表示されている基準(つまり、質量誤差、CCS% の差(ライブラリー値から)、RT 誤差(メソッド A で得られた値から)、フラグメントイオンの存在)の範囲内でした。
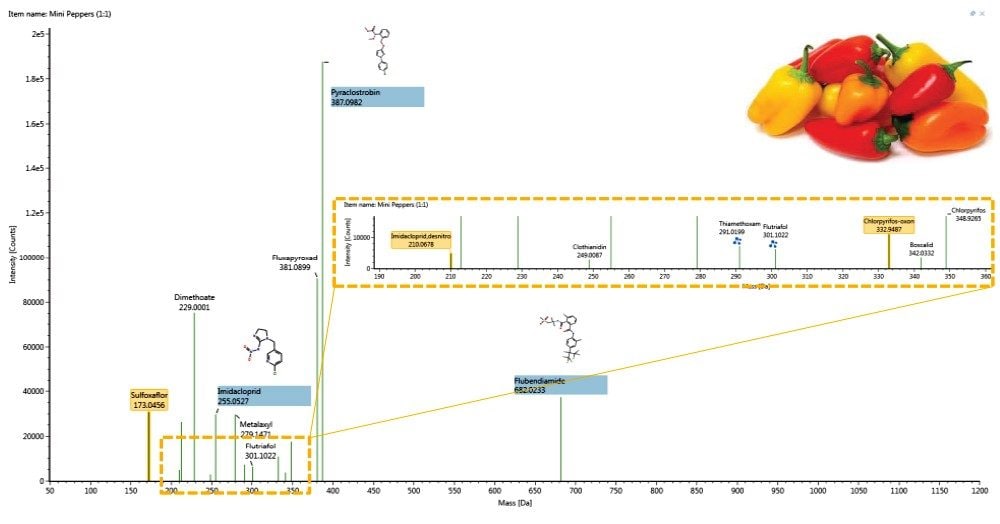
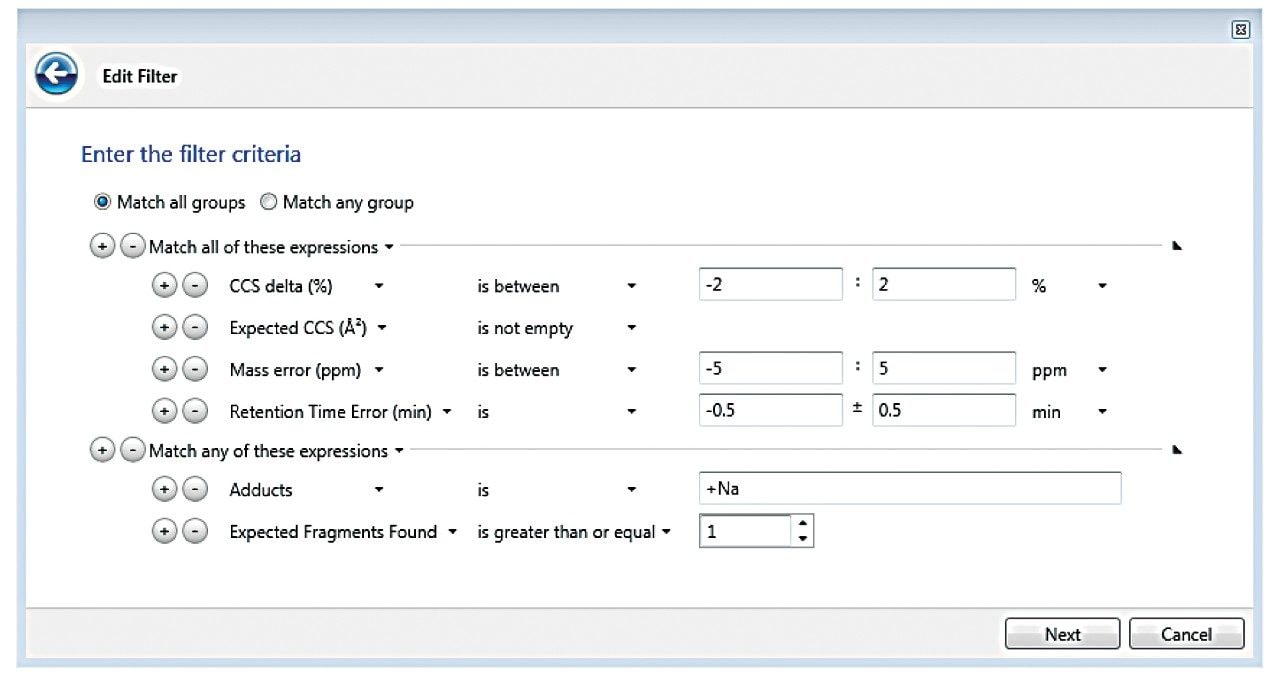
付加イオンの発生は、以前より回避されていますが、ESI 実験では避けられないことがあります。したがって、CCS などのフラグメントイオンに加えて、他の基準点を使用して同定を行うことが、偽陰性を避けるのに役立つことがあります。+Na 付加イオンの影響で予想フラグメントイオンがなくなる場合があることを考慮するため、予想フラグメントが存在しなければならない、あるいは +Na 付加イオンが観察された主なイオンであるなどの条件関数をフィルターに容易に導入することができます。図 2 ではこれを Match any of these expressions(これらの式のいずれかにマッチする)という演算子を使用して行っています。図 3 は、このフィルターがインゲンのサンプルの[レビュー]タブでどのように見えるかを示しています。フルベンジアミドでは +Na 付加イオンのみが表示され、フラグメントは表示されていません。ただし、他の重要な基準(CCS、質量精度、RT)は、指定された基準に対して正確でした。フィルターを作成すると、これが分析レビューのワークフローのステップに追加され、そのステップを選択するとデータに自動的に適用されます。ただ 1 つの同定、ミニパプリカで 10 ppb 以下のレベルであったフルトリアホールでは、+H 付加イオンが含まれており、予想フラグメントが含まれていませんでしたが、他の基準はすべて満たされていました。
![インゲン抽出物の[レビュー]タブ](/content/dam/waters/en/app-notes/2017/720006120/720006120en-f3.jpg.82.resize/img.jpg)
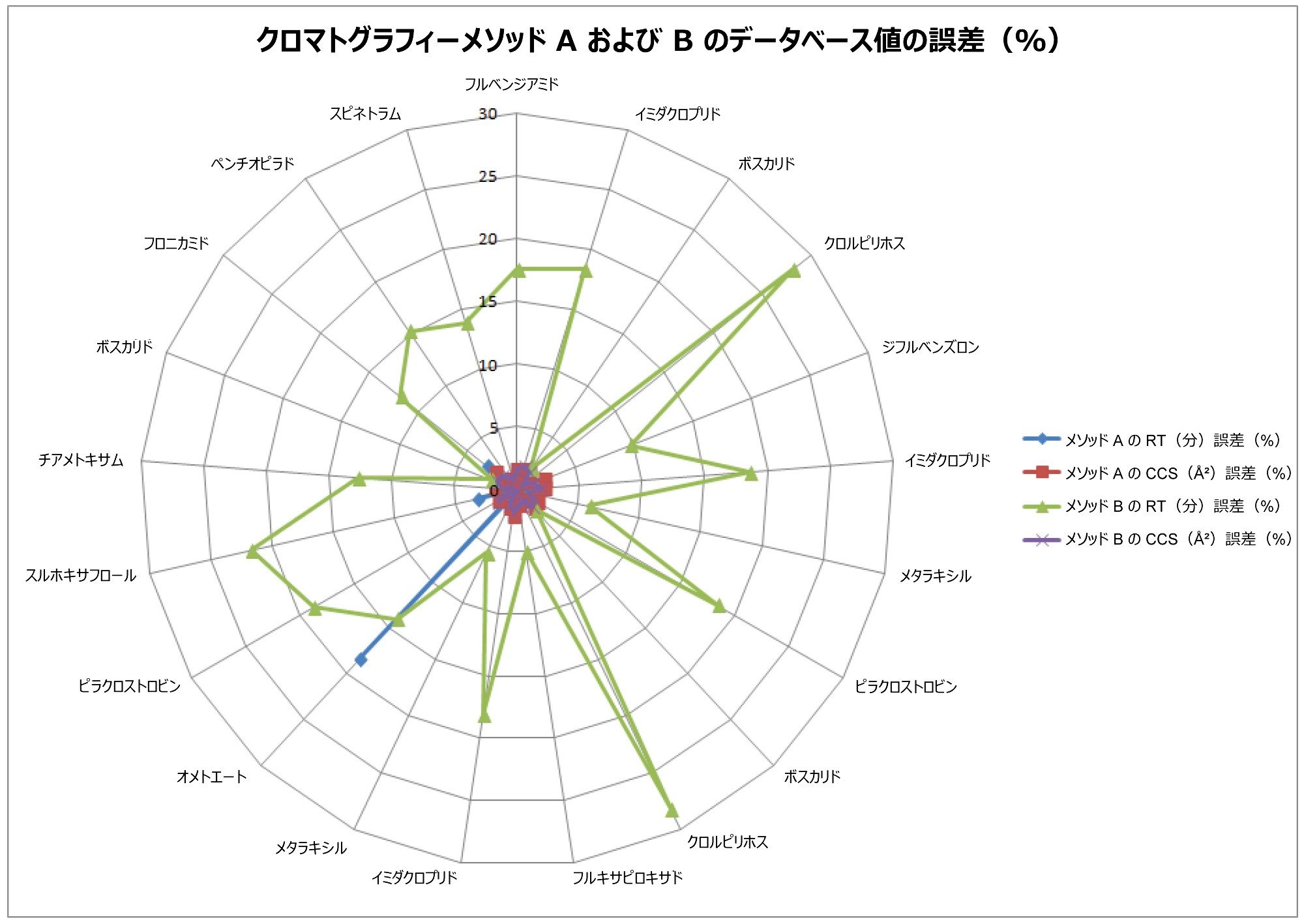
LC メソッド A による最初の分析に続いて、別のメソッド(LC メソッド B)が提案されました。このメソッドが提案されたのは、分析結果を改善するために考えられるメカニズムとして、移動相に異なる添加剤を使用し(酢酸アンモニウムではなくギ酸)、別のカラム(BEH C18 ではなく HSS T3)およびグラジエントを使用しているためです。結果として、さまざまな分析種の保持時間が、以前に使用したライブラリーのスクリーニングでの保持時間と大きく異なっていました。ただし、CCS 値は気相で取得されており、分子の固有の特質を示しているため、2 つの LC メソッドの間で CCS の測定値に大きな違いは見られませんでした。図 4 に、RT と CCS の両方についてのメソッド A と B の間のバリエーションをまとめており、上記の傾向が見られます。CCS を使用して同定ポイントの数を拡張することで、RT などの実験的に変動するパラメーターへの依存を少なくすることができました。同定を行う際に使用するフィルターの変更は、RT 要件を除くだけで瞬時に行えます。2 種類のクロマトグラフィーメソッドを使用して両方で正しい同定が行えた場合の結果を比較するため、新しいワークフローのステップを追加して、このフィルターがデータレビューアプローチに自動的に含まれるようにしました。
スクリーニングライブラリーやオンラインデータベースに含まれない可能性のある新規農薬の導入は、スクリーニングおよびモニタリングにおいて困難な課題をもたらします。ユーザーは、理論的に無限の数のターゲットイオンを検索することに加えて、共通ニュートラルロス、ハロゲンマッチ、質量欠損、バイナリー比較、多変量解析などのツールを使用して、対象の未知イオンについてデータを調査することができます。このようなツールの有用性は、一部のサンプル中に新規に製造された殺虫剤を同定した以下の例で実証されています。共同研究者によると、この殺虫剤はサンプル中には存在しますが、500 種類を超える農薬および代謝物が含まれている検索ライブラリーには含まれていませんでした。
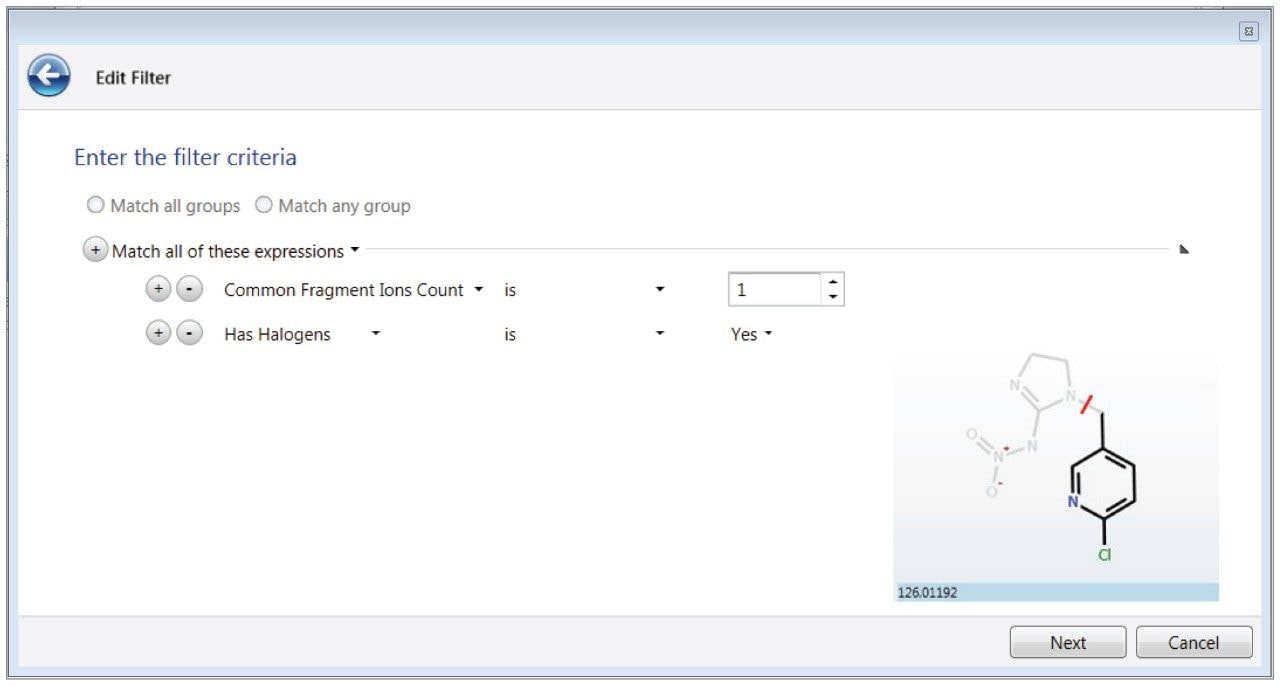
イミダクロプリドは、ミツバチの集団における蜂群崩壊症候群(CCD)と関連している可能性があるため、最近ヨーロッパと北米で監視下におかれているネオニコチノイド系殺虫剤です4。この相関関係を裏付けるデータにより、フランス5 とカナダではイミダクロプリドを含むネオニコチノイド系農薬の禁止が提案されています6。ほとんどの禁止物質の場合と同様に、ネオニコチノイド系農薬と同様の作用を持つ、より毒性の低い代替化合物の製造が求められています。この試験で分析したサンプル中の類似化合物をターゲットにする試みとして、既存のネオニコチノイドの構造比較を行ったところ、新しい化学配合中に存在する可能性のある共通フラグメントが判明しました。イミダクロプリド、アセタミプリド、チアクロプリド、ニテンピラムには、図 5 に示すように、分子の一部に 1 つの塩素置換基が導入された窒素含有環状構造が認められ、衝突誘起解離(CID)の間にこれからフラグメントが生成する可能性が高いと考えられます。
UNIFI フィルターを使用してすべてのデータを表示する基準を作成し、ハロゲンマッチツールとともにこの共通フラグメントを組み合わせました(図 5)。このフィルターは、完全なデータセット内の同定済み成分と未同定成分の両方において、低コリジョンエネルギースペクトルの塩素同位体分布パターンの組み合わせ、および高コリジョンエネルギースペクトルの質量 126.0119(C6H5ClN)のフラグメントを探します。このフィルターをミニピーマンのデータに適用すると、(ターゲットのリストに対して割り当てられていない)候補質量、およびイミダクロプリドとその代謝物であるイミダクロプリドデスニトロが表示されます(図 6)。後者は、以前のターゲット分析種のフィルターで同定されており、これらは単一の塩素および共通フラグメントのハロゲン同位体分布パターンを示すため、当然のことながら新しいフィルターでも同定されます。2 つの候補質量の文献調査により、質量 289.0554 は、最近製造されたブテノリド系殺虫剤のフルピラジフロンであることがわかりました。これは、ネオニコチノイドと同じく、ニコチン性アセチルコリン受容体をターゲットとするメカニズムで吸汁性昆虫を駆除する代替農薬です7,8。前述のように、共同研究者によってこの農薬はサンプルに含まれるとしてリストされていましたが、当社の元のスクリーニングリストには含まれていませんでした。したがって、本稿で説明したツールの使用により、フルピラジフロンを迅速かつ効果的に同定できました。1 回の右クリックで、この化合物を、含有残留物であることを示す修飾語をつけて UNIFI サイエンスライブラリーに追加できます(例:フルピラジフロン含有インゲン)。
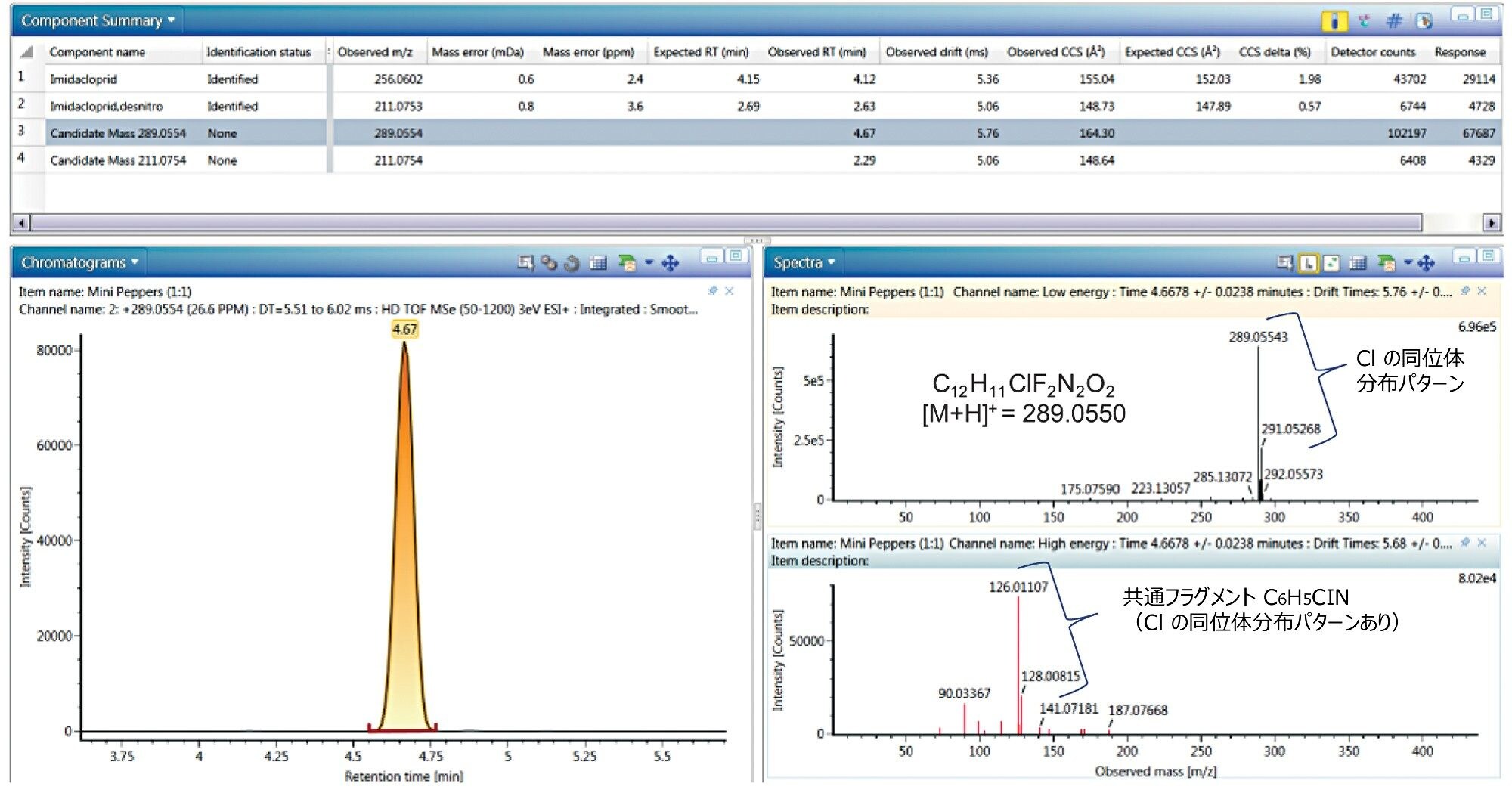
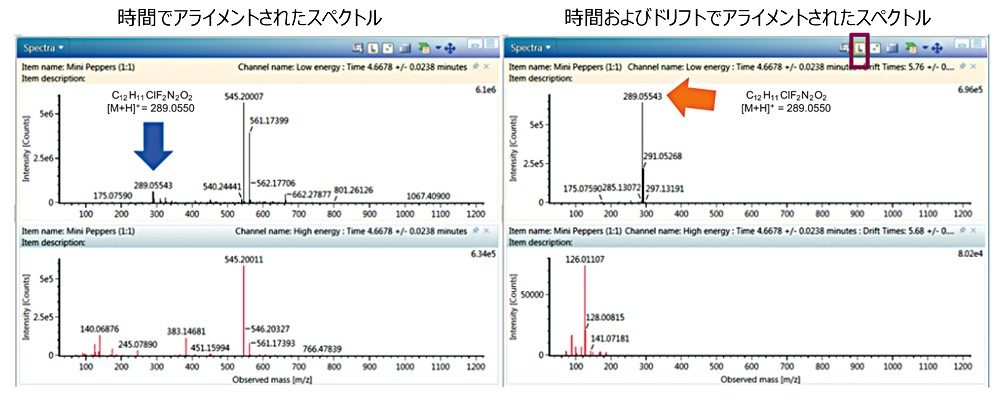
すでに強調した利点に加えて、図 7 のスペクトルの比較からわかるように、HDMSE データは同等の MS データよりきれいであることに注意してください。図 7 で強調表示されているボタンを切り替えると、保持時間とドリフト時間で揃えたスペクトルイオンが表示されます。図 7 に 2 つのスペクトルの比較も示します。IMS 対応スペクトルクリーンアップを使用すると、明らかによりきれいで解釈しやすいスペクトルになります。
HDMSE 法を使用して実施した農薬スクリーニング実験において、情報が豊富なデータセットが生成されました。プリカーサーイオンとフラグメントイオンの精密質量、RT、CCS などの同定基準に基づいて、データのフィルタリングやレビューを簡単に行うことができました。農薬スクリーニングに IMS を組み込むことで、CCS の測定が行えました。CCS により化合物の同定の信頼性が向上し、スクリーニング実験の追加パラメーターとして使用できます。UNIFI ソフトウェアには CCS の表示と比較計算がシームレスに組み込まれているため、レビューが簡単になります。フルスペクトル取り込みに対して処理されたこの UNIFI ライブラリースクリーニングアプローチは、取り込んだ包括的なデータを自動検索する手段を提供します。付加イオンの情報も、自動的にデータ解析に含められ、ユーザーに提示される化合物情報に組み込まれました。最後に、化学的特性に基づくインテリジェントフィルタリングを使用して、対象のノンターゲット化合物をデータセット内で検索できます。このアプローチを利用して、野菜抽出物中に存在する新しい殺虫剤を同定しました。
著者らは、サンプルをご提供いただいた米国ワシントン州 Bothell の FDA の Greg Mercer 氏および William Coole 氏に感謝致します。
720006120JA、2017 年 10 月