La cartographie peptidique et oligonucléotidique est un processus essentiel pour confirmer l’identité, la pureté et la séquence de biomolécules complexes comme les protéines ou l’ARN thérapeutiques. Conçus pour l’analyse LC/MS des produits biothérapeutiques modernes, les enzymes RapiZyme permettent des digestions rapides et reproductibles pour :
Ces outils permettent d’obtenir des procédures de caractérisation robustes et de haute résolution dans le domaine de la biothérapeutique moderne pour les utilisateurs de LC/MS . Amélioration du temps de traitement des échantillons et des opérations de laboratoire, et prise de décisions critiques rapide et en toute confiance avec les solutions Waters.

Comparaison de digestifs 1:5 entre un concurrent leader du secteur (panneau supérieur) et la trypsine RapiZyme (panneau inférieur), avec section agrandie de la fenêtre de temps de rétention de 14 à 40 min. Les flèches rouges mettent en évidence l’autolyse de la trypsine et les pics inconnus.
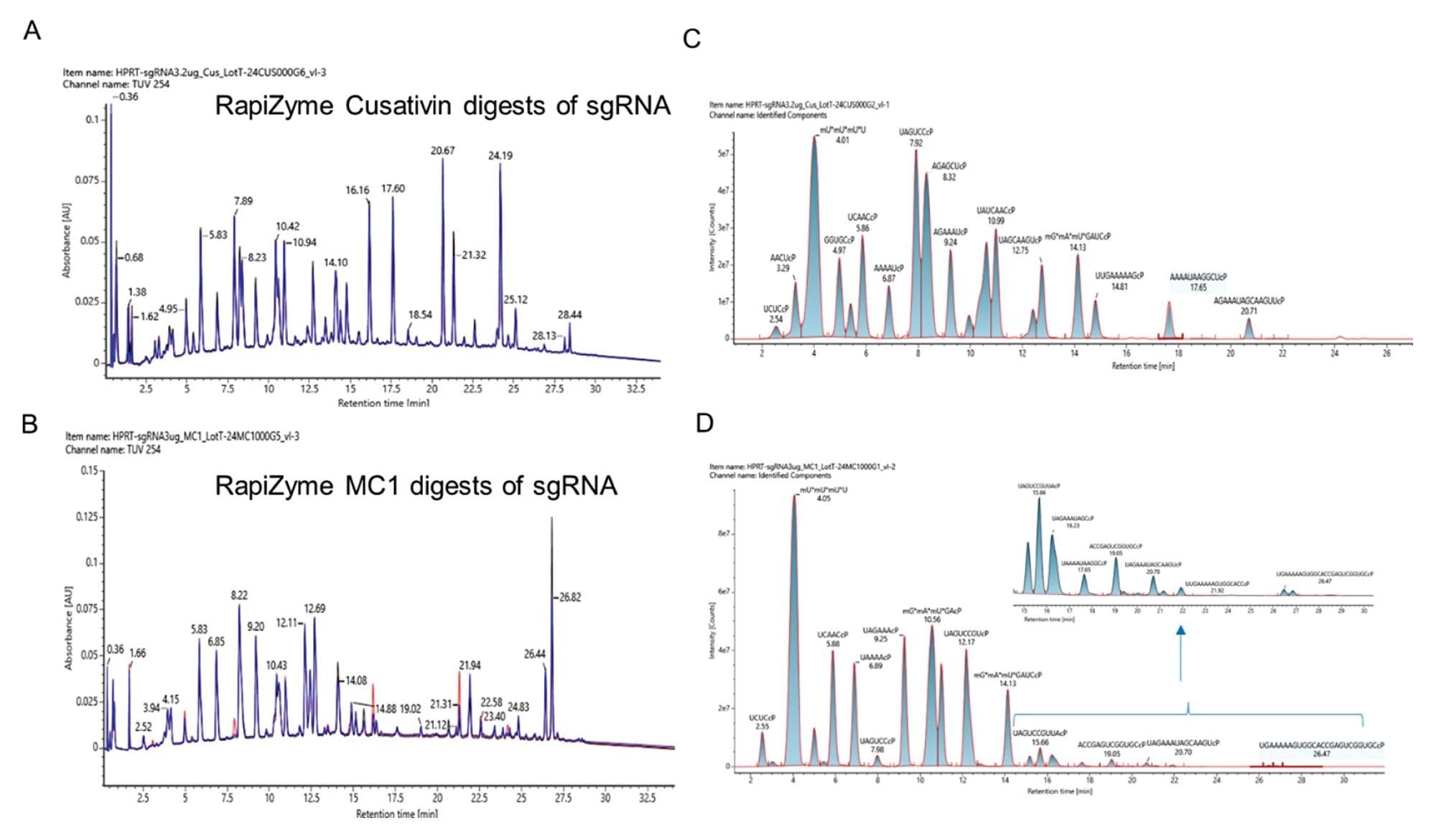
Analyse par LC-UV-MS de l’ARNsg HPRT digéré avec RapiZyme Cusativin et MC1 recombinantes. Trois lots indépendants d’enzymes recombinantes de RapiZyme Cusativin (A) et de RapiZyme MC1 (B) ont été utilisés pour digérer de l’ARNsg HPRT. Les trois chromatogrammes UV (tracés noirs, bleus et rouges) sont superposés l’un au-dessus de l’autre pour indiquer la reproductibilité du comportement de digestion de trois préparations. (C) Superposition du chromatogramme d’ion extrait des produits de digestion RapiZyme Cusativin identifiés, indiquant leur abondance dans le tracé TUV correspondant. (D) Superposition du chromatogramme d’ion extrait des produits de digestion RapiZyme MC1 identifiés, indiquant leur abondance dans le tracé TUV correspondant. Le profil des produit de digestion à faible abondance des produits de digestion RapiZyme MC1 est illustré sur la figure en encart.
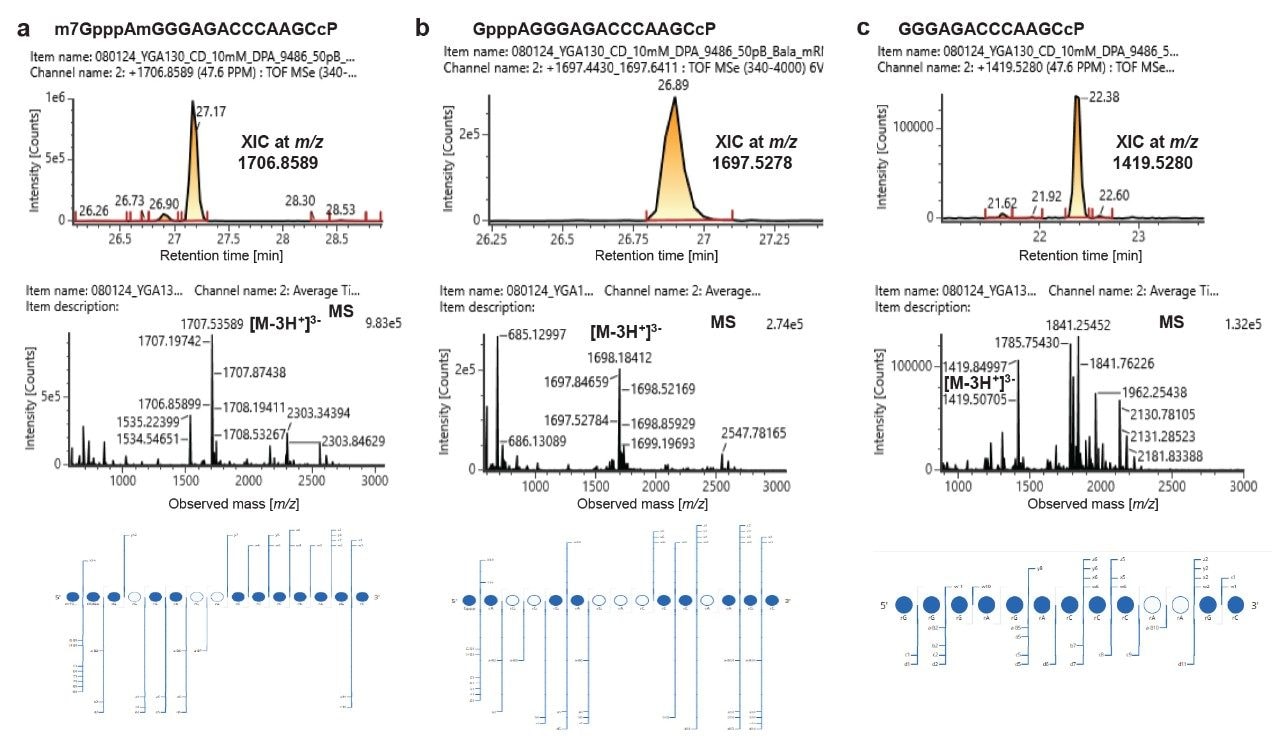
Caractérisation des différentes formes d’oligonucléotides coiffés en 5’ dans le digest par l’ARNase MC1 de l’ARNm 1 (XIC pour le panneau supérieur, le spectre de masse pour le panneau central, carte à points des ions fragments MSE pour le panneau inférieur). Détection de l’oligonucléotide coiffé (a), de la forme non méthylée de la version coiffée (b) et de la version non coiffée (c).
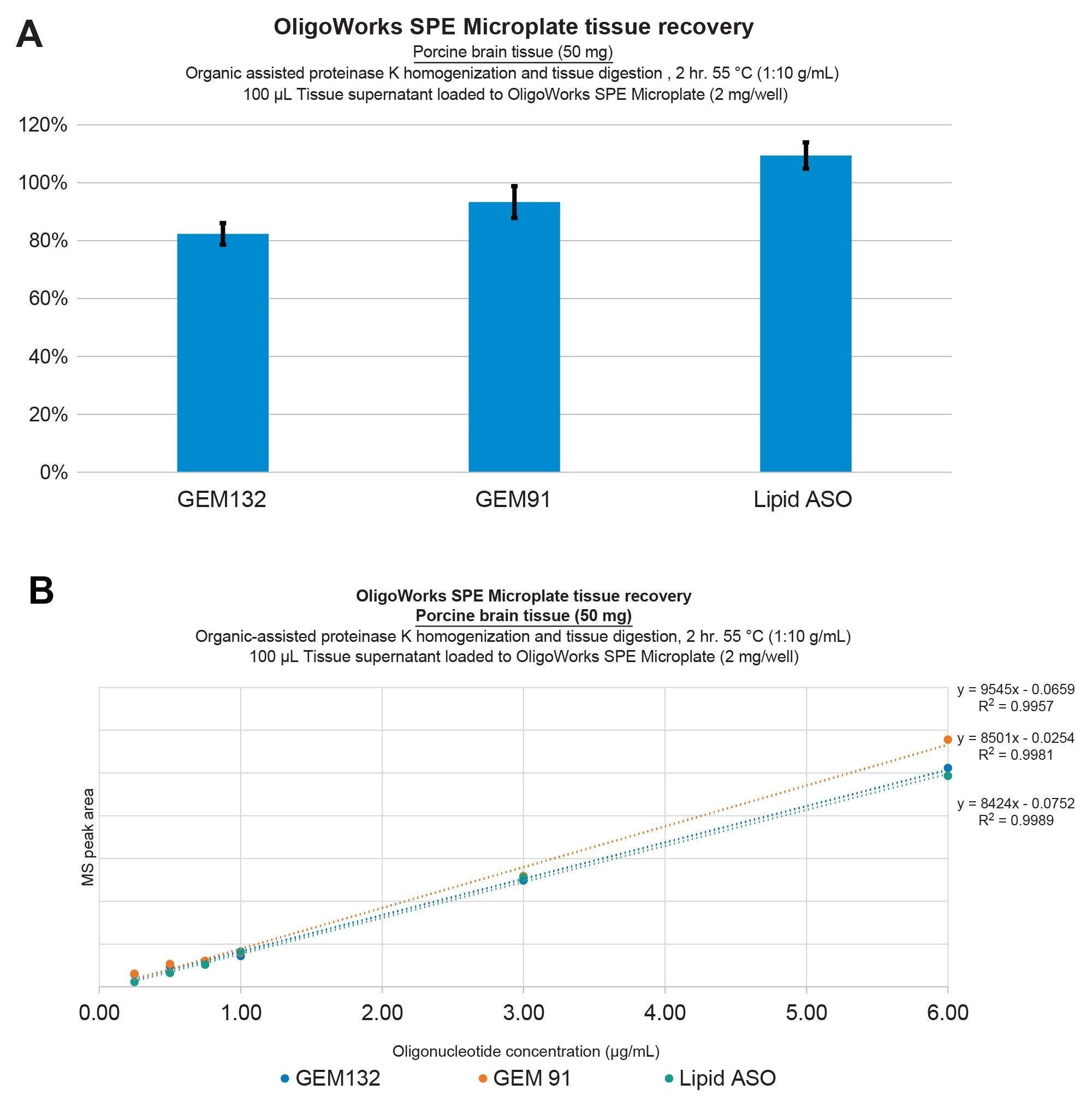
Démonstration des performances d’extraction des oligonucléotides pour GEM91, GEM132 et pour un ASO lipidique conjugué à partir de tissus cérébraux porcins. Les récupérations ont été déterminées comme étant ≥ 80 % avec une excellente répétabilité (écarts-types ≤ 10 %) (A). La linéarité de l’extraction a été déterminée comme étant ≥ 0,99 sans correction par un étalon interne (B) en utilisant une homogénéisation et une digestion du tissu Protéinase K assistées par solvant, suivies d’un mélange Purification en mode SPE à l’aide de la microplaque SPE et des réactifs OligoWorks.
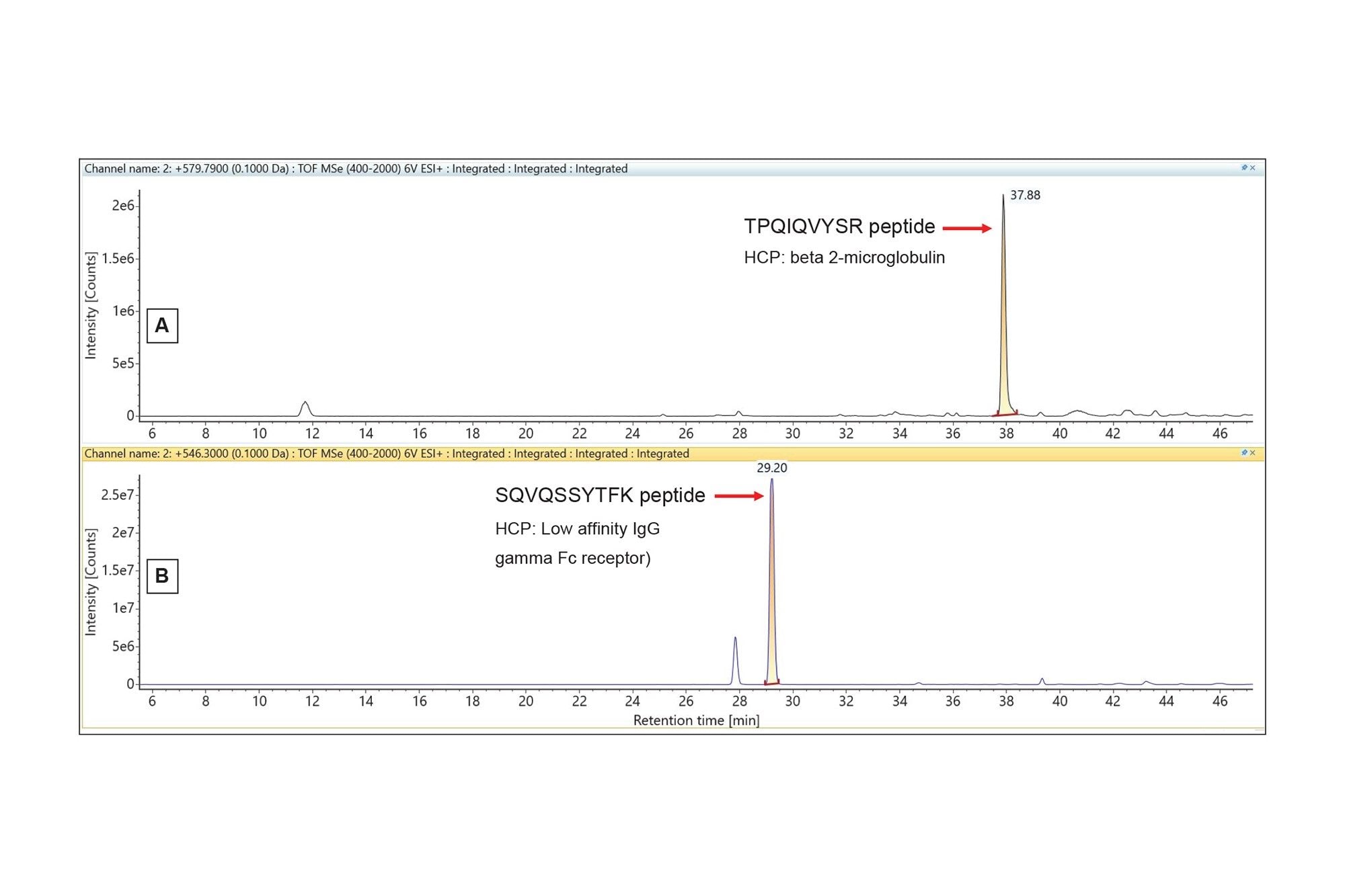
Chromatogrammes de masse extraits pour deux peptides HCP de faible abondance identifiés dans la digestion du mAb NIST à l’aide de l’analyse HCP de découverte.