基于Xevo™ MRT和全球天然产物社会分子网络系统(GNPS) 进行绿茶提取物成分分析
张永明,何青秀,龚智宏,曾玮 沃特世公司
Waters Corporation, United States
Published on September 10, 2025
摘要
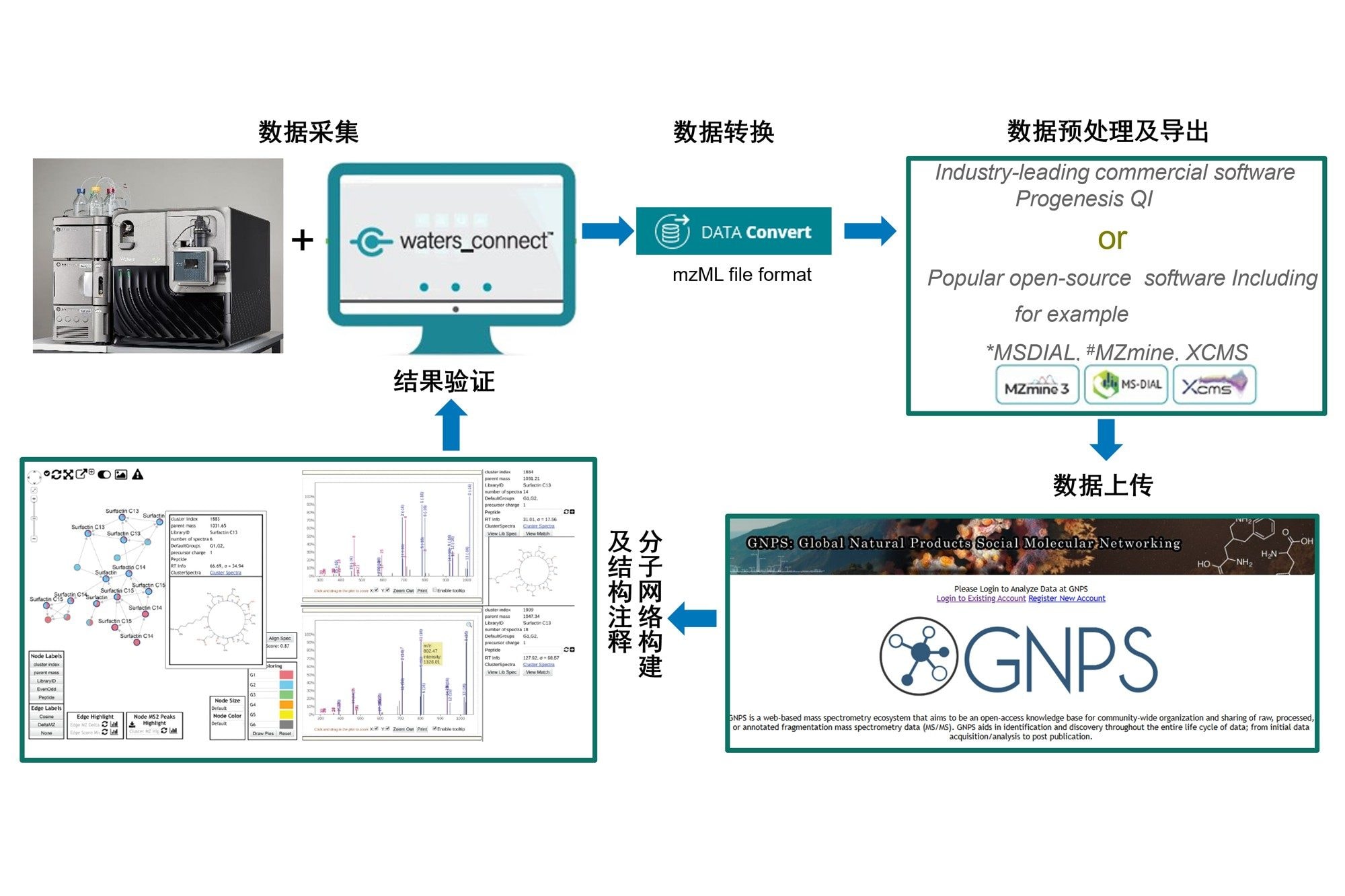
全球天然产物分子网络系统(Global Natural Products Social Molecular Networking,GNPS)是一个基于网络的质谱分析生态系统,它通过分子网络分析、谱图库搜索、高通量去重复等功能,助力天然产物和代谢组学研究。其中特征分子网络分析(Feature-Based Molecular Networking,FBMN)是其核心功能之一:它通过计算质谱图间相似性,将结构相似的有机物聚集成簇,形成 “分子家族”,这能直观呈现各成分关系,有助于发现结构类似物或新化合物。而 waters_connect平台自带的DATA Convert数据转换工具,在数据采集的同时可以直接将数据转换为.mzML通用格式,能够非常方便兼容第三方软件进行数据处理。我们以绿茶提取物为例,演示如何基于Xevo MRT MS和GNPS进行天然产物成分分析,图2为其工作流程图。
产品特点
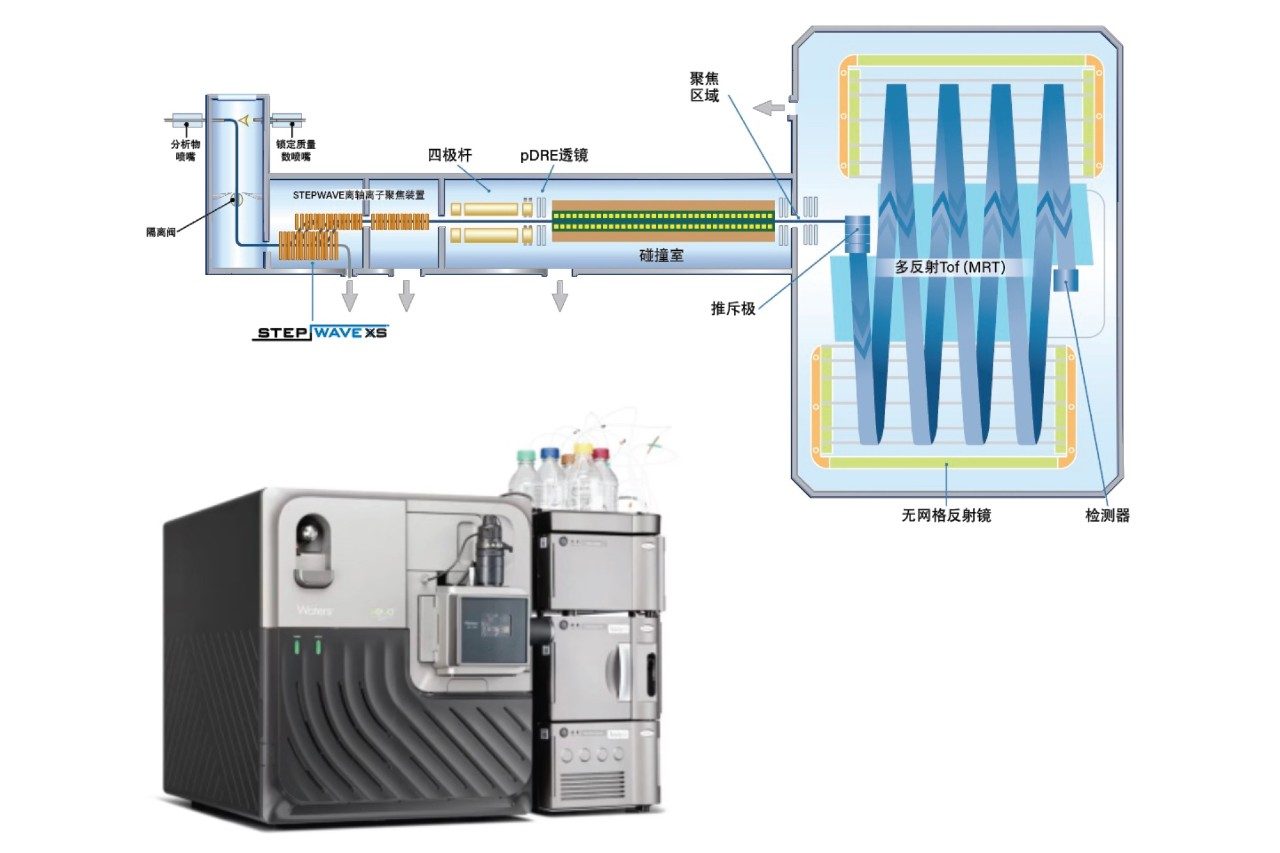
- Waters Xevo™ MRT MS采用多反射超高分 辨率飞行时间(MRT)技术可在较宽的m/z 范围内提供始终如一的质谱分辨率,即使在100 Hz的扫描速率下分辨率结果也不会受到影响。
- Xevo MRT MS经过精心设计,将高端技术 融入台式平台中,可帮助用户解决棘手难题。创新的采集系统配备双增益模数转换器,在高动态范围内亦可保持长期的系统稳定性,从而确保始终如一的优异数据质量、耐用性和重现性。
- 在Xevo MRT质量分析器内(见图1),4 m飞行 路径将发生总共8次反射,在高达100 Hz的扫描速率下,分辨率可达100,000 FWHM, 实现出色的MS/MS性能。
实验方法及参数
液相色谱条件
|
液相系统: |
ACQUITY™ Premier UPLC System |
|
样品: |
Green Tea Extract Mix p/n: 186006962 |
|
色谱柱: |
ACQUITY UPLC HSS T3 (2.1 x 100 mm, 1.8 μm) |
|
流动相A: |
0.01% 甲酸水 |
|
流动相B: |
乙腈 |
|
柱温: |
40 ˚C |
|
样品室温度: |
10 ˚C |
|
进样量: |
1 μL |
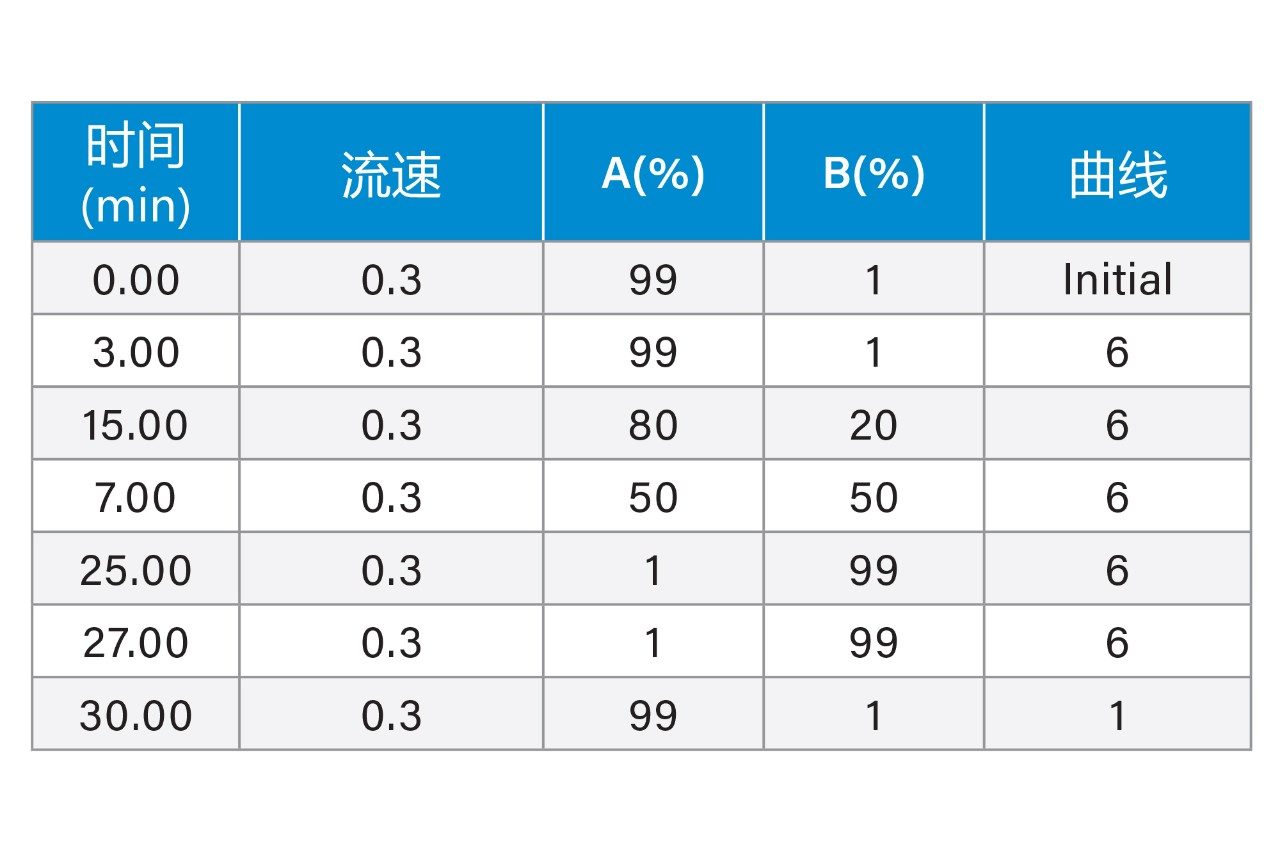
梯度
质谱条件
|
质谱系统: |
Xevo MRT MS |
|
|
采集模式: |
DDA, Top 15 |
|
|
采集范围: |
m/z 50–1,200 |
|
|
MS采集速率: |
10 Hz |
|
|
MS/MS采集速率: |
20 Hz |
|
|
离子化模式: |
ESI- |
|
|
离子源温度: |
120 ˚C |
|
|
脱溶剂气温度: |
550 ˚C |
|
|
毛细管电压: |
1.0 kV |
|
|
锥口电压: |
20 V |
|
|
锥口气流速: |
100 L/hour |
|
|
脱溶剂气流速: |
1,000 L/hour |
数据处理软件
使用waters_connect™ 进行数据采集,后续使用Progenesis QI或MS DIAL进行数据预处理后导出文件,上传预处理文件至GNPS1网站进行特征分子网络分析。
实验流程
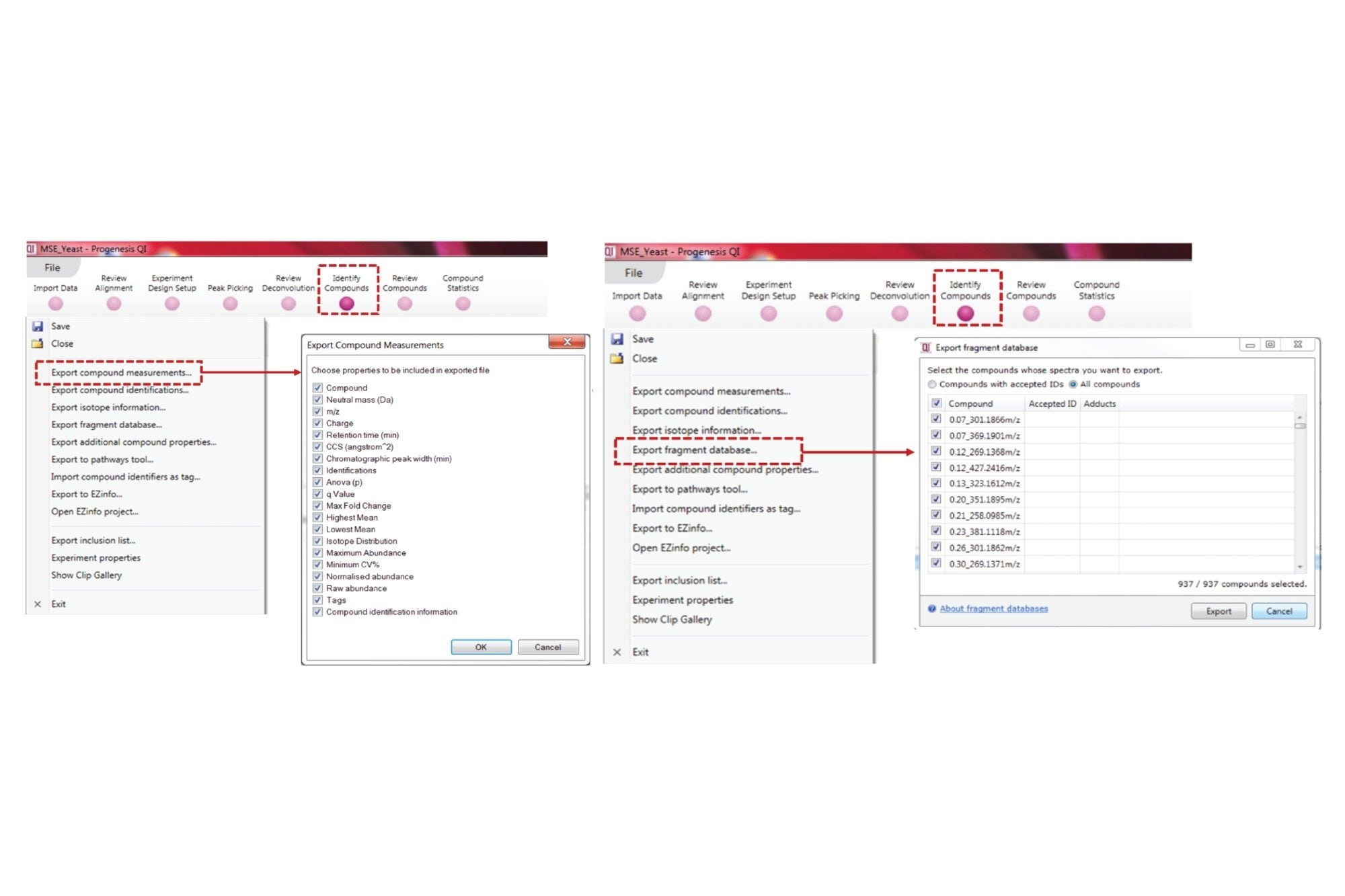
1. 数据预处理:在进行特征分子网络分析之前,首先需要借助代谢组学软件进行数据预处理(峰提取,峰对齐,样本分组,解卷积等),waters_connect平台自带的DATA Convert数据转换工具,在进行数据采集的同时可以直接将数据转换为.mzML通用格式,供Progenesis QI,MS DIAL2,Mzmine3, XCMS4 等组学软件对数据进行处理,这确保了在适应多样化工作流程时具备相当高的灵活性,同时通用格式也简化了跨团队的数据共享。 以沃特世官方组学软件 Progenesis QI为例,在使用其完成数据预处理后, 可以选择导出“Feature quantification table”和“MS2 MGF”两个文件,用于后续的FBMN分析,见图3。
此外,以第三方代谢组学软件MS DIAL为例,可以选择直接加载.mzML格式数据,数据导入并处理后可以得到5,559个特征离子的MS/MS谱图,见图4。
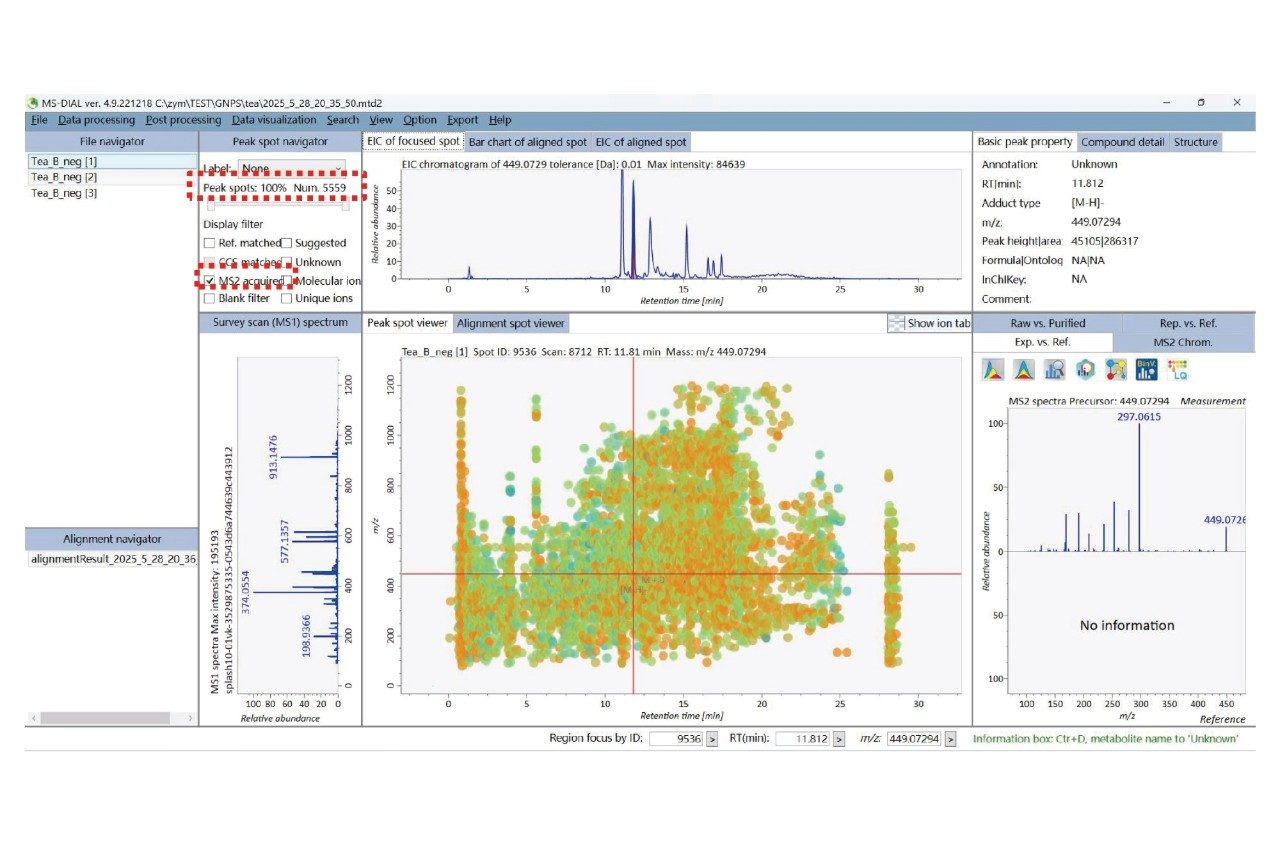
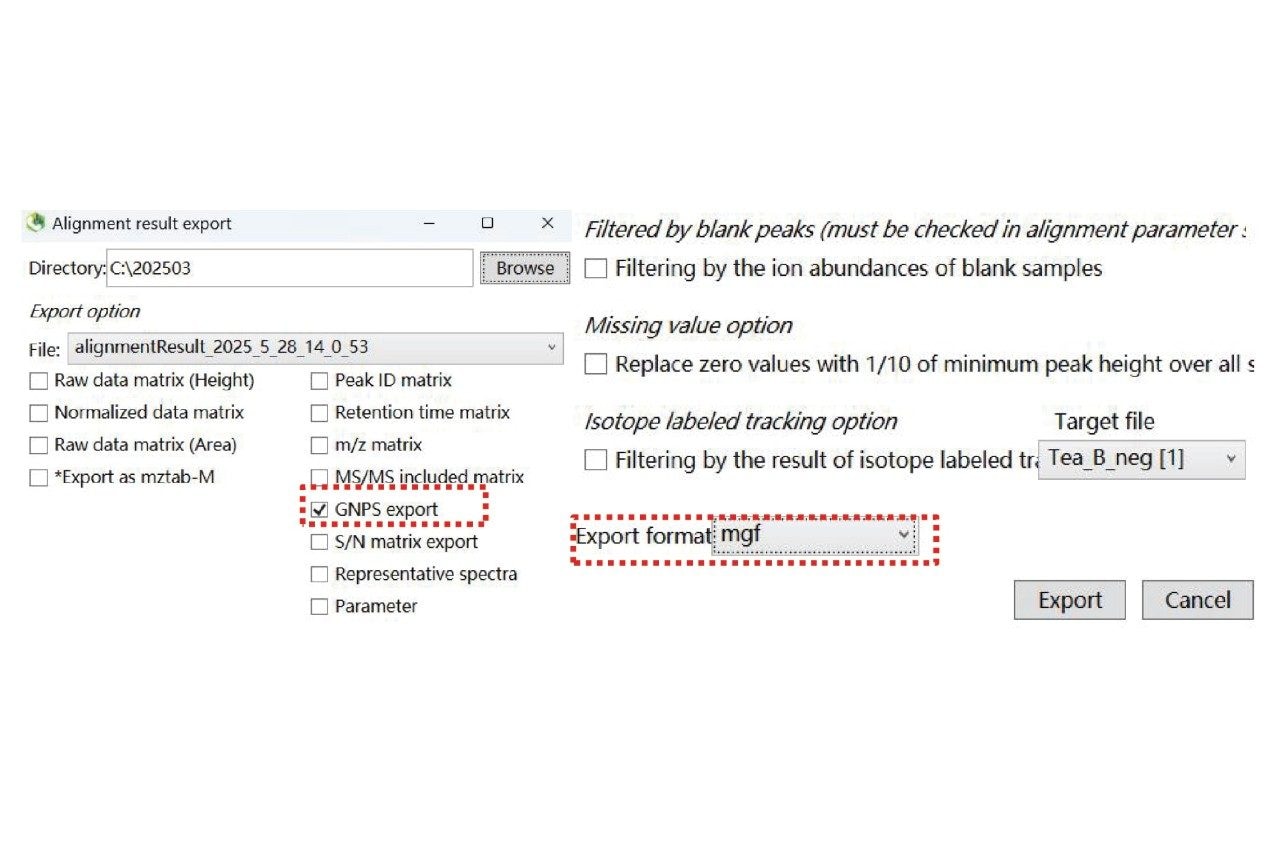
在MS DIAL菜单栏,选择“Export>Alignment result”,可导出“Feature quantification table”和”MS2 MGF”两个文件,用于后续的FBMN分析 ,见图5。
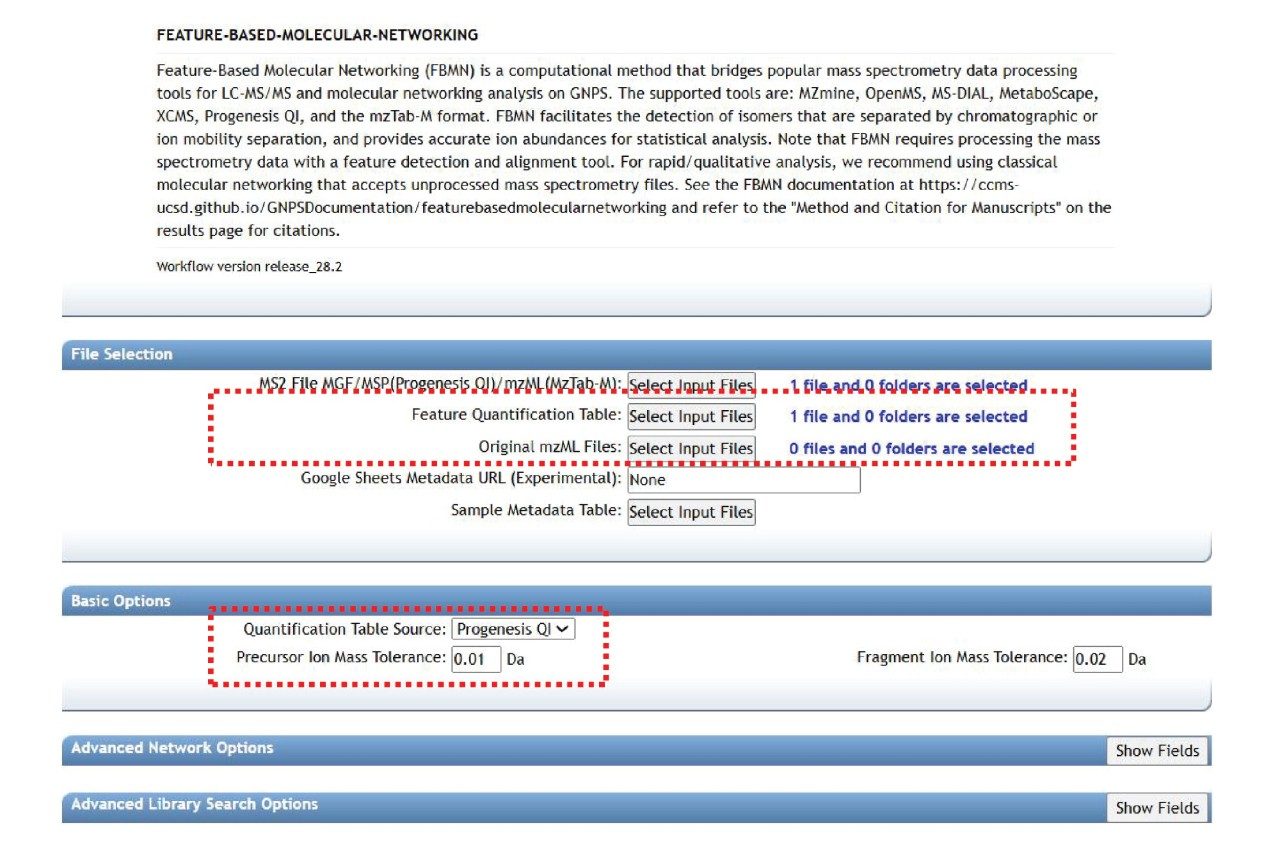
2. FBMN分析:登录GNPS网站,选择“Feature Networking”(注意和“Molecular Networking”有区别), 进入FBMN界面,提交上述的两个文件;此外可以选择加载.msp格式的MS/MS数据库(程序会自动加载GNPS网站自带的数据库包),并适当调整峰提取参数后,提交并等待结果,见图6。
实验结果
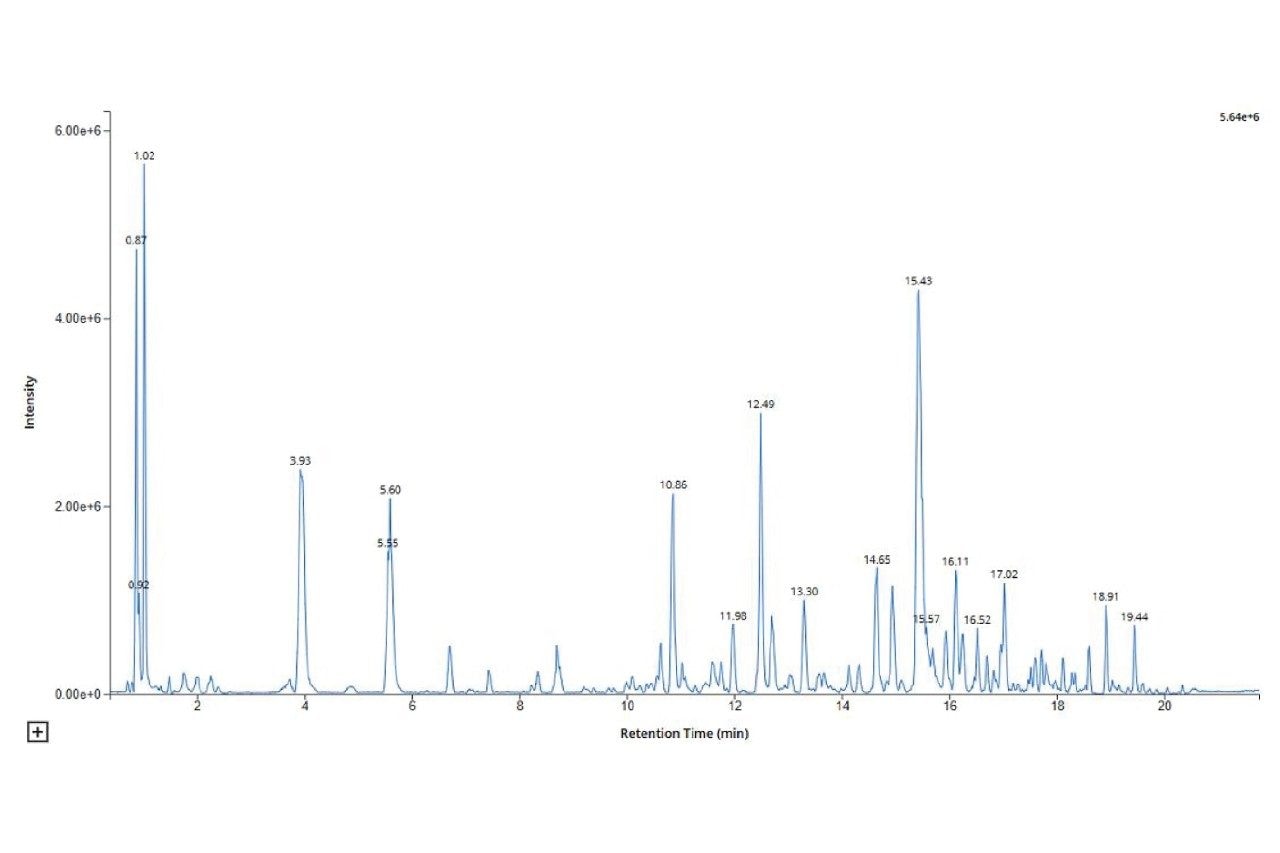
1. 绿茶提取物样品BPI图,见图7,其色谱分离度良好。
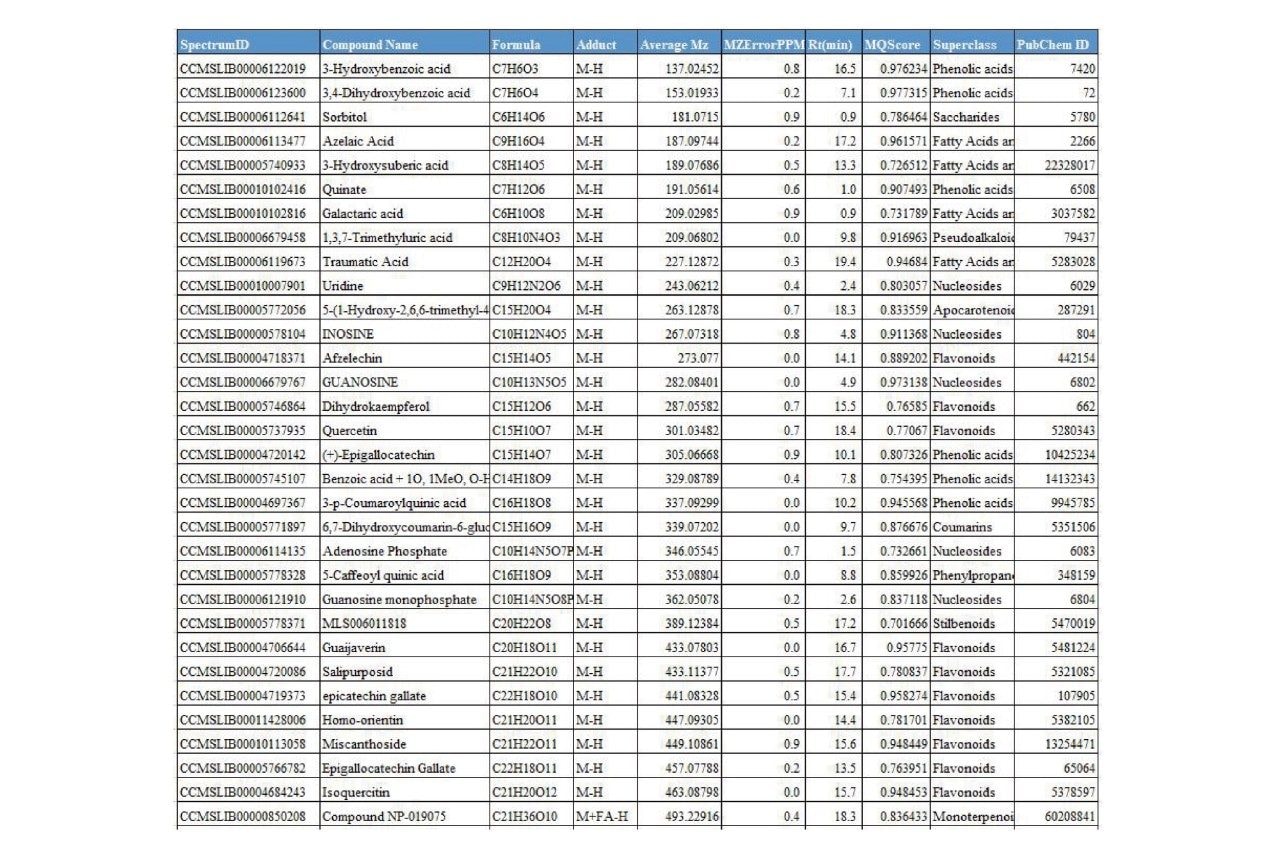
2. 样品鉴定结果。 GNPS网站搜库共得到203个化合物的鉴定结果,包含84种黄酮、22种酚酸、9种苯丙素、12种有机酸及其衍生物、12种核苷、5种香豆素、6种糖类、19种脂质,以及34种其他类型化合物,部分化合物鉴定结果如下表所示,质量偏差均小于1 ppm,显示出Xevo MRT优异的质量精度,见表1。
3. 天然产物结构类似物分析结果 通过FBMN分析,可以得到236个分子网络团簇,如表2所示。
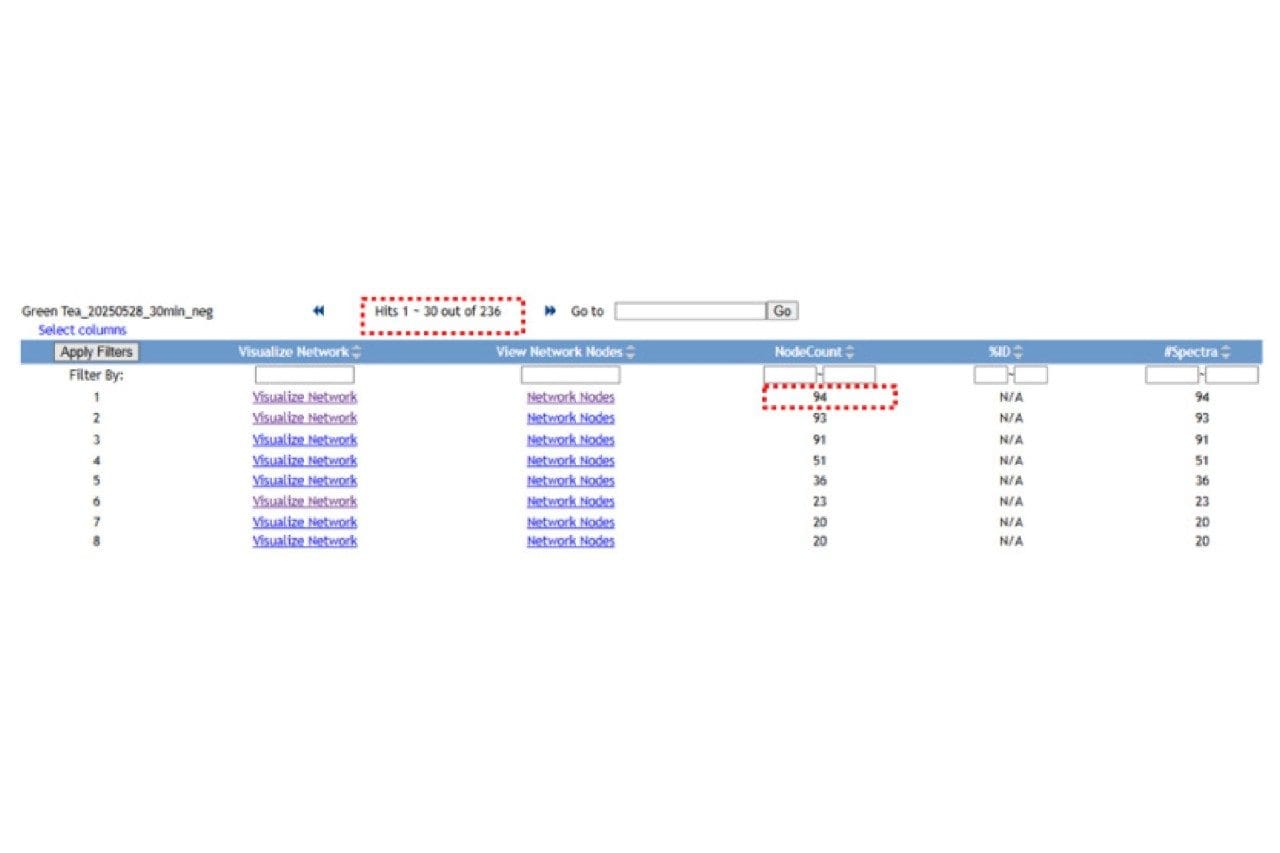
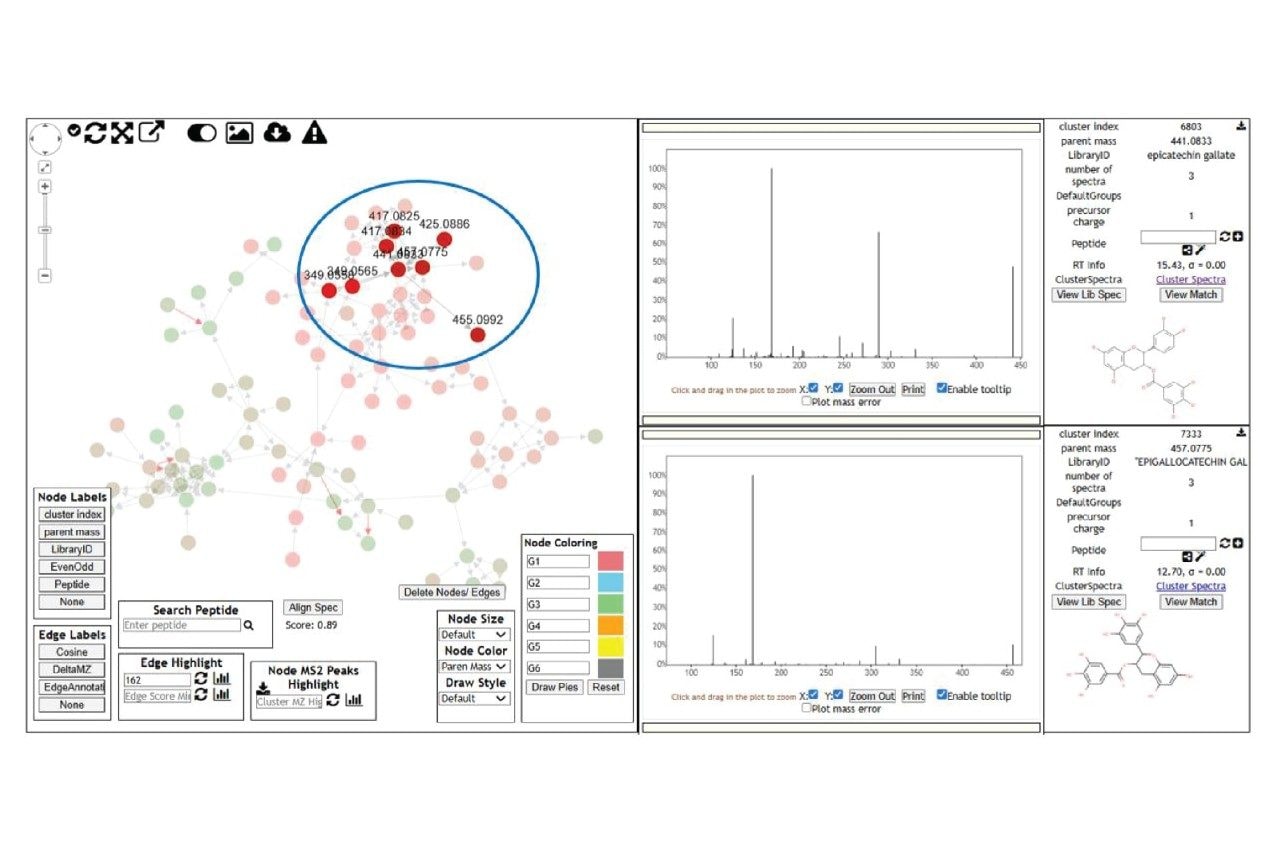
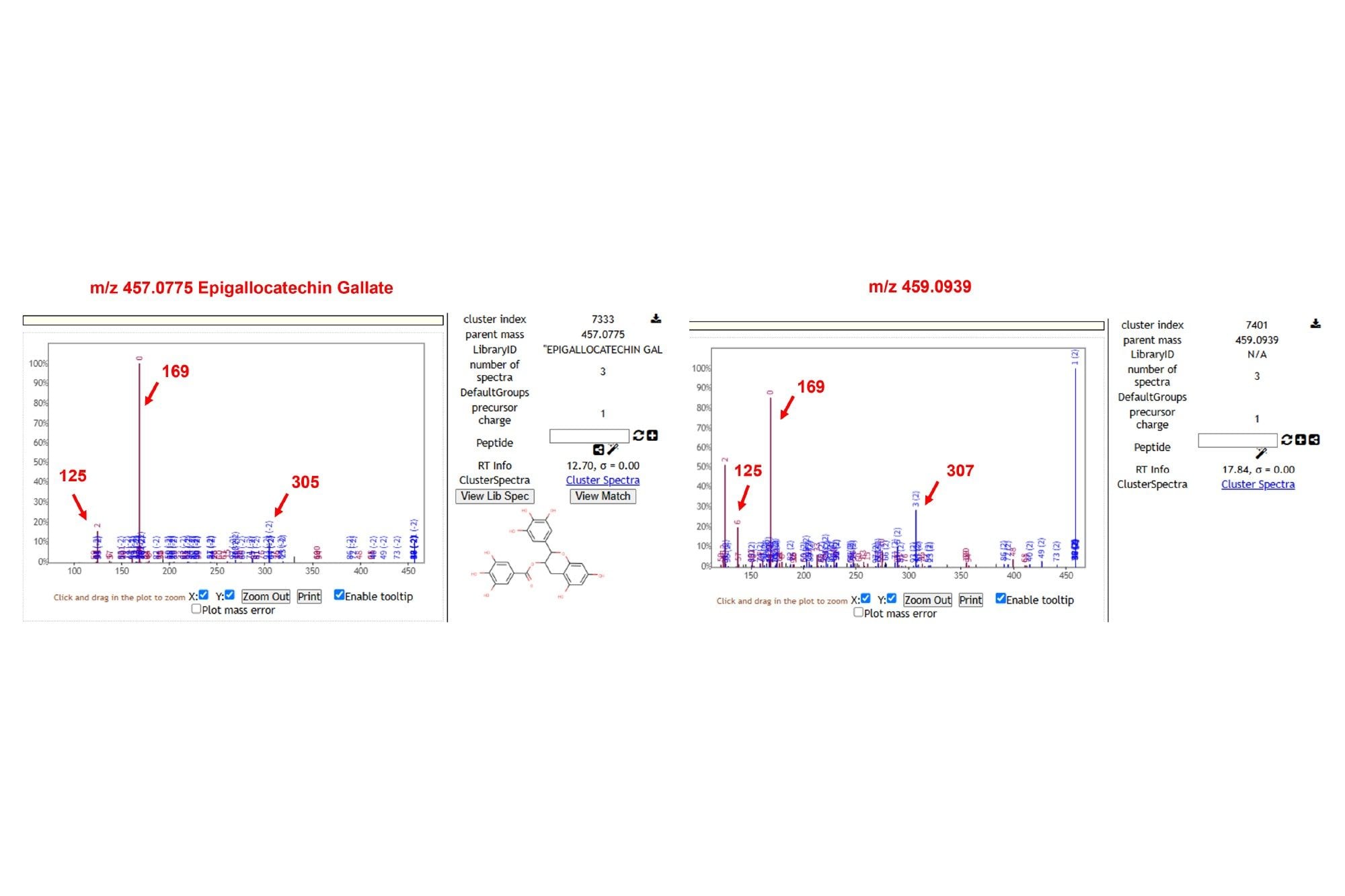
如图8所示,以第一个分子网络为例,共有94个节点,其中离子m/z 441.0833 GNPS搜库结果为Epicatechin gallate(表儿茶素没食子酸酯),与其结构相似度较高的离子有m/z 417.0825、m/z 425.0886、m/z 431.0980、m/z 455.0992、m/z 457.0775,除m/z 457.0775被鉴定为Epigallocatechin Gallate之外,其余四个离子均未被GNPS搜库匹配到。
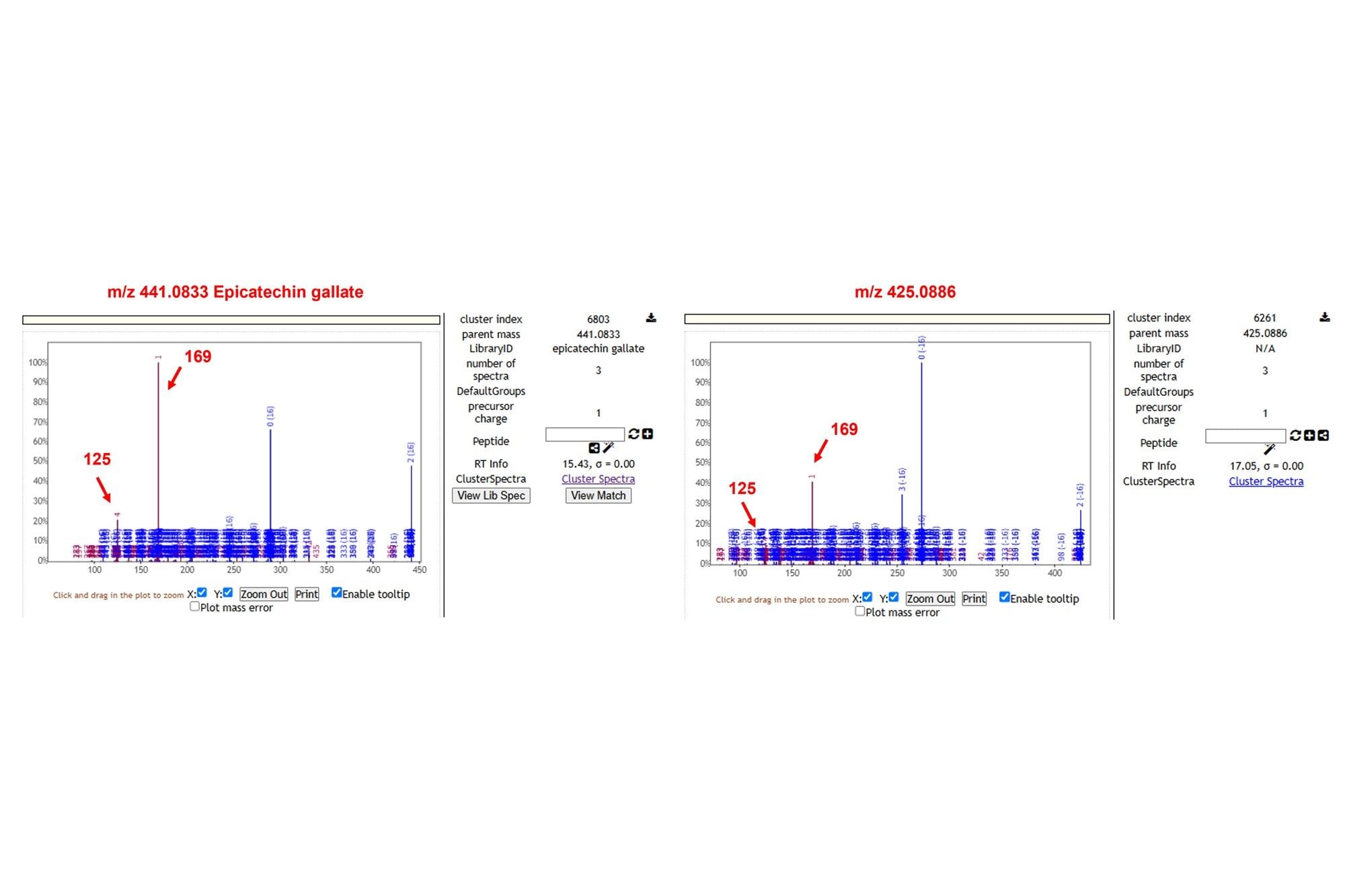
以离子m/z 425.0886为例,其与Epicatechin gallate的MS/MS谱图对比如图9所示,存在两个共有碎片离子125、169,对应没食子酸基团;另外,两者有多个碎片离子质荷比相差16(少一个羟基),表明该物质黄酮骨架上缺少一个羟基基团;最终推测其为Epiafzelechin-3-O-gallate。 m/z 425.0886、m/z 441.0833、m/z 457.0775三个化合物属于结构类似物,都含有没食子酸基团,主要区别在于黄酮骨架上酚羟基的数目不同。
再以m/z 459.0939为例,其与m/z 457.0775 Epigallocatechin Gallate的MS/MS谱图对比,如图10所示,同样存在两个共有碎片离子125、169,表明其存在没食子酸基团;另外存在一个质荷比相差2的离子(2H),推测其黄酮骨架发生了加氢开环反应,未见文献报道,可以对其结构进行合理推测。
4. 使用waters_connect平台进行结果验证
如图11所示,分别为m/z 425.08、m/z 441.08、m/z 457.07和m/z 459.0939对应的二级谱图及推测分子结构,并基于waters_connect平台对其二级碎片进行了结构注释,它们各自所有的高强度碎片离子均被注释,进一步表明了推测结果的合理性。
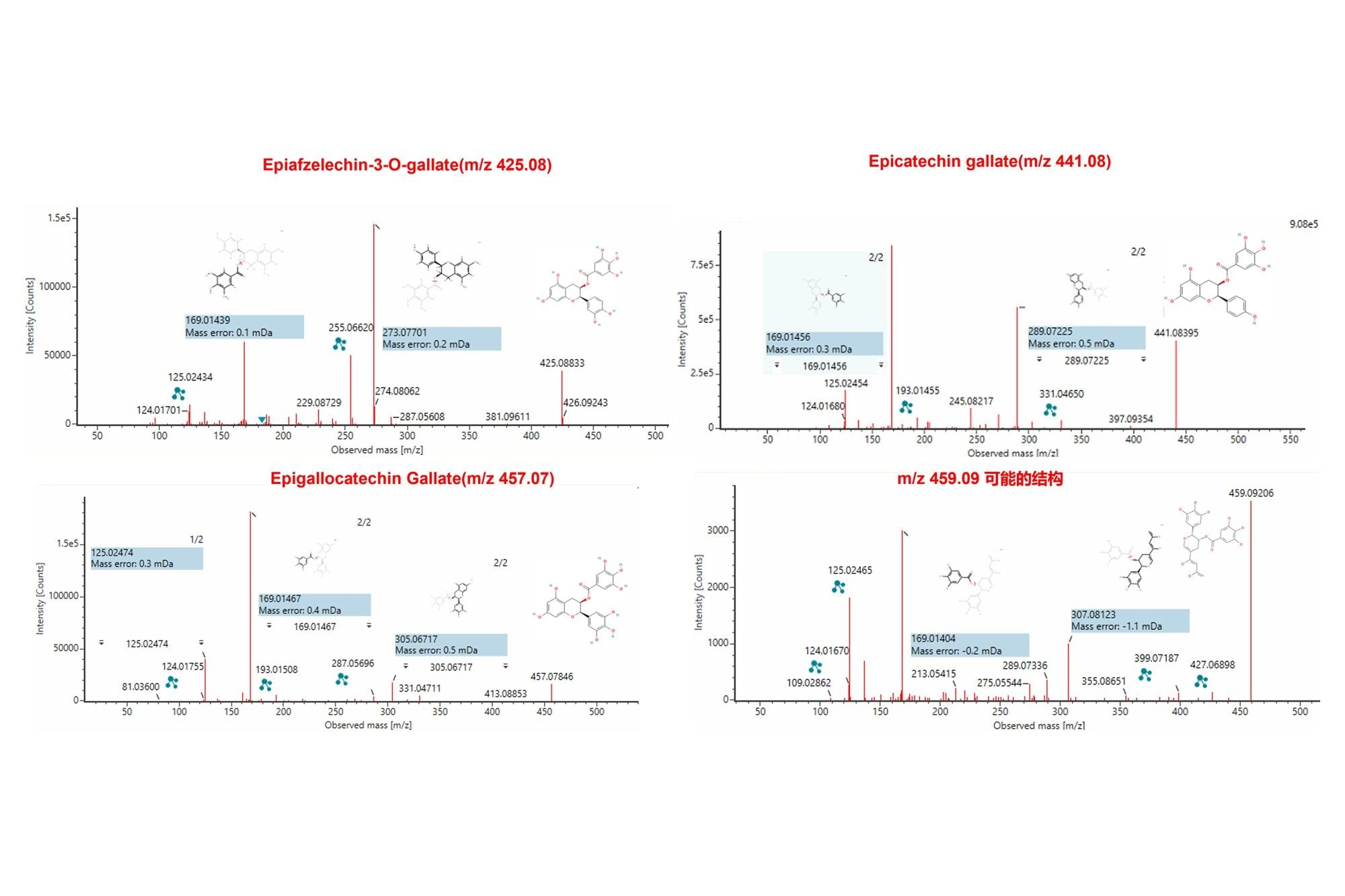
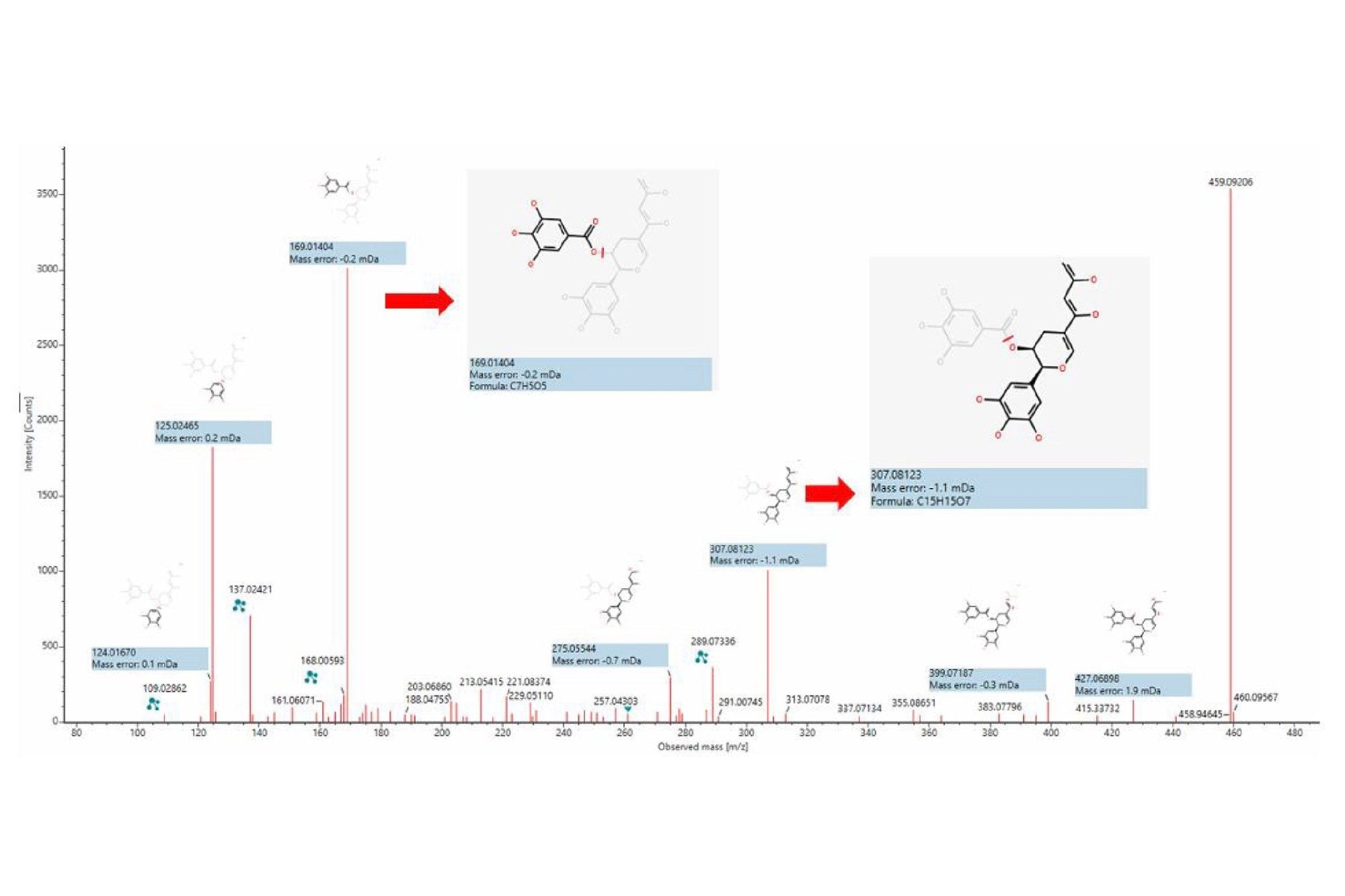
其中m/z 459.09为推测的分子结构,两个关键碎片离子169和307对应的碎片结构断裂合理,如图12所示,可能属于绿茶提取物中新发现的化合物,后续还需要进一步证明。
类似地可以进行其他结构类似物离子的MS/MS谱图比对和结构解析,其中m/z 417.0825推测为Kaempferol 3-O-α-L-arabinoside,m/z 431.0980推测为kaempferol 3-rhamnoside,m/z 455.0992推测为 Epicatechin 3-O-(3-O-methylgallate)。
实验结论
使用Xevo MRT MS联合GNPS对绿茶提取物进行化学成分分析:
- 得益于Xevo MRT MS的高分辨率及高扫描速度,使用DDA TOP15进行数据采集,可以获得非常多高质 量的MS及MS/MS谱图,绿茶提取物样本在单一负模式下可以得到5,559个特征离子的MS/MS谱图;
- waters_connect平台自带的DATA Convert数据转换工具,在数据采集的同时可以直接将数据转换 为.mzML通用格式,能够非常方便地供Progenesis QI、MS DIAL等组学软件对数据进行处理,提高数据分析效率;
- GNPS利用Xevo MRT MS提供的高质量MS/MS谱图,可以快速鉴定到203个化合物,包含84种黄酮、22 种酚酸、9种苯丙素、12种有机酸及其衍生物、12种核苷、5种香豆素、6种糖类、19种脂质,以及34种其他类型化合物,该方法也可以应用于其他复杂天然产物样品的快速分析鉴定;
- 通过FBMN分析,可以得到236个分子网络团簇,其中以m/z 441.0833 epicatechin gallate为例,可以找 出6种结构类似物,其中有5种未被GNPS注释鉴定,通过MS/MS谱图比对和结构解析,分别被推测鉴定,并基于waters_connect平台对其分子结构进行注释验证,其中包含一种未被文献报道的物质,应该属于绿茶提取物中新发现的化合物 。
参考文献
- Louis-Félix Nothias, et al. “Feature-based molecular networking in the GNPS analysis environment.” Nature Methods (2020):1–4.
- Tsugawa, Hiroshi , et al. “MS-DIAL: data-independent MS/MS deconvolution for comprehensive metabolome analysis.” Nature Methods 12.6(2015):523–526.
- Heuckeroth, Steffen, et al. “Reproducible mass spectrometry data processing and compound annotation in MZmine 3.” Nature Protocols 19.9(2024).\
- Forsberg, et al. “Data processing, multi-omic pathway mapping, and metabolite activity analysis using XCMS Online.” Nature Protocols Erecipes for Researchers (2018).
720008922ZH, 2025年6月