利用Xevo™ G3 QTof进行细胞外囊泡的代谢组学和脂质组学分析
仅供研究使用,不适用于诊断。
摘要
脂质组学和代谢组学的LC-MS工作流程被广泛用于确定潜在的疾病生物标志物,以及深入了解所研究体系的潜在生物学机制。生物标志物通常以多种形式存在,常规上需要从血浆、血清或尿液等生物体液中分离。然而,近年来,细胞外囊泡(EV)作为潜在的候选生物标志物引起了广泛关注。在本应用纪要中,我们评估了可应用于EV的不同样品前处理缓冲液,以实现理想的脂质组学和代谢组学提取效果。
优势
- 全面的代谢组学和脂质组学分析,结合相对免标记定量,采用数据非依赖型工作流程MSE
- Xevo G3 QTof可对脂质组和代谢组进行深入分析,展现样品内高达5个数量级的动态范围
- 应用ACQUITY™ Premier LC系统可提高极性代谢物/脂质(例如磷酸化物质)的回收率,这一点对于低丰度生物相关化合物尤为重要
- 使用内部软件工具(即waters_connect™)完成数据处理和化合物鉴定,该工具同时兼容Lipostar和MARS等第三方解决方案
- 内部和第三方软件工具的输出结果与统计/数据可视化工具(如MetaboAnalyst)的兼容性,展现了工作流程的灵活性,提供更大的统计和化合物鉴定覆盖率
简介
细胞外囊泡(EV)是一类具有脂双层膜结构的异质性、非增殖性纳米颗粒。几乎所有的细胞都会释放EV,它们携带着反映细胞来源的DNA、RNA、脂质、蛋白质和代谢物。EV在包括外周血在内的多种生物体液中循环,并且可以方便地监测,其生理特性也与其亲代细胞相符。它们在生物体液中的浓度似乎也与疾病状态相关1。 近年来,EV被视为潜在的疾病生物标志物,特别是在肿瘤学和神经学等领域2-3。 随后,针对囊泡通信途径的组学研究相继开展。本研究的目的是优化一种与LC-MS分析兼容的多组学样品前处理方法,用于EV富集样品的分析。
实验
样品前处理
根据美国典型培养物保藏中心(American Type Culture Collection, ATCC)的指导原则,将C2C12小鼠成肌细胞培养增殖至汇合状态,然后向肌管方向分化4。 随后,通过差速超速离心法从培养基中分离EV。为了评估用于LC-MS分析的样品提取效果,使用了三种不同的裂解缓冲液。这些缓冲液为:(i) 2%十二烷基硫酸钠(SDS);(ii) 2% SDS,含20 mM Tris;(iii) 无裂解缓冲液(NLB)。
液相色谱条件(代谢组学 - HILIC)
|
液相色谱系统: |
ACQUITY Premier (FTN) |
|
色谱柱: |
ACQUITY UPLC™ BEH™ Amide(2.1 mm × 100 mm, 1.7µm;P/N:186009505) |
|
柱温: |
40 ˚C |
|
进样体积: |
2 µL不充满定量环 |
|
流速: |
0.7 mL/min |
|
流动相A: |
5:95乙腈:水 + 0.1%甲酸,10 mM甲酸铵 |
|
流动相B: |
95:5乙腈:水 + 0.1%甲酸,10 mM甲酸铵 |
|
梯度: |
初始条件为100% B;0.1~5.0 min,100%~80% B;5.0~6.0 min,80%~50% B;6.0~6.5 min保持50% B;6.5~7.0 min,50%~100% B;7.0~10.0 min,重新平衡至初始条件。 |
液相色谱条件(脂质组学- 反相)
|
液相色谱系统: |
ACQUITY Premier (FTN) |
|
色谱柱: |
ACQUITY Premier CSH™ C18(2.1 × 100 mm, 1.7 µm;P/N: 186009461) |
|
柱温: |
55 °C |
|
进样体积: |
2 µL不充满定量环 |
|
流速: |
0.4 mL/min |
|
流动相A: |
600:390:10乙腈:水:1 M甲酸铵,0.1%甲酸 |
|
流动相B: |
900:90:10 IPA:乙腈:1 M甲酸铵,0.1%甲酸 |
|
梯度: |
初始条件为50% B;0.5 min,50%~53% B;0.5~4.0 min,53%~55% B;4.0~7.0 min,55%~65% B;7.0~7.5 min,65%~80% B;7.5~10 min,80%~99% B;10~11 min,99% B;11~12 min,重新平衡至初始条件。 |
质谱条件
|
质谱系统: |
Xevo G3 QTof |
|
离子源: |
ESI |
|
电离模式: |
ESI+/- |
|
毛细管电压: |
2.8 (+)/1.5 (-) kV |
|
采样锥孔电压: |
40 V |
|
离子源温度: |
150 °C |
|
脱溶剂气温度: |
500 °C |
|
锥孔气流速: |
150 L/h |
|
脱溶剂气流速: |
900 L/h |
|
碰撞能量: |
从20~45 eV逐步递增 |
|
扫描时间: |
0.1 s |
信息学软件
数据处理使用了多种软件平台,包括Lipostar和MARS(Mass Analytica,西班牙巴塞罗那)。统计分析除了使用MetaboAnalyst以外,还使用了数据处理软件平台中内嵌的统计工具5。 进一步的数据可视化通过Spotfire(Tibco,美国加利福尼亚州帕洛阿托)。
结果与讨论
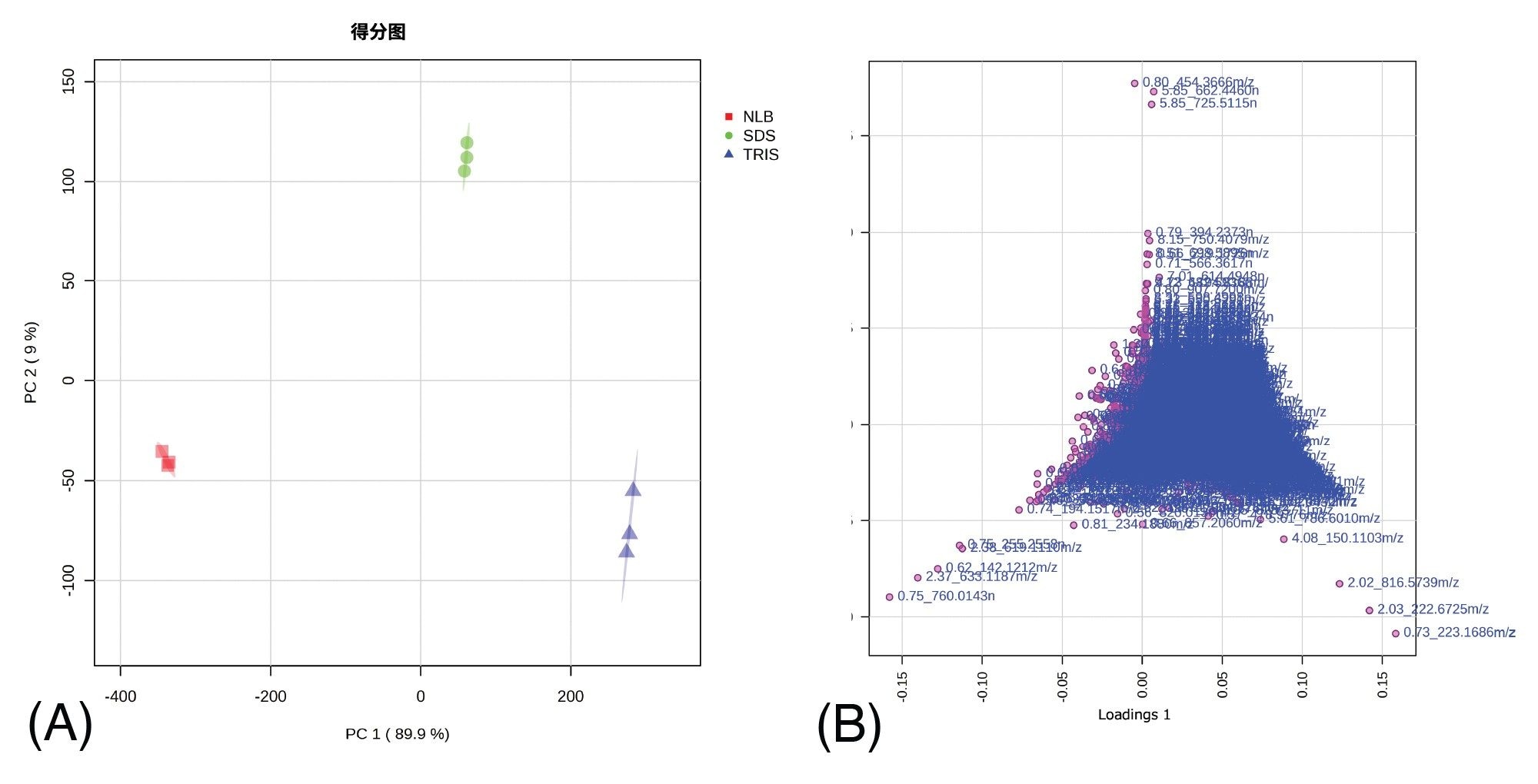
根据不同提取方案的EV相关的代谢组学和脂质组学提取数据,使用Xevo G3 QTof以非数据依赖性采集(DIA)模式进行MSE采集。随后,这些数据通过多种信息学工具进行处理(图1)。对脂质组学提取物相关的处理数据进行统计分析,结果显示不同提取方法之间存在显著差异。图2展示了代表反相(RP)分析(ESI+)的无监督主成分分析(PCA)。每种提取方法的技术重复样(n=3)都紧密聚集,表明该系统具有较高的技术重现性(这一点在ESI-数据中也得到了验证)。三种提取方案之间的明显分离表明,所提取的分析物和/或各分析物的提取量存在显著差异。相关的Loadings plot标识了导致PCA差异的特征,这些特征随后经过数据库检索进行确认。
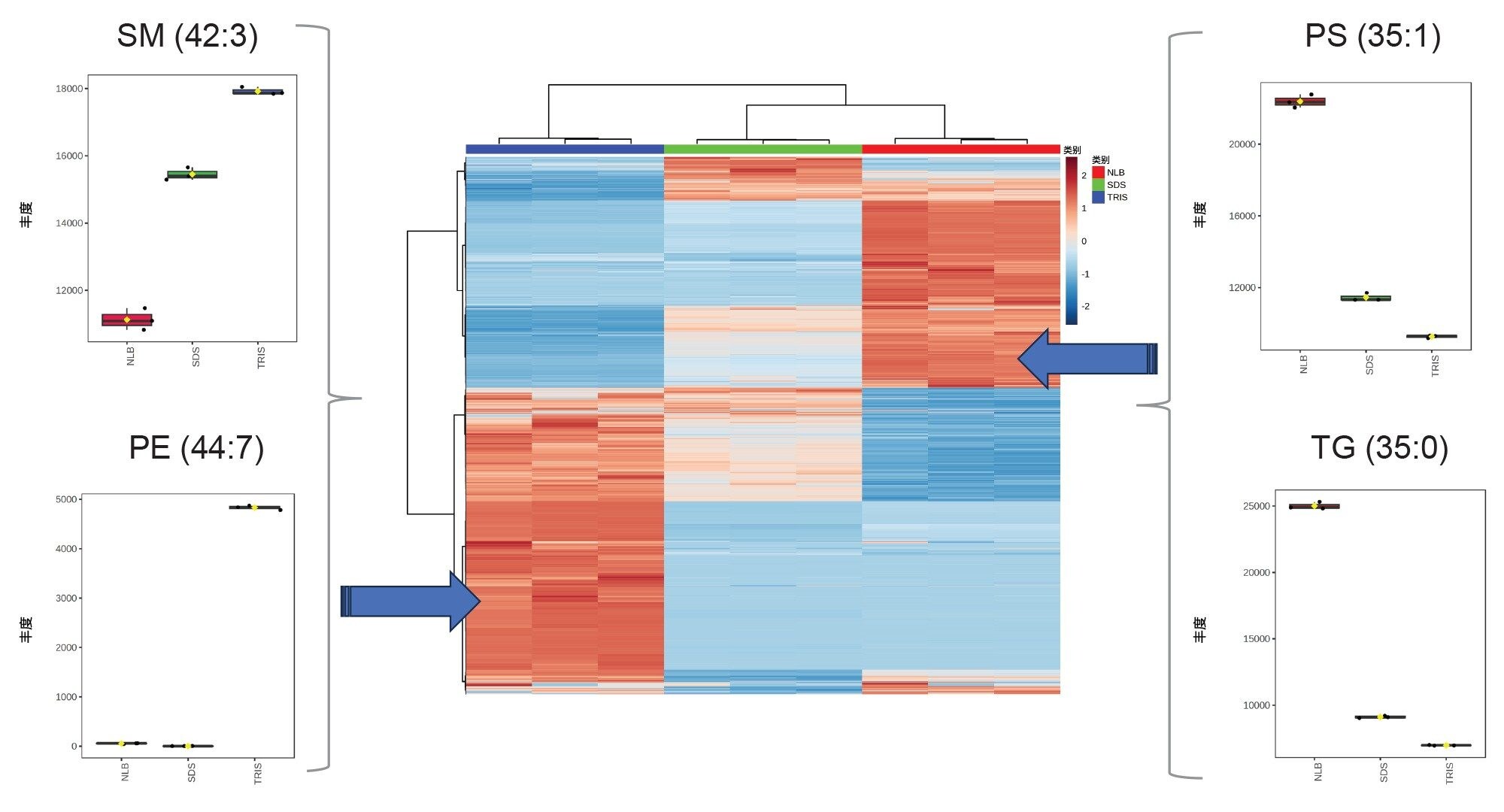
在对最具统计学相关性的脂质进行比较时,主要鉴定出的脂质类别包括甘油三酯、鞘磷脂和磷脂(图3)。比较三种缓冲液条件发现,大多数磷脂/鞘磷脂在Tris缓冲液中检测到较高的丰度,而使用NLB条件时,仅少部分磷脂的丰度较高(图3 - 热图/箱线图)。
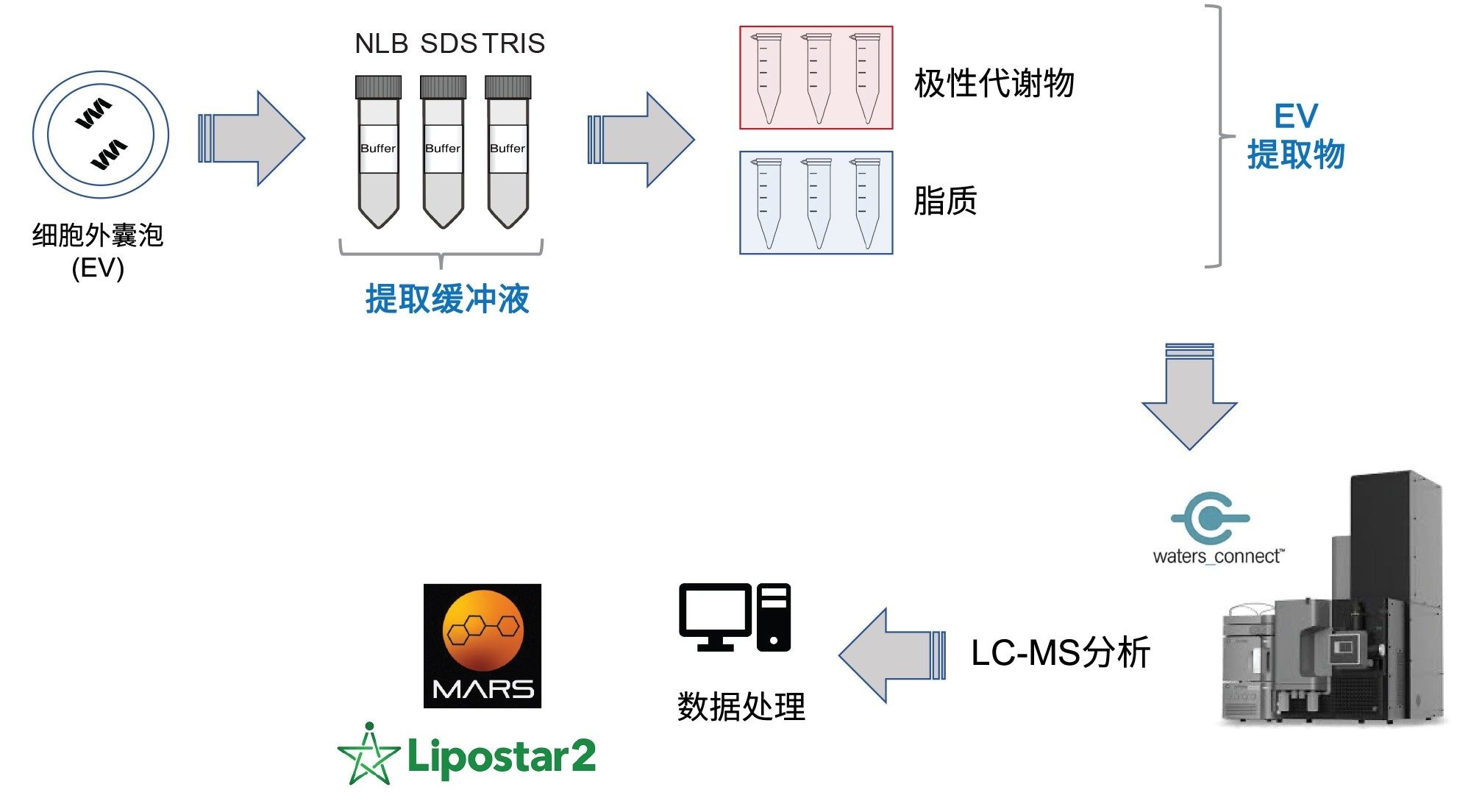
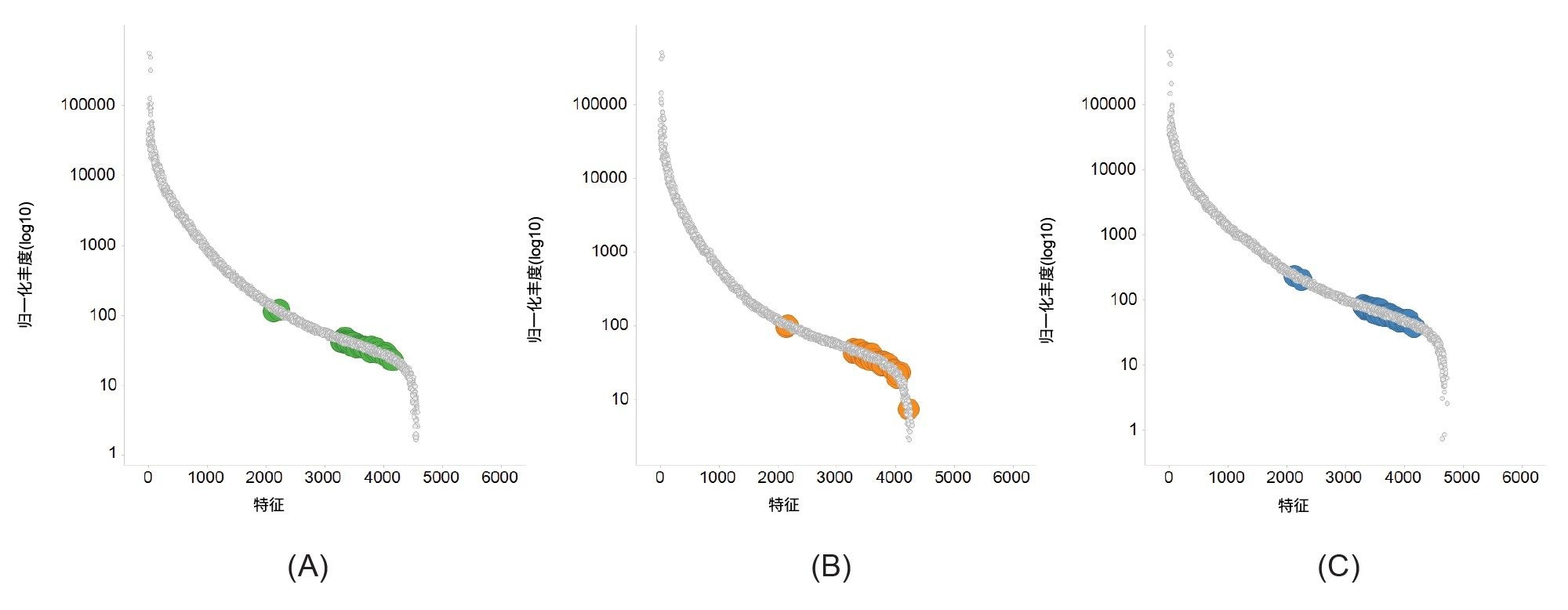
在所有三种缓冲液条件下均可以明显检测到磷脂,而这些关键磷脂的丰度相对较低,因此需要Xevo G3 QTof提供的高灵敏度和宽动态范围。归一化丰度曲线(图4)展示了样品内实现的宽动态范围(约5个数量级),其中大部分磷脂在Tris缓冲液中检测到的丰度略高。
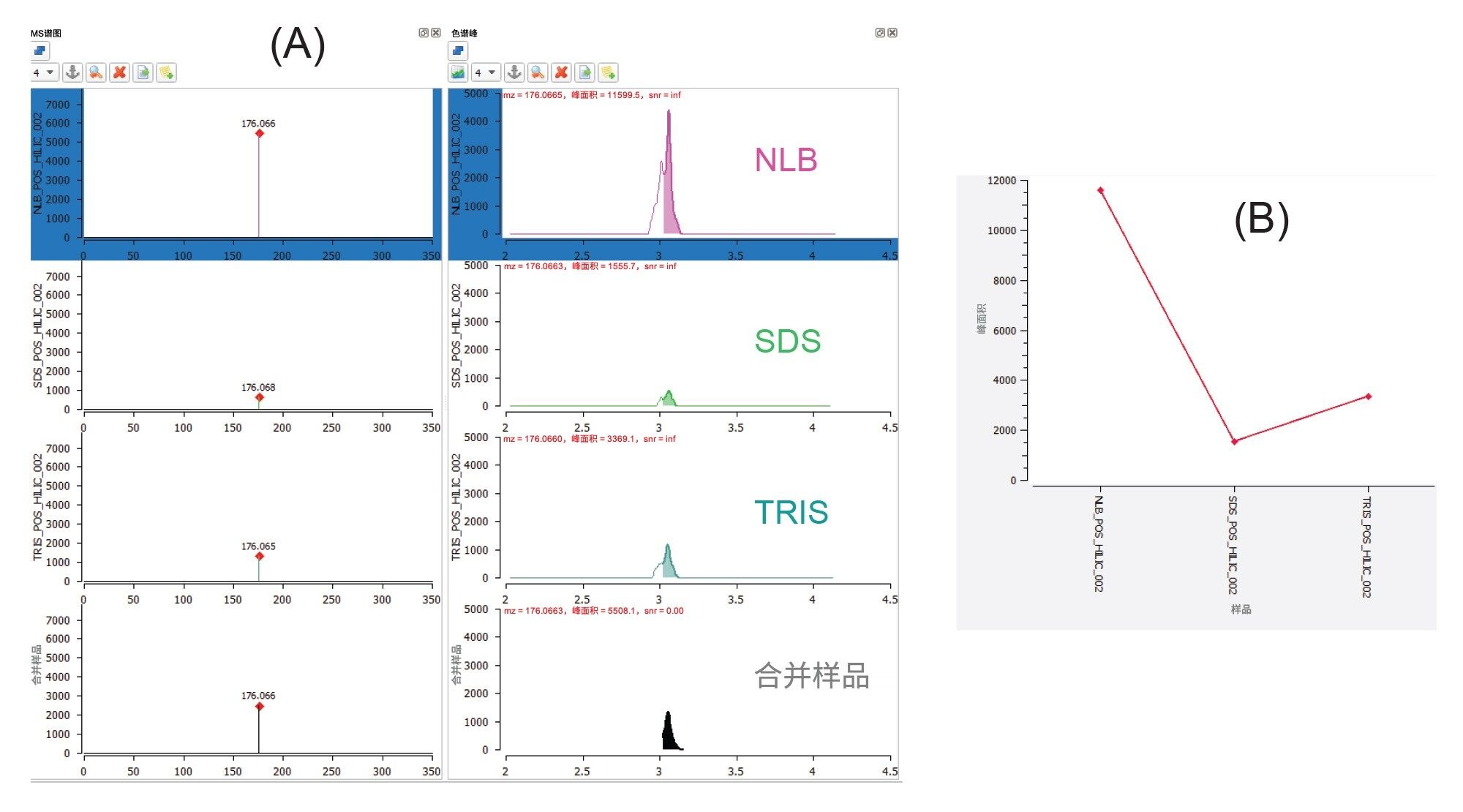
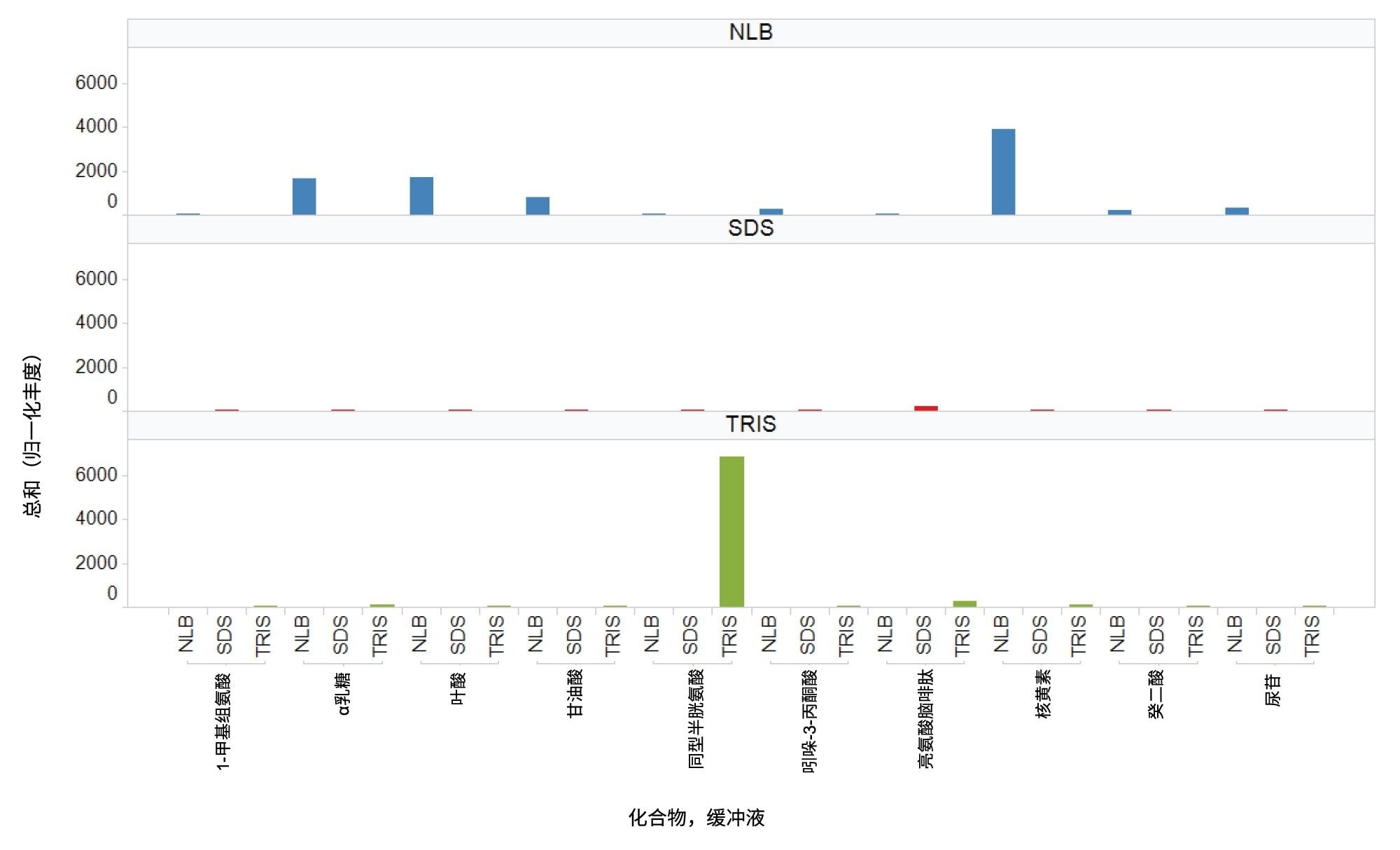
从富集EV样品中提取的极性代谢物的数量和种类在很大程度上取决于样品前处理过程中所用的提取缓冲液类型。一般来说,采用NLB条件可以(i)鉴定独特的代谢物,或(ii)对于三种缓冲液共有的代谢物,NLB通常可以获得更高的回收率。waters_connect API实用程序提供了额外的灵活性,支持使用第三方信息学工具解析数据。利用MARS代谢组学信息学软件包进行了峰提取和化合物鉴定搜索,同时提供了相对定量分析。胍基琥珀酸(图5)是其中的一种示例代谢物,可通过三种缓冲液条件提取;但在NLB提取物中的丰度至少高出四倍(如提取色谱图(图5A)和相关趋势图(图5B)所示)。此前已有研究报告这类化合物与癌症衍生细胞相关6。 图6进一步展示了这一观察结果在几种其他化合物类别(同型半胱氨酸除外)中的体现。
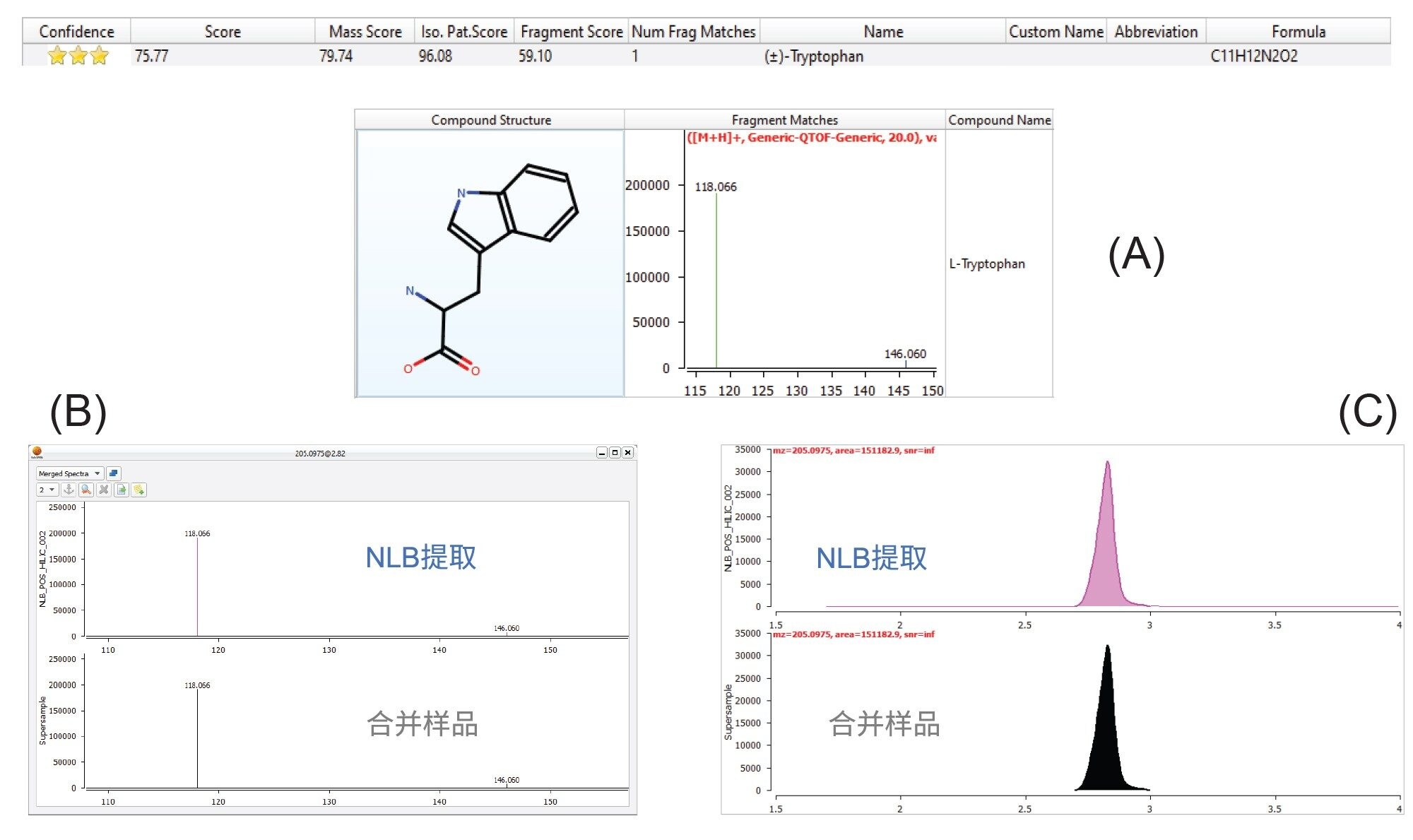
研究中通过MARS工作流程鉴定出了几种重要的生物学相关极性代谢物,包括氨基酸、脂肪酸、吡啶和吲哚。其中,色氨酸(图7)仅能从NLB提取物中鉴定出,其鉴定结果高度可信,此前的研究表明,它在骨骼肌的信号通路中发挥着关键作用7。质量精度(母离子和碎片离子)、同位素模式、碎片离子匹配以及保留时间等因素都会共同影响整体可信度得分。
结论
本研究通过采用三种不同的缓冲液条件(NLB、SDS和Tris),成功展示了EV中脂质和极性代谢物的LC-MS分析。总体而言,提取缓冲液的选择会影响检出的脂质类别及其丰度,而对极性代谢组而言,缓冲液的选择影响更为显著(即,对于丰度和独特极性代谢物的检测方面)。Xevo G3 Qtof的引入,实现了在宽动态范围内低浓度化合物的检测。通过API与第三方信息学工具(如MARS)的集成,实现了高可信度的化合物鉴定和相对定量分析,展示了一套灵活的数据分析工作流程,适用于发现研究和小分子组学分析。
参考资料
- Rikkert, L.G., Beekman, P., Caro, J., Coumans, F.A.W., Encisco-Martinez, A., Jenster, G., et al.Toward Identification of Cancer by Tumor-Derived Extracellular Vesicles in Blood.Front.Oncol.(2020) (doi: 10.3389/fonc.2020.00608).
- Liu, L., Kawashima, M., Sugimoto, M., Sonomura, K., Pu, F. et al.Discovery of Lipid Profiles in Plasma-Derived Extracellular Vesicles as Biomarkers for Breast Cancer Diagnosis. Cancer Science (2023) (doi: 10.1111/cas.15935).
- Blommer, J., Pitcher, T., Mustapic, M, Eren, E., Yao, P.J., Vreones, M.P. et al.Extracellular Vesicle Biomarkers for Cognitive Impairment in Parkinson’s Disease.Brain (2023) (doi:10.1093/brain/awac258).
- https://www.atcc.org/resources/culture-guides/animal-cell-culture-guide.
- Pang, Z., Zhou, G., Ewald, J., Chang, L., Hacariz, O., Basu, N., and Xia, J. Using MetaboAnalyst 5.0 for LC-HRMS Spectra Processing, Multi-Omics Integration and Covariate Adjustment of Global Metabolomics Data. Nature Protocols (2022) (doi: 10.1038/s41596–022–00710–w).
- Harmati, M., Bukva, M., Bőrőczky, T., Buzás, K., Gyukity-Sebestyén, E. Cancer and Metastasis Reviews (2021) (doi: 10.1007/s10555–021–10014–2).
- Dukes, A., Davis, C., Refaey M.E., Upadhyay, S., Mork, S., Arounleut, P., Johnson, M.H., Hill, W.D., Isales, C.M., Hamrick, M.W. The Aromatic Amino Acid Tryptophan Stimulates Skeletal Muscle IGF1/p70s6K/Mtor Signaling in Vivo and the Expression of Myogenic Genes in Vitro.Nutrition (2015) (doi: 10.1016/j.nut.2015.02.011).
720008142ZH,2023年12月