使用高灵敏度离子对LC-MS对PCR引物和探针试剂进行质量数确认和纯度分析
供体外诊断使用。仅在部分国家/地区销售。
摘要
在SARS-CoV-2大流行疫情中,业界使用RT-qPCR试剂盒进行常规感染检测。此类疾病监测工作受益于使用经认证的高质量qPCR试剂。本应用纪要介绍了使用优化的IP-RPLC和符合法规要求的台式Tof仪器对SARS-CoV-2 CDC分析中使用的PCR检测试剂进行的质量评估。借助这种灵敏的方法,能够从10 pmol的核衣壳引物N1和N2(正向和反向序列)以及N1和N2探针中收集大量纯度信息。所有PCR试剂均产生了尖锐、对称的峰。采集负离子质谱图,可轻松观察到多电荷物质(高达[M-10H]10-),使我们能够明确测定RT-qPCR试剂盒试剂的分子量。使用UV与MS检测相结合的方法,评估试剂以及未纯化原料的纯度。因此,探讨了一个有趣的例子,即N2探针质量数漂移42.9的杂质。总之,这种结合高分离度、高灵敏度LC-UV-MS的方法被证明提供了有益于RT-qPCR试剂认证的深层次信息。
优势
- 将二异丙基乙胺离子对应用于PCR试剂分析,采用色谱分离度和灵敏度高的UV-MS BioAccord系统实现在线MS检测和精确质量数峰鉴定
- 将ACQUITY Premier色谱系统应用于PCR试剂表征
简介
根据世界卫生组织的数据,SARS-CoV-2大流行疫情目前已经造成超过400万人死亡(2021年9月)1。 全世界已接种超过50亿剂疫苗1,但遗憾的是,现有疫苗的有效性和接受率差异很大。病毒在人与人之间的长时间传播以及随后的突变积累,增加了病原体基因组发生显著改变的概率,并可能增加其传播性或传染性2。迄今为止,在为减缓SARS-CoV-2传播所做的努力中,利用逆转录酶定量聚合酶链反应(RT-qPCR)进行常规检测是一个基本要素3,4。美国食品药品监督管理局(FDA)已给予疾病控制和预防中心(CDC) 2019-nCoV RT-qPCR试剂盒的紧急使用授权。该检测基于对核衣壳RNA基因(N1和N2)中两个不同位置的检测,方法检测限为约65个病毒拷贝/µL4。重要的是,方法灵敏度可能取决于引物/探针结合区内的核苷酸取代3、RNA提取试剂盒试剂质量4以及寡核苷酸合成错误5。因此,用于研究和确认PCR检测试剂质量的分析方法非常重要。本文使用与飞行时间(Tof)质谱联用的离子对反相色谱法对RT-qPCR引物/探针试剂盒进行高纯度评估。
实验
样品信息
化学合成的寡核苷酸购自供应商,将其直接用于SARS-CoV-2检测以及仅对原料脱盐。这些样品用无核酸酶的10 mM Tris、0.1 mM EDTA、pH 7.5缓冲液制成10 pmol/μL,然后转移至300 μL聚丙烯自动进样器瓶(部件号:186002639)中。
液相色谱条件
|
液相色谱系统: |
ACQUITY UPLC I-Class系统 |
|
检测器: |
ACQUITY UPLC TUV检测器 |
|
波长: |
260 nm |
|
色谱柱: |
ACQUITY Premier BEH C18, 2.1 × 50 mm, 130 Å, 1.7 µm |
|
柱温: |
60 °C |
|
样品温度: |
4 °C |
|
进样: |
1 μL (10 pmol) |
|
流速: |
0.3 mL/min |
|
流动相A: |
含0.1% N,N-二异丙基乙胺(DIPEA)(作为IP试剂)和1% 1,1,1,3,3,3-六氟异丙醇(HFIP)的去离子水 |
|
流动相B: |
含0.0375% DIPEA和0.75% HFIP的65:35乙腈/水 |
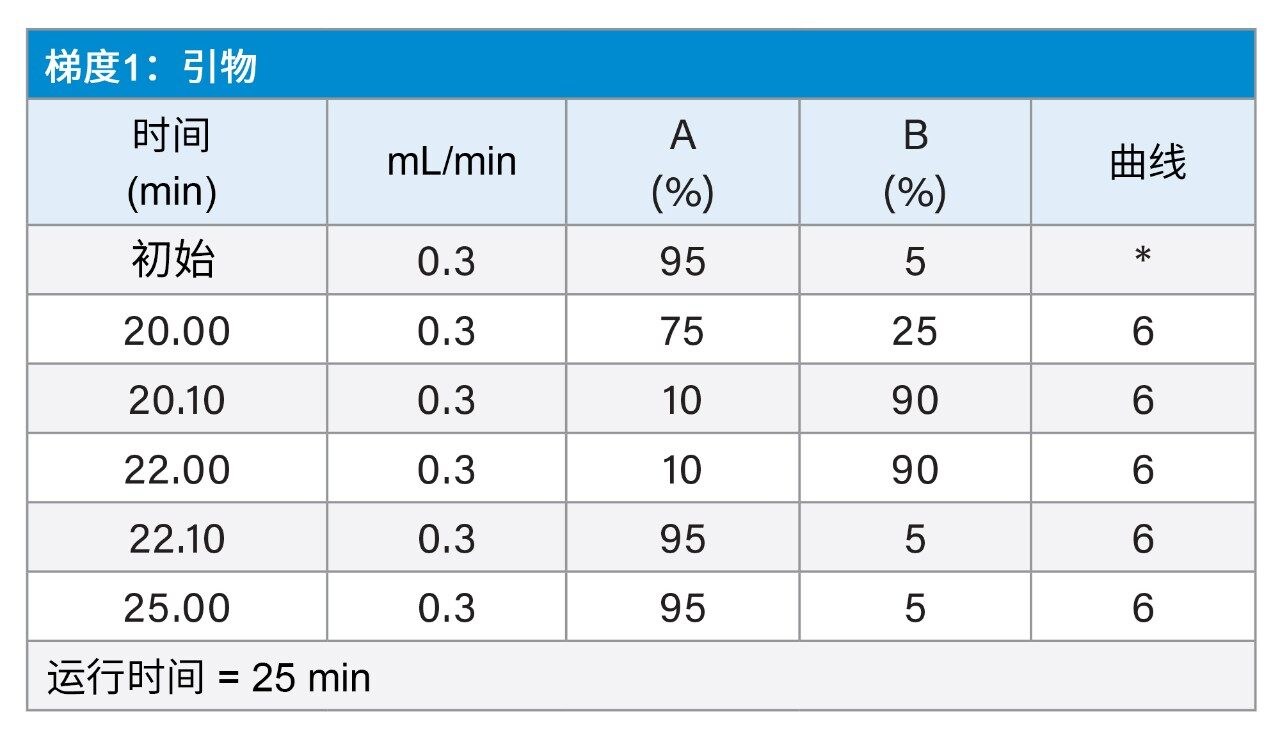
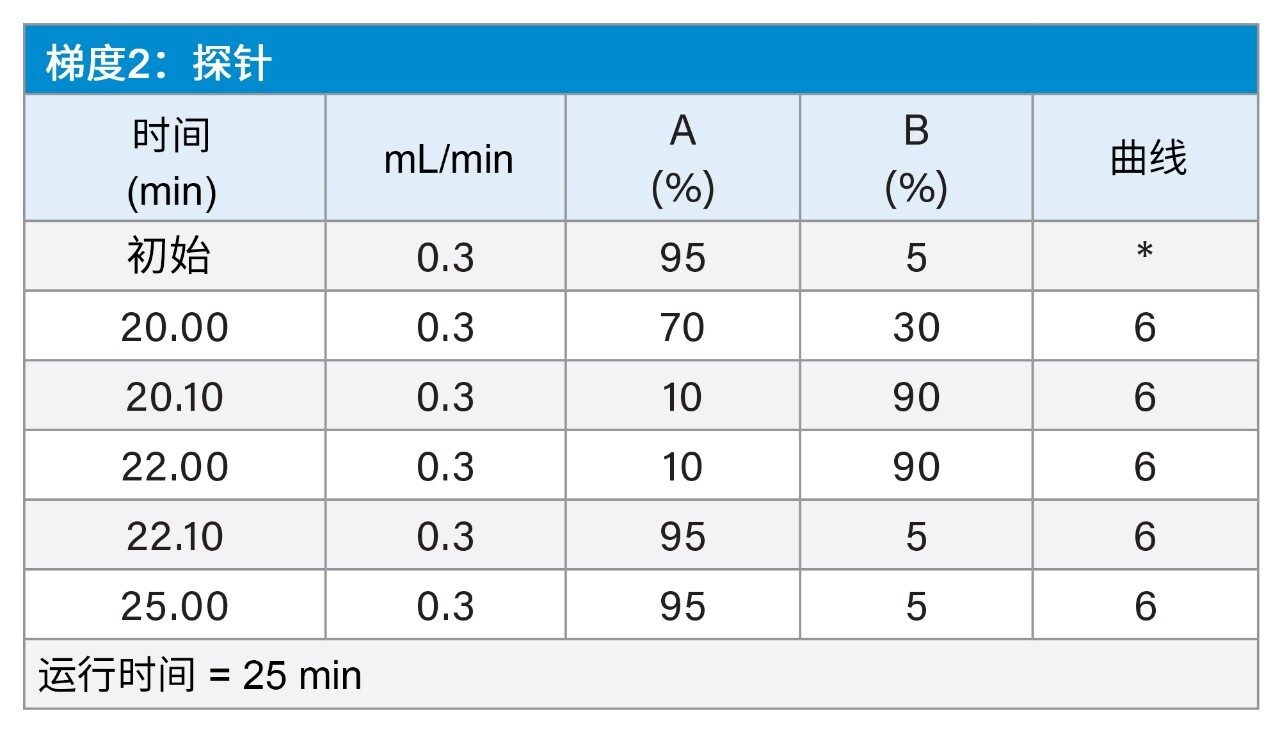
质谱条件
|
质谱系统: |
BioAccord |
|
检测器: |
ACQUITY RDa检测器 |
|
模式: |
全扫描 |
|
极性: |
负 |
|
锥孔电压: |
40 V |
|
质量范围: |
高(400~5000 m/z) |
|
扫描速率: |
2 Hz |
|
毛细管电压: |
0.80 kV |
|
脱溶剂气温度: |
400 °C |
结果与讨论
在离子对反相色谱(IP-RPLC)中,流动相包含离子对试剂(通常为烷基胺),可吸附在C18固定相6-8上,从而引入了一种类似混合模式的保留机制,其中寡核苷酸电荷和荧光团的疏水性均参与IP-RPLC保留6-8。本应用中所用的流动相与光学UV检测和负离子模式质谱均兼容8,9。
本研究采用一种MS兼容的流动相体系,该体系使用二异丙基乙胺(DIPEA)作为IP试剂并且额外添加1,1,1-六氟异丙醇(HFIP)。HFIP已被证明能够通过改善带负电荷的分析物在C18固定相中的保留以及电喷雾电离过程中的电荷集中过程来提高IP-RPLC-MS分析的灵敏度6。为了将这种方法付诸实践,利用装配ACQUITY UPLC TUV检测器的ACQUITY UPLC I-Class,用包含水和乙腈的流动相开发梯度。寡核苷酸样品以10 pmol的量注入ACQUITY Premier C18 (2.1 × 50 mm, 130 Å, 1.7 µm)寡核苷酸分析专用柱中。在UV检测之后,利用台式BioAccord LC-MS系统的ACQUITY RDa检测器,通过负离子模式质谱验证洗脱的寡核苷酸PCR试剂特性。
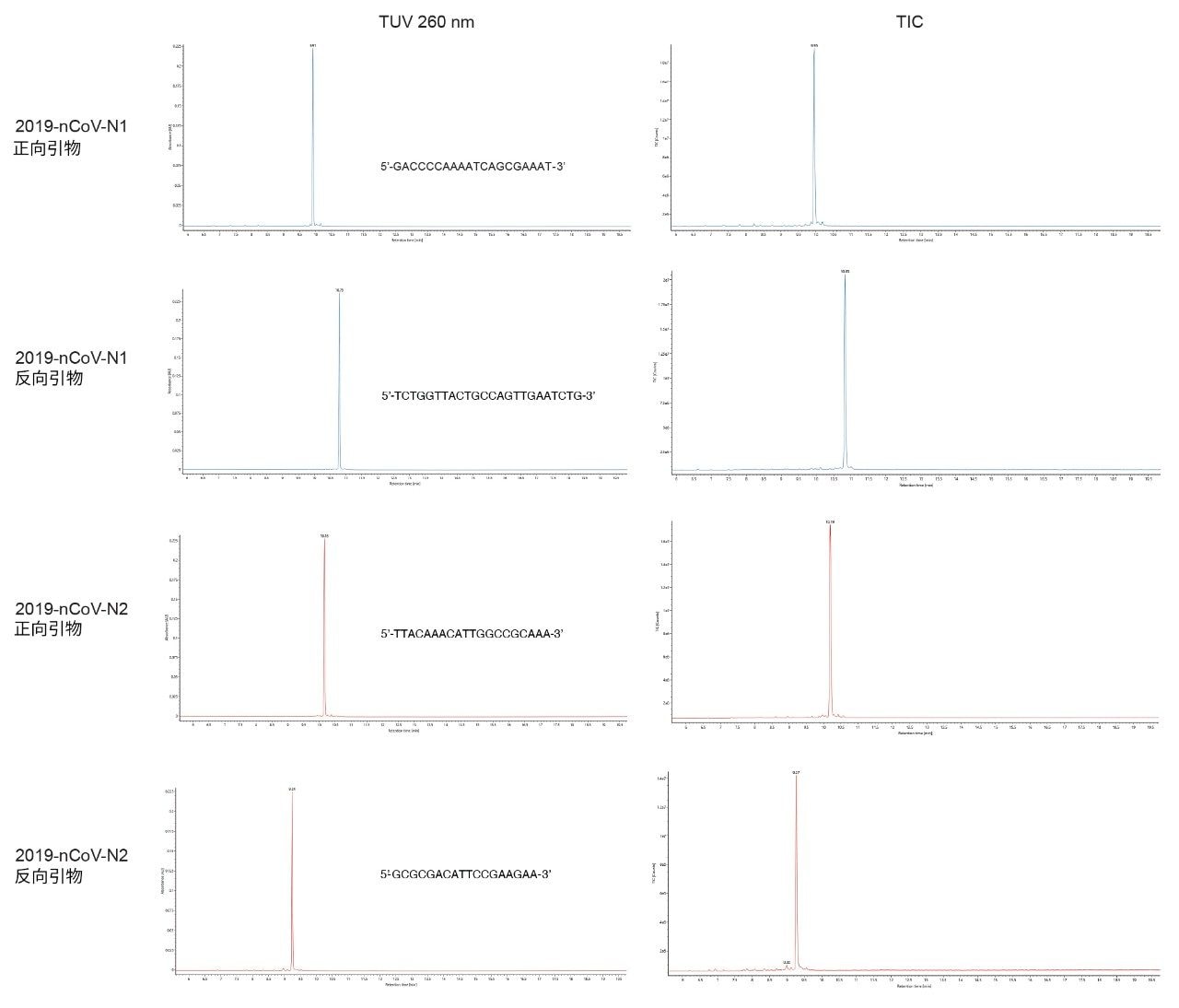
使用流动相B在20 min内从5%增加到25%的梯度,来自CDC RT-qPCR试剂盒的2019 SARS-CoV-2核衣壳N1和N2引物(正向和反向序列)在11 min内从色谱柱上洗脱。在UV和MS色谱图中,均观察到尖锐、对称的峰(图1)以及可重现的保留时间(RT)。MassPREP寡核苷酸标准品(部件号:186004135)在类似的条件下运行,相隔4个月得到的保留时间重现性在0.1 min以内。对于N1和N2引物,正向和反向寡核苷酸之间的RT差异约为1 min。
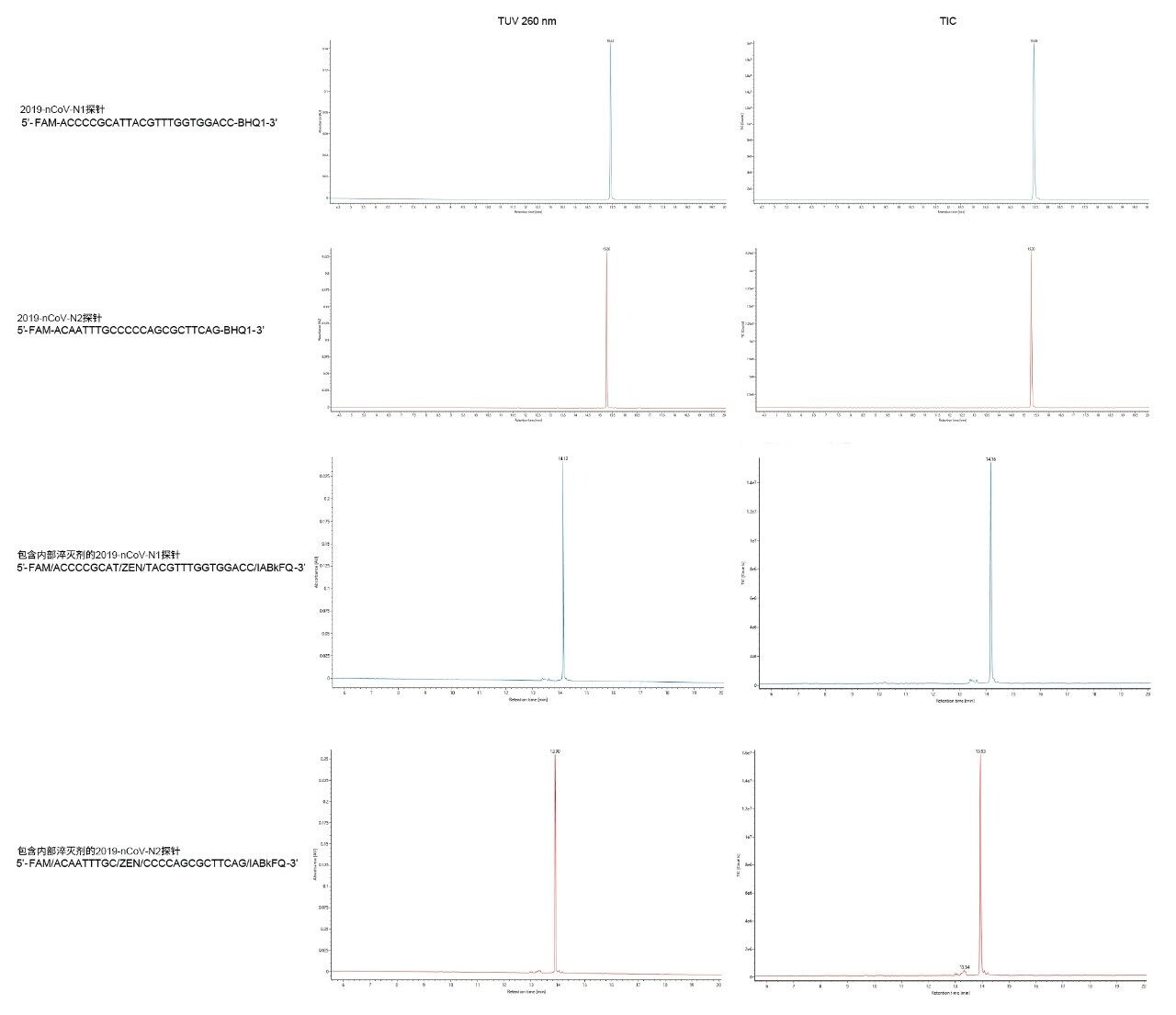
CDC RT-qPCR分析中使用的探针在寡核苷酸序列的5'端用荧光团标记,在3'端用淬灭剂标记。qPCR分析的灵敏度高度依赖于荧光团的正确合成及其通过连接子连接至正确的寡核苷酸序列8。 N1和N2探针也获得了尖锐的峰形(图2)。将梯度修改为流动相B增加到30%,以更好地确保洗脱保留性更强的探针。最终洗脱强度的这一变化导致探针在16 min而不是20 min的保留时间处洗脱。这些结果显示了单个淬灭剂探针的保留行为。除3'淬灭剂以外,还分析了利用内部淬灭剂的探针。利用这些所谓的双淬灭探针实现的分离结果显示在图2的下图中。包含额外的内部淬灭剂的探针在约15 min的较早保留时间处洗脱。
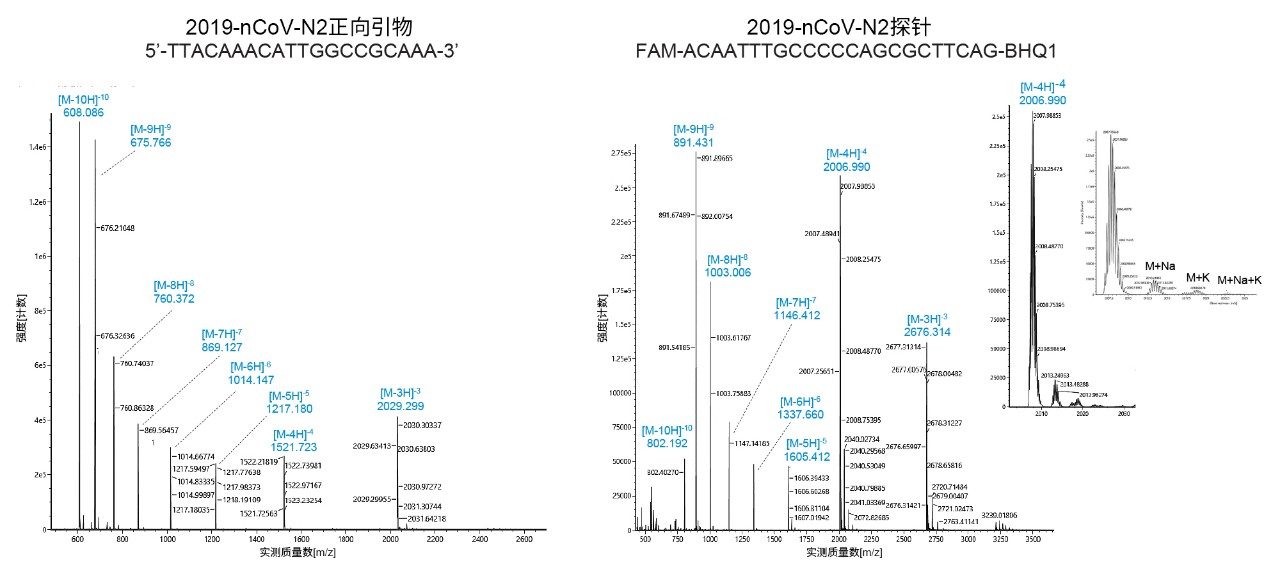
使用BioAccord ACQUITY RDa检测器进行质谱分析使质量数确认成为可能。在260 nm下进行UV检测后,在负离子模式下采集400~5000 m/z之间的质谱图,扫描速率为2 Hz。锥孔电压和毛细管电压采用默认值,分别为40 V和0.80 kV;所用的脱溶剂气温度为400 °C。切换阀设置为在1.5 min后切换至MS,并在22 min后切换回废液。获得的N2正向引物和N2探针的示例质谱图如图3所示。引物和探针的质谱图通常显示出高达[M-10H]10-的多电荷物质,可以对RT-qPCR试剂盒试剂进行明确的精确质量数测定(表2)。图3右侧的插图对应于N2探针的[M-4H]4-离子;在该谱图上观察到的钠和钾加合物不到基峰信号的10%。使用EPFL在线计算器,基于寡核苷酸序列确定引物的元素组成和单同位素质量数10。利用HRMS详细分析样品,单同位素峰的精确质量数分析产生的质量数误差在0.7~7 ppm之间。接下来将BioAccord应用于相应的质量数确认测量。在实践中,此处重点关注单同位素峰的手动解析。但是,UNIFI/waters_connect软件可与MaxEnt1或BayesSpray去卷积算法配合使用,以提供零电荷质谱图。这种数据分析方法对于质量数分析和质量数确认同样具有吸引力11,12。有关PCR探针的分子细节为专有信息;我们使用内部Tof HRMS仪器基于精确质量数测量确定了最可能的分子式,然后使用BioAccord LC-MS系统进行质量数确认。所有质量数测量结果均列于表1中。
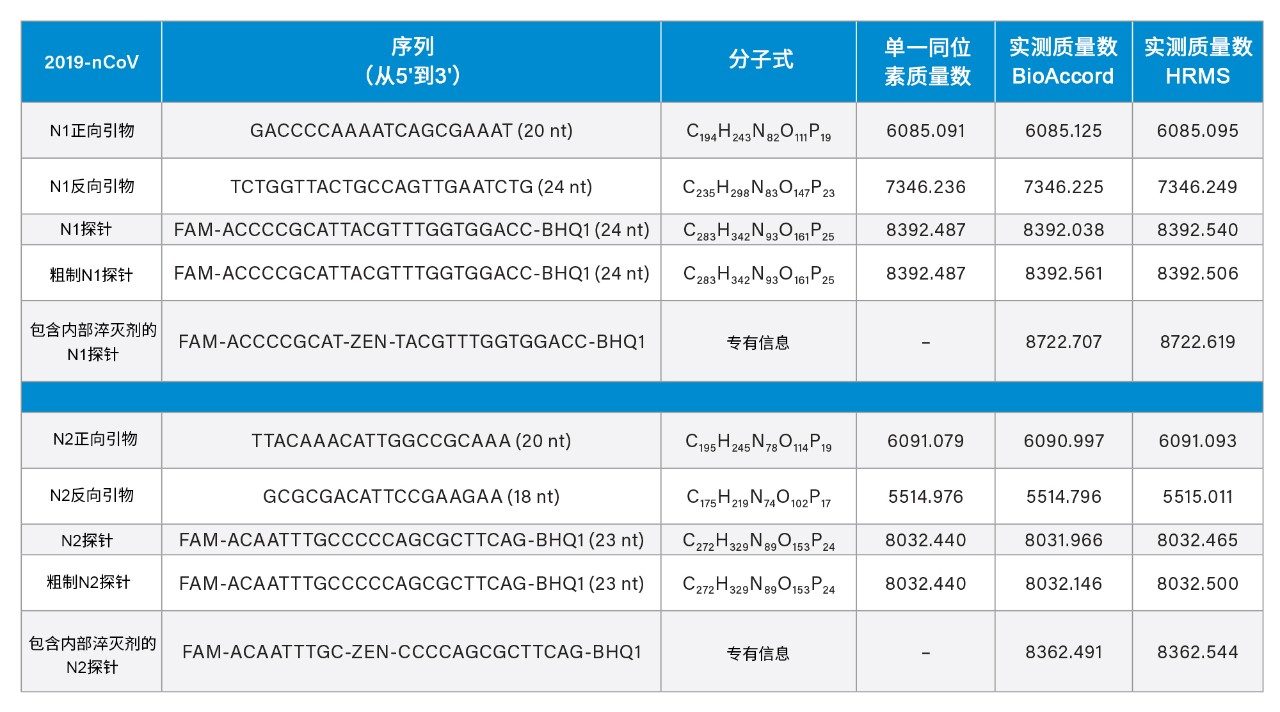
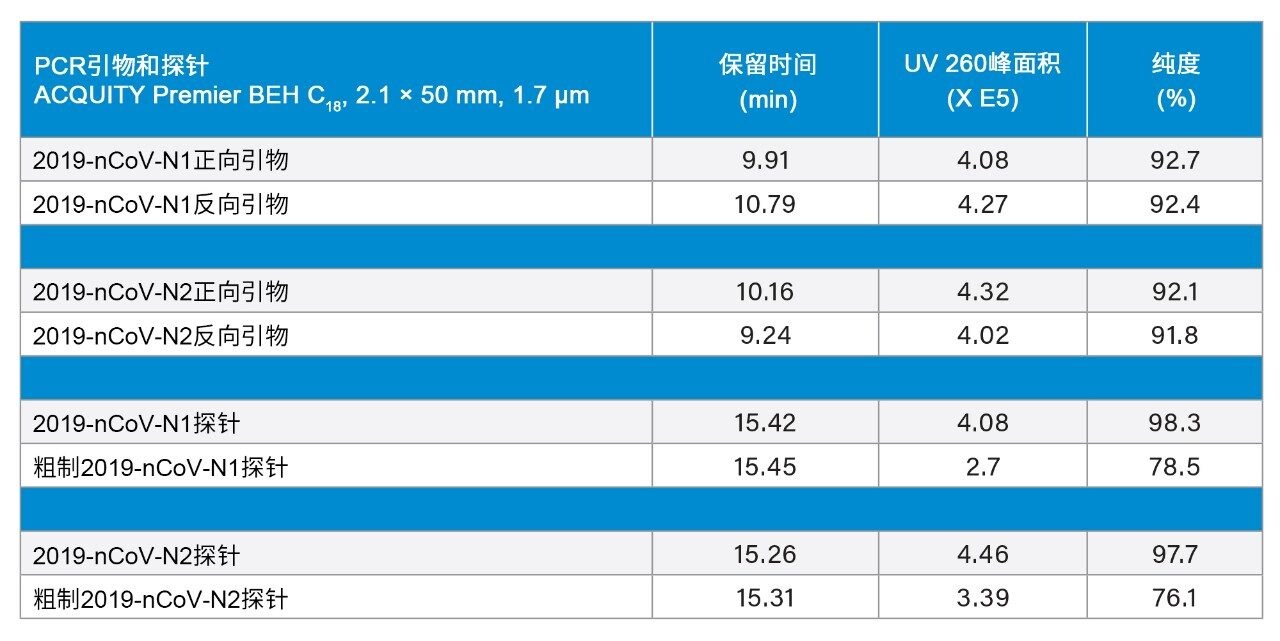
除精确质量数测量以外,还将IP-RPLC与UV和MS结合使用,以评估试剂以及仅在化学合成后脱盐的未纯化原料的纯度。因此,使用采集的UV色谱图对色谱峰进行积分。图4显示了一幅积分色谱图,目标是计算粗制N2探针的百分比纯度。使用UNIFI进行自动峰积分。保留时间、色谱峰面积和纯度列于表2中。结果发现,经确认用于CDC分析的引物纯度为92%,相应的原料纯度为约90%。经确认用于CDC分析的探针具有高纯度(约98%),但发现粗制(仅脱盐)对应物的纯度仅约76%。
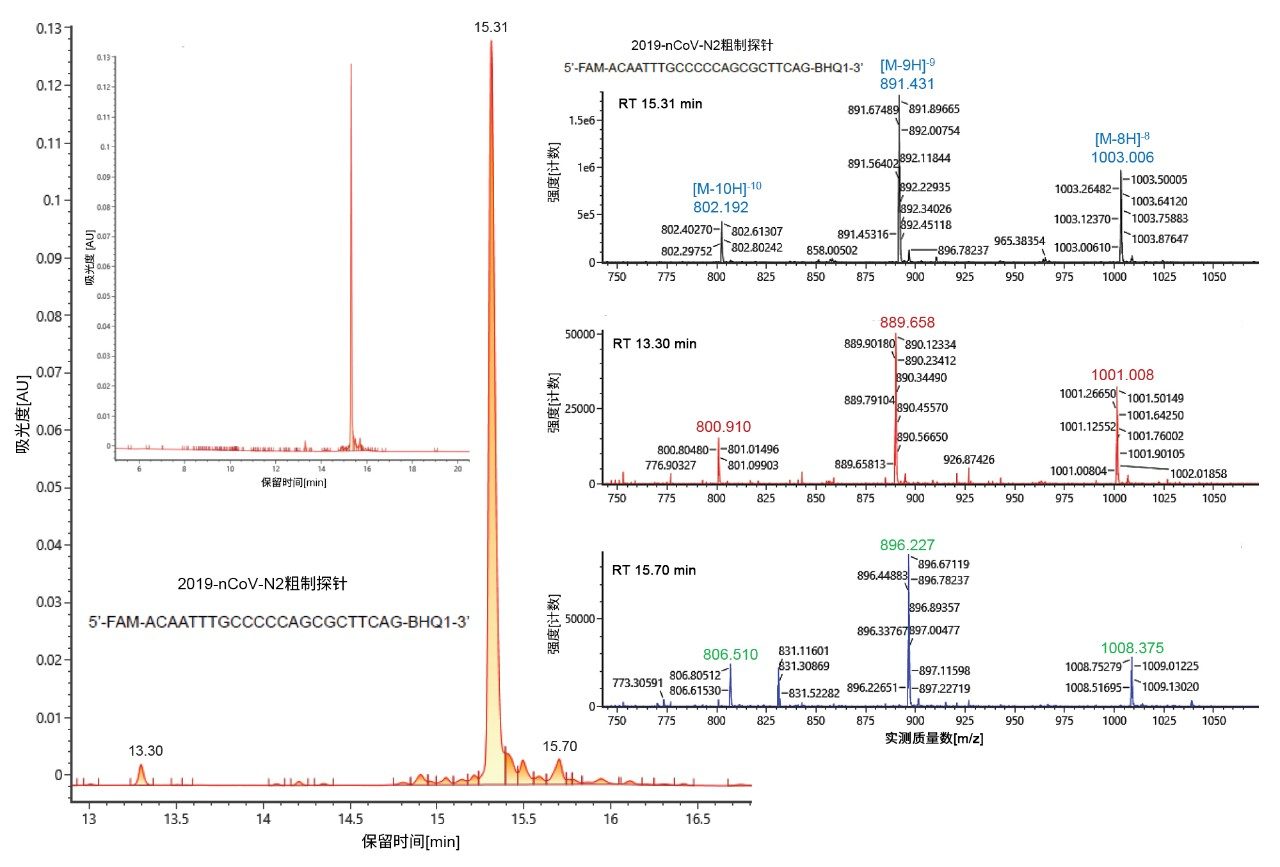
我们更详细地考察了粗制探针内的杂质。除15.31 min处的主色谱峰以外,粗制N2探针的UV迹线在13.30 min和15.70 min处分别表现出一个小峰(图4)。获得的这三个峰[15.31 min(顶部)、13.30 min(中间)和15.70 min(底部)]的累加质谱图如图4的右图所示。与主色谱峰相比,13.30 min处的峰对应于质量数偏移为-16 Da的物质,表示丢失一个氧的探针结构;15.70 min处的晚洗脱峰为推定的杂质,对应于42.9 Da的正质量数偏移。总之,表2所列以及图4所示的数据说明了使用ACQUITY UPLC TUV检测器和BioAccord RDa检测器进行PCR试剂纯度评估的连续检测能力。
结论
本应用纪要介绍了使用优化的IP-RPLC和符合法规要求的台式Tof仪器对SARS-CoV-2 CDC分析中使用的PCR检测试剂进行的质量评估。借助这种灵敏的方法,能够从10 pmol的核衣壳引物N1和N2(正向和反向序列)以及N1和N2探针中收集大量纯度信息。所有PCR试剂均产生了尖锐、对称的峰。在260 nm下进行UV检测后,采集负离子模式质谱图,可轻松观察到多电荷物质(高达[M-10H]10-)。钠和钾加合物不到基峰信号的10%,使我们能够明确测定确定RT-qPCR试剂盒试剂的分子量。使用UV与MS检测相结合的方法,评估试剂以及未纯化原料的纯度。在这些纯度测量示例中,使用UNIFI对UV色谱图进行自动峰积分。此外,本应用纪要中所述的分析方法能够对杂质进行更详细的考察,如针对单一淬灭剂N2探针的未纯化合成粗产物的研究所示。总之,这种结合高分离度、高灵敏度LC-UV-MS的方法被证明提供了有益于RT-qPCR试剂认证的深层次信息。
参考资料
- WHO Coronavirus (COVID-19) Dashboard [Internet].[cited 2021 Sept 02] https://covid19.who.int/.
- Akkiz, H., Implications of the Novel Mutations in the SARS-CoV-2 Genome for Transmission, Disease Severity, and the Vaccine Development.Front.Med.2021, 8, 10.
- Vogels, C. B. F.; Brito, A. F.; Wyllie, A. L.; Fauver, J. R.; Ott, I. M.; Kalinich, C. C.; Petrone, M. E.; Casanovas-Massana, A.; Catherine Muenker, M.; Moore, A. J.; Klein, J.; Lu, P.; Lu-Culligan, A.; Jiang, X.; Kim, D. J.; Kudo, E.; Mao, T.; Moriyama, M.; Oh, J. E.; Park, A.; Silva, J.; Song, E.; Takahashi, T.; Taura, M.; Tokuyama, M.; Venkataraman, A.; Weizman, O.-E.; Wong, P.; Yang, Y.; Cheemarla, N. R.; White, E. B.; Lapidus, S.; Earnest, R.; Geng, B.; Vijayakumar, P.; Odio, C.; Fournier, J.; Bermejo, S.; Farhadian, S.; Dela Cruz, C. S.; Iwasaki, A.; Ko, A. I.; Landry, M. L.; Foxman, E. F.; Grubaugh, N. D., Analytical Sensitivity and Efficiency Comparisons of Sars-Cov-2 RT–qPCR Primer–Probe Sets.Nature Microbiology 2020, 5 (10), 1299–1305.
- Pearson, J. D.; Trcka, D.; Lu, S. Y.; Hyduk, S. J.; Jen, M.; Aynaud, M. M.; Hernandez, J. J.; Peidis, P.; Barrios-Rodiles, M.; Chan, K.; Woodgett, J.; Mazzulli, T.; Attisano, L.; Pelletier, L.; Cybulsky, M. I.; Wrana, J. L.; Bremner, R., Comparison of SARS-CoV-2 Indirect and Direct RT-qPCR Detection Methods.Virol.J. 2021, 18 (1), 12.
- Filges, S.; Mouhanna, P.; Ståhlberg, A., Digital Quantification of Chemical Oligonucleotide Synthesis Errors.Clinical Chemistry 2021.
- Guo, L.; Worth, A. J.; Mesaros, C.; Snyder, N. W.; Glickson, J. D.; Blair, I. A., Diisopropylethylamine/Hexafluoroisopropanol-Mediated Ion-Pairing Ultra-High-Performance Liquid Chromatography/Mass Spectrometry for Phosphate and Carboxylate Metabolite Analysis: Utility for Studying Cellular Metabolism.Rapid Commun Mass Spectrom 2016, 30 (16), 1835–1845.
- Birdsall, R. E.; Gilar, M.; Shion, H.; Yu, Y. Q.; Chen, W., Reduction of Metal Adducts in Oligonucleotide Mass Spectra in Ion-Pair Reversed-Phase Chromatography/Mass Spectrometry Analysis.Rapid Commun Mass Spectrom 2016, 30 (14), 1667-1679.
- Fountain, K.; Gilar, M.; Budman, Y.; Gebler, J., Purification of Dye-Labeled Oligonucleotides by Ion-Pair Reversed-Phase High-Performance Liquid Chromatography.Journal of Chromatography.B, Analytical Technologies in the Biomedical and Life Sciences 2003, 783, 61–72.
- Jiang, T.; Yu, N.; Kim, J.; Murgo, J.-R.; Kissai, M.; Ravichandran, K.; Miracco, E. J.; Presnyak, V.; Hua, S., Oligonucleotide Sequence Mapping of Large Therapeutic mRNAs via Parallel Ribonuclease Digestions and LC-MS/MS.Anal.Chem.2019, 91 (13), 8500–8506.
- Ortiz, D.; Gasilova, N.; Sepulveda, F.; Patiny, L.; Dyson, P. J.; Menin, L., Aom2S: A New Web-Based Application for DNA/RNA Tandem Mass Spectrometry Data Interpretation.Rapid Communications in Mass Spectrometry 2020, 34 (23), e8927.
720007429ZH,2021年12月