仅供研究使用,不适用于诊断。
高度糖基化的SARS-CoV-2刺突蛋白与COVID-19的传染性有关,因此,研究人员在表征衣壳刺突蛋白的糖基化模式上开展大量工作,以便为疫苗和免疫药物的开发提供指导。本研究利用新型SELECT SERIES Cyclic IMS的新颖配置,对在刺突蛋白的弗林蛋白酶切位点附近发现的O-联糖肽进行糖基结构和键表征;弗林蛋白酶切会使刺突蛋白的融合序列暴露,导致S1亚基与S2亚基分离,这一过程是病毒进入细胞的必要条件。另外,还鉴定出同时具有α2-3和α2-6唾液酸键的扩展核心1和核心2型结构;研究表明,糖基化会改变底物识别机制,核心2和扩展核心1结构的比率可能影响新型冠状病毒的毒力。
SELECT SERIES Cyclic IMS的新颖设计能够对SARS-CoV-2刺突蛋白中低水平O-联糖肽的糖基结构进行位点特异性、键特异性表征。利用捕集阱碎裂模式从O-糖区域释放出氧鎓离子碎片,并用高分辨率离子淌度技术分离和分析碎片离子。
近期爆发的COVID-19大流行疫情由SARS-CoV-2冠状病毒引起,已导致数百万人患病,迄今为止已经造成全球200万人死亡1。由于是一种新病毒,科学界一直致力于快速表征和编译其生物物理特征,希望能够帮助开发有效的疫苗和免疫药物。SARS-CoV-2病毒颗粒通过用突起的跨膜刺突蛋白与宿主细胞ACE2(血管紧张素转化酶2)受体结合而进入细胞2。 SARS-CoV-2冠状病毒刺突蛋白为同源三聚体I类融合蛋白,由两个高度糖基化的亚基S1和S2组成3,4。 之前对已知病毒病原体(包括流感)的研究表明,外壳的糖基成分在免疫逃逸中发挥至关重要的作用,特别是通过免疫识别位点的空间位阻起作用5-7。
SARS-CoV-2病原体的表征工作包括对冠状病毒刺突蛋白糖基化的多项综合研究。迄今为止,这些研究已经表明,在22个可用的N-糖基化位点中有14个共有占用位点,其余7个位点的占用则存在冲突8-16。 此外,Shajahan等人的研究证明,S1结构域上有三个O-联糖基化位点,其中一个已由Sanda等人确证10,11。在SARS-CoV-刺突蛋白的序列分析中,Andersen和同事预测了S1和S2亚基侧翼连接区中的弗林蛋白酶切位点,该位点对蛋白质激活至关重要2。他们进一步预测,这个含有弗林蛋白酶切位点的区域包含至多三个额外的O-联糖基化位点,推测这些位点的功能与传染性和传播性有关2。 众所周知,O-糖的表征非常困难,原因有:缺乏共有序列、糖型之间的异质性高、相对丰度低。因此,表征这类分析物需要采用高性能色谱与高分辨率、高灵敏度质谱。先前使用沃特世SYNAPT平台进行的研究表明,该平台能够解析游离寡糖异构体的支链和线性糖基结构,并从游离寡糖标准品中分离出α2-3和α2-6键合异构体12-16。SELECT SERIES Cyclic IMS的MS/MS碎裂为多元弗林蛋白酶切位点之前沿连接区的O-联糖基化提供了清晰的证据,并利用环形离子淌度技术对O-糖结构进行位点特异性分离和表征。此项独特实验提供的可扩展分辨率表明存在核心1、扩展核心1和核心2结构混合物,为NeuAcα2-3Galβ1-3GalNAc和NeuAcα2-6Galβ1-4GlcNAc提供了分辨率,这两种异构体具有非常相似的碰撞截面。
重组SARS-CoV-2刺突蛋白在HEK 293细胞中表达,购自Acrobiosystems。用DTT还原刺突蛋白中的二硫键,并用碘乙酰胺进行处理,使半胱氨酸残基烷基化,然后用PNGaseF和胰蛋白酶进行酶解。
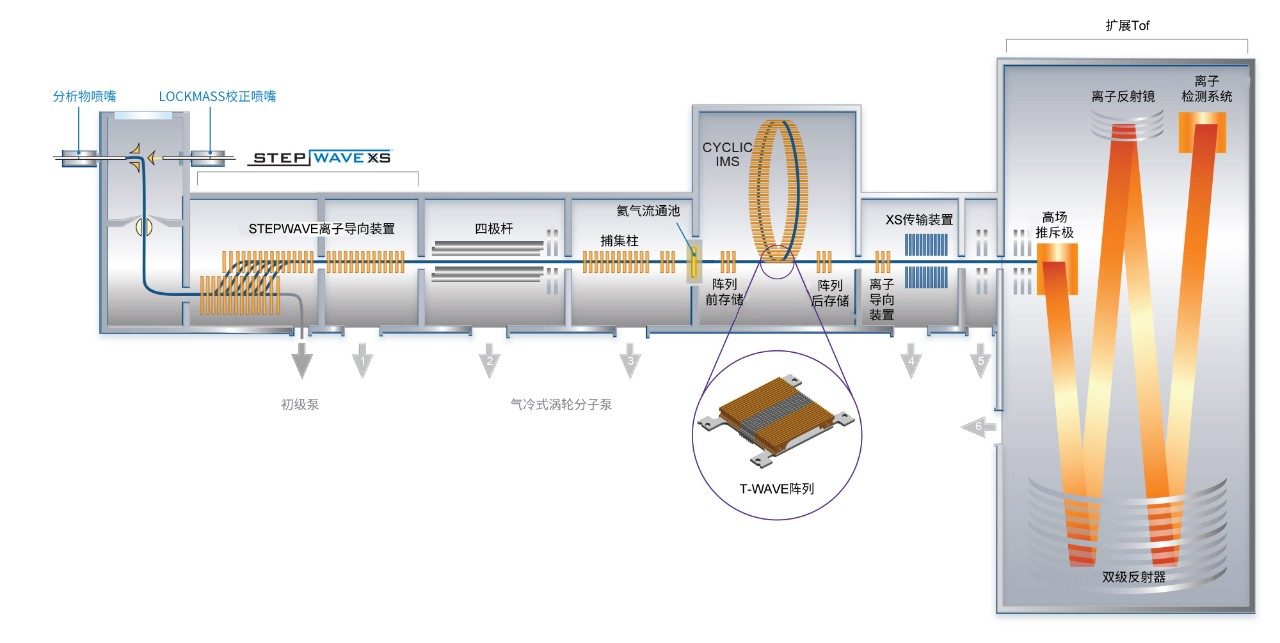
表1、2和3列出了色谱和质谱分析的一般条件。使用五圈环形装置的Cyclic IMS方法之前已经用源自血红素结合蛋白的O-联糖肽针对三糖和二糖碎片进行了优化。源自SARS-CoV-2刺突蛋白的O-糖肽经四极杆分离,然后在SELECT SERIES Cyclic IMS的捕集区中发生碰撞活化(如图1所示)。使用环形离子淌度池分离糖肽的碎片。进行方法优化,使三糖碎片在喷射和检测之前通过装置五次。
|
液相色谱系统: |
ACQUITY UPLC M-Class |
|
检测: |
SELECT SERIES Cyclic IMS |
|
样品瓶: |
QuanRecovery |
|
色谱柱: |
nanoEase M/Z HSS T3 100 Å, 1.8 µm (75 µm × 15 cm)(部件号:186008816) nanoEase M/Z Symmetry C18 100 Å, 5 µm (180 µm × 20 mm)捕集阱(部件号:186008821) |
|
柱温: |
60 °C |
|
样品温度: |
8 °C |
|
进样体积: |
1–5 μL |
|
流速: |
500 nL/min |
|
流动相A: |
H2O,0.1% FA,1 ppm柠檬酸 |
|
流动相B: |
ACN,0.1% FA,1 ppm柠檬酸 |

|
MS系统: |
Cyclic IMS |
|
电离模式: |
ESI+ |
|
采集范围: |
m/z 50~2000 |
|
毛细管电压: |
2.8 kV |
|
碰撞能量: |
30 V–70 V |
|
锥孔电压: |
20 V |
|
圈数: |
5 |
|
Racetrack TW高度: |
22 V |
|
Racetrack TW速度: |
375 m/s |
|
IMS顺序: |
进样、分离、喷射和采集 |
|
IMS运行周期: |
100 ms |
|
色谱软件: |
MassLynx SCN1016 Release 3 |
|
MS软件: |
用于Cyclic IMS Release 4的沃特世嵌入式分析仪平台 |
|
信息学软件: |
DriftScope 2.9,针对Cyclic IMS进行了改进 |
对观察到的胰蛋白酶解肽AGC(carbamidomethyl)LIGAEHVDNSYEC(carbamidomethyl)DIPIGAGIC(carbamidomethyl)ASYQTQTNSPR(标记为T56)的O-联糖型进行靶向离子淌度MS/MS实验,在捕集区中进行碎裂,生成肽段和氧鎓离子碎片,然后进入离子淌度池。通过碰撞活化释放后,利用环形离子淌度(cIM)仪器的可扩展分辨率分离糖基异构体。之前针对三糖碎片1次和5次通过循环装置开发出两种方法,利用这两种方法分离T56-HexNAc(1)-Hex(1)-NeuAc(1)、T56-HexNAc(1)-Hex(1)-NeuAc(2)、T56-HexNAc(2)-Hex(1)-NeuAc(1)、T56-HexNAc(2)-Hex(2)-NeuAc(1)和T56-HexNAc(2)-Hex(2)-NeuAc(2)的HexNAc-Hex和HexNAc-Hex-NeuAc氧鎓离子碎片的同分异构体。这些肽在表1中以糖肽A-D列出。
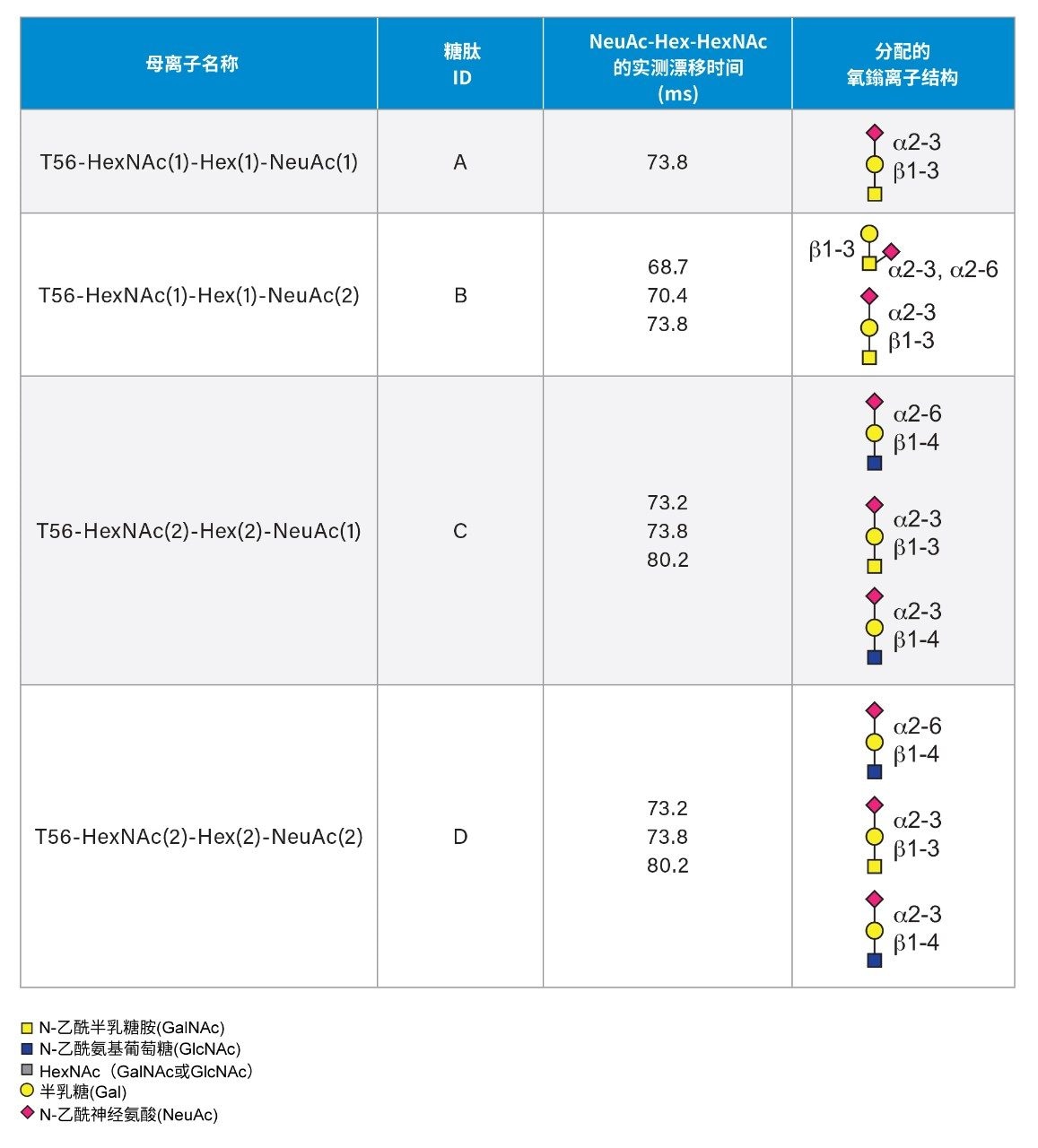
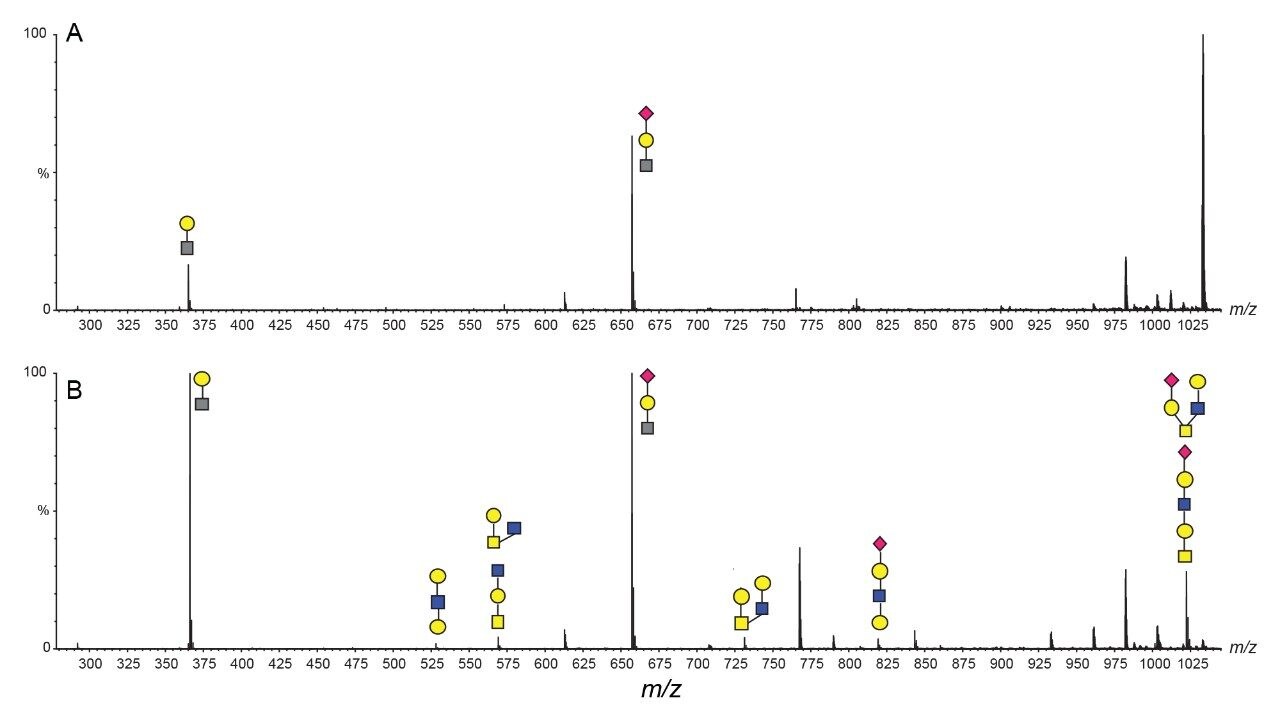
利用DriftScope 2.9对产生三电荷未修饰T56肽段碎片的色谱峰进行积分,提取离子淌度信息。一般来说,每种糖型可观察到2~4个色谱峰,表明某些具有异质性的异构体可通过反相色谱法分离。T56糖肽的碰撞诱导碎裂通常会产生肽段、氧鎓与肽+糖基碎片的混合物。图2展示了A)糖肽A和B)糖肽C的MS/MS谱图,证明较大的氧鎓离子碎片具有高产率,对于糖肽C而言尤其如此。m/z 1022处HexNAc(2)-Hex(2)-NeuAc(1)氧鎓离子碎片的观察结果确认存在单个五糖糖型,而不是单独的三糖和二糖成分各自位于两个不同位点。m/z 528和819处的氧鎓离子确认存在扩展核心1结构。
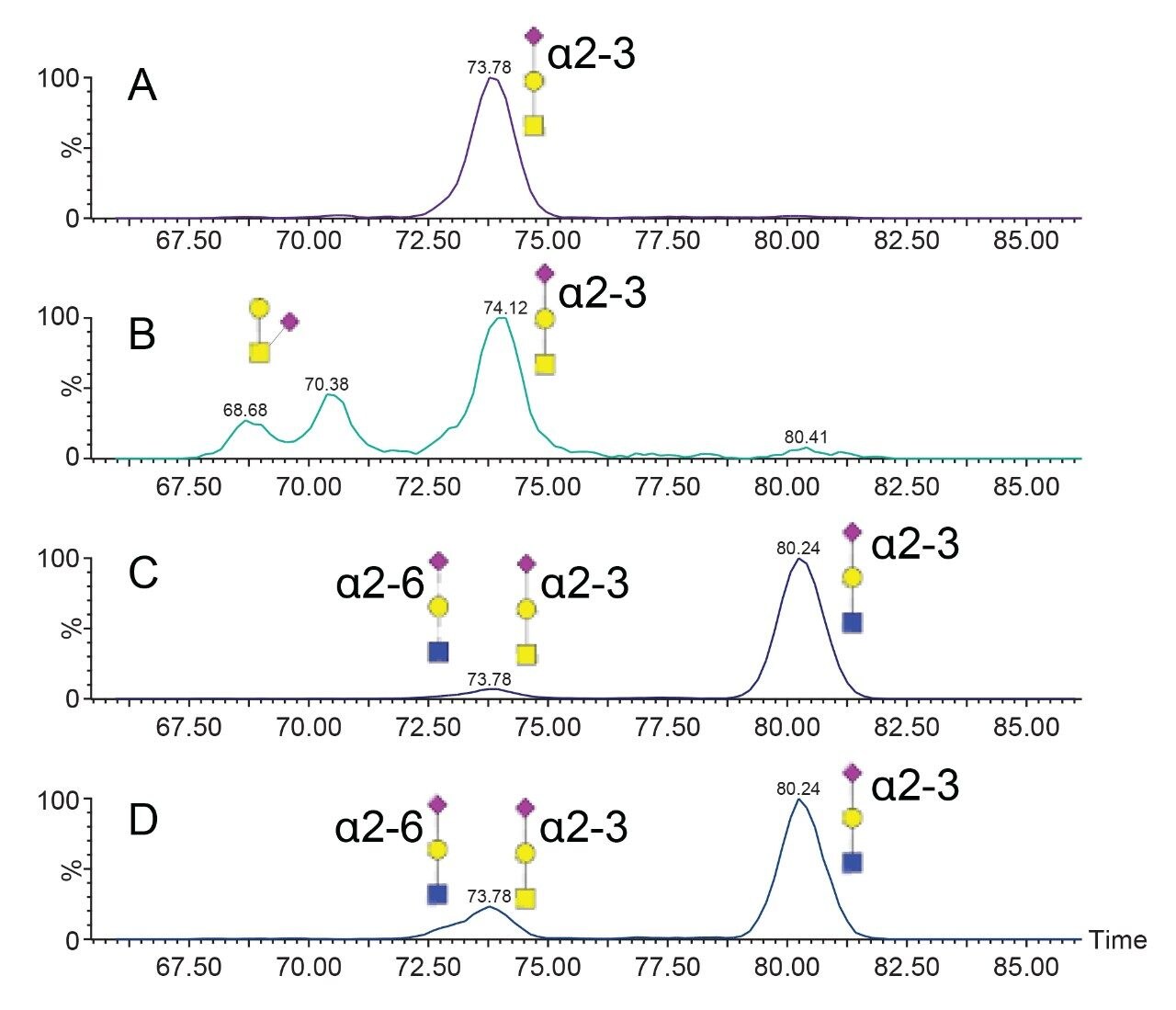
对HexNAc-Hex-NeuAc氧鎓离子实施单圈和多圈(5圈)离子淌度实验。使用变化的碰撞能量分析糖肽C所得的到达时间分布如图3 (B-D)所示。使用单圈环形离子淌度时,可明显看到两个离子淌度群;使用五圈离子淌度时,更紧凑的物质进一步分离为两种部分分离的异构体。使用Major Mix IMS校准标样作为参比,计算单圈得到的碰撞截面。单圈实验观察到的两个到达时间分布计算出的碰撞截面分别为234.1和245.4 Å2。Guttman等人12之前测得NeuAcα2-6Galβ1-4GlcNAc和NeuAcα2-3Galβ1-4GlcNAc的CCS值分别为236.9 Å2和247.2 Å2。测得的最细长的HexNAc-Hex-NeuAc异构体与NeuAcα2-3Galβ1-4GlcNAc高度吻合。使用五圈环形离子淌度装置时,在234.1 Å2处观察到的单个离子淌度峰分离为两种子结构。先前的研究表明,α2–6唾液酸键比α2–3键更稳定;因此使用变化的碰撞能量进行实验,结果见图3 (B–D)17。对于通过5圈方法可分离的三种最紧凑的异构体,提高碰撞能量能够增加相对丰度。核心1型结构(NeuAcα2-3Galβ1-3GalNAc)是最常见的O-联糖肽结构;因此,5圈分离中更细长且更不稳定的紧凑异构体与NeuAcα2-3Galβ1-3GalNAc 1型核心结构一致。更紧凑且更不稳定的结构可分配给NeuAcα2-6Galβ1-4GlcNAc,其符合核心2结构的预期碎裂结果。
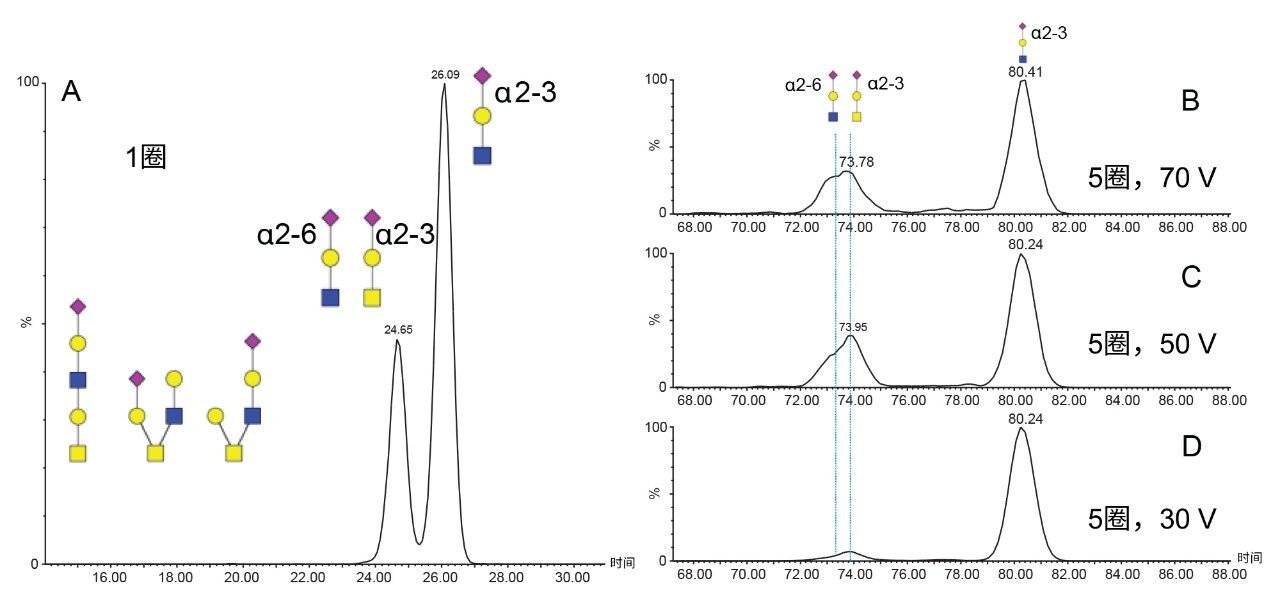
利用五圈环形离子淌度技术分析剩余糖肽中的m/z 657 (HexNAc-Hex-NeuAc)氧鎓离子,结果见图4。拥有两个HexNAc残基(C和D)的糖肽得到类似的离子淌度到达时间分布(ATD),表明存在扩展核心1和核心2结构的混合物,如图3所示。最简单的糖肽具有单个HexNAc、Hex和NeuAc残基(A),在73.8 ms处形成单个离子淌度分布,将其分配给T56-HexNAc(2)-Hex(2)-NeuAc(1)糖肽的NeuAcα2-3Galβ1-3GalNAc,符合已知的糖生物学信息。相比之下,从T56-HexNAc(1)-Hex(1)-NeuAc(2)糖肽中分离出的m/z 657氧鎓离子在68.7 ms和70.4 ms处产生两个额外的离子淌度峰。这些可能是支化结构,在这些结构中,NeuAc残基通过2-3或2-6键与GalNAc连接。在糖肽D中未观察到这些峰,表明核心GalNAc残基(图4B)的唾液酸化对于T56糖肽而言较为罕见,仅存在于简单的核心1结构中。
本研究证明SELECT SERIES Cyclic IMS能够分离SARS-CoV-2刺突蛋白O-糖肽的氧鎓离子碎片,提供有关糖肽中糖结构的位点特异性和键特异性信息。Cyclic IMS仪器拥有高灵敏度、可扩展的离子淌度分辨率和独特的几何结构,可用于低含量O-糖肽的靶向CID-cIM-MS实验,实现高异质性蛋白质的位点特异性结构表征。该策略能够为正在全球开展的SARS-CoV-2病毒表征、理解和靶向作用研究提供更多详细信息。
720007081ZH,2020年11月