
监管机构要求作物保护产品在注册之前必须进行大量有记录且经审查的研究,研究重点应主要针该产品的优点及使用过程中可能存在的风险。生产商必须就产品对人类健康和环境的潜在影响提供相关证据。此类证据的一个方面就是新产品中活性成分的代谢归趋以及潜在代谢物对整个环境的影响或对人类的毒性。因此,准确测定代谢物对于评估产品的暴露风险和进行全面的产品风险评估来说非常重要。本应用纪要介绍了一种利用UPLC以及与UNIFI科学信息系统结合的高分辨率质谱鉴定复杂基质中代谢物的综合性方法,此方法可简化新型农作物保护产品的注册流程。
监管机构(如U.S. EPA)要求作物保护产品在注册之前必须进行大量有记录且经审查的研究,研究重点应主要针该产品的优点及使用过程中可能存在的风险1。 例如,生产商必须就产品对人类健康和环境的潜在影响提供相关证据。此类证据的一个方面就是新产品中活性成分(AI)的代谢归趋以及潜在代谢物对整个环境的影响或对人类的毒性。因此,准确测定代谢物对于评估产品的暴露风险和进行全面的产品风险评估来说非常重要。
我们一般通过高分辨率质谱获取母离子和产物离子数据来进行代谢物鉴定研究,因为该方法获得的结果可提高注册申请的通过率2。 高分辨率质谱技术能够提供全面的信息,而且其配备的智能软件工具具有数据查询功能,能为科学家们提供极大的便利。创新质谱技术利用“MSE”建立了全面的母离子和产物离子数据集,通过单次分析进样即可同时采集母离子和产物离子的全扫描谱图,而无需对母离子进行预选。由于该方法不需要分析物的详细信息,因此简化了数据采集过程3。该方法采用非靶向的方式采集高质量精度质谱数据。因此,完整分子离子和所有碎片离子的元素组成信息均可获得。
在本应用纪要中,研究人员将阿特拉津(一种已有大量科学文献报道过的市售除草剂)及其代谢物加标到土壤浸出液中,作为说明沃特世基于UNIFI的代谢物鉴定应用解决方案工作流程的模型。加标土壤浸出液中阿特拉津及其代谢物的分析采用ACQUITY UPLC I-Class与Xevo G2-XS QTof质谱联用系统。阿特拉津(图1)是一种三嗪类除草剂,广泛用于控制多种作物(包括玉米、甘蔗和高粱)种植过程中的杂草4-8。 使用UNIFI的数据评估工具可轻松将阿特拉津与其代谢物之间的结构关系可视化。
称取10 g土壤,装入50 mL离心管中。向土壤样品中加入5 mL水和10 mL乙腈,然后振摇混合物20 min。完成盐析步骤并对所得的乙腈层进行纯化之后,直接将阿特拉津及其代谢物加标到土壤浸出液中,供分析使用。
|
UPLC系统: |
ACQUITY UPLC I-Class |
|
色谱柱: |
ACQUITY UPLC HSS T3, 1.8 μm, 2.1 x 100 mm |
|
流动相(A): |
10 mM甲酸铵水溶液 |
|
流动相(B): |
乙腈 |
|
流速: |
0.6 mL/min |
|
柱温: |
45 °C |
|
进样体积: |
1 μL |
|
UPLC梯度: |
0 min 1% B,1 min 1% B,5 min 80% B,6 min 90% B,然后返回初始条件 |
|
MS系统: |
Xevo G2-XS QTof |
|
电离模式: |
ESI+ MSE分辨率模式 |
|
毛细管电压: |
1.0 kV |
|
锥孔电压: |
20 V |
|
脱溶剂气温度: |
400 °C |
|
源温度: |
120 °C |
|
锥孔气流速: |
50 L/h |
|
脱溶剂气流速: |
900 L/h |
|
MSE低碰撞能(CE): |
2 eV |
|
MSE高碰撞能(CE)梯度: |
17~45 eV |
|
MS扫描范围: |
50~950 m/z |
|
扫描时间: |
0.15 s |
|
数据处理 |
使用UNIFI软件进行数据采集和数据处理。 |
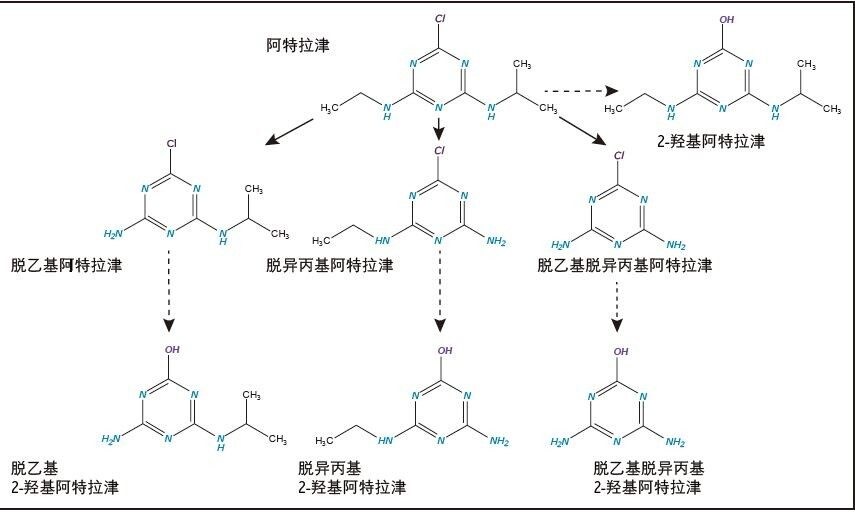
本研究将阿特拉津作为模型化合物,展示了采用UPLC-MSE和UNIFI科学信息系统进行的高效代谢物鉴定。根据文献报道,阿特拉津的代谢生物转化是通过2-羟基化和N-脱烷基化途径实现的(图1)9,10。 还有文献指出,偶联谷胱甘肽也是其代谢生物转化途径之一。因此,我们以已知的代谢产物作为研究的切入点。
UNIFI科学信息系统由一系列工作流程步骤组成,这些步骤专为实现整个数据集的全面可视化而设计,以便用户能够以最少的用户干预轻松获取制定决策所需的信息。仅通过单击鼠标,用户就能自定义数据的可视化过程。该方法有利于用户一致、准确、快速地在一份分析报告中审查完整样品数据。这些步骤是完全可自定义的,而且可专门针对特定分析而创建。
图2展示了专为代谢物鉴定数据审查而设计的工作流程示例。
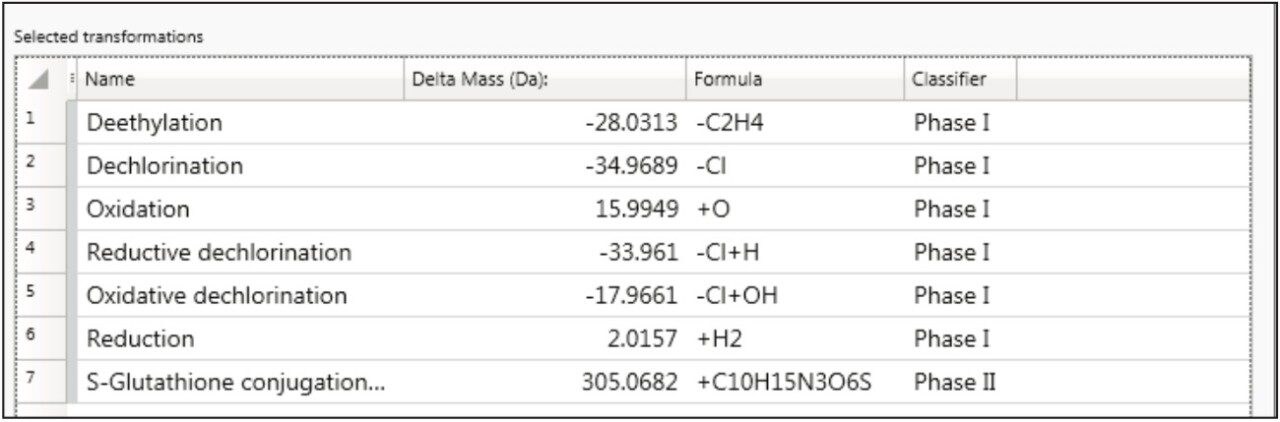
使用UNIFI进行代谢物鉴定时需要可存入科学数据库的目标分子结构(mol文件)。还需要选择可能的转化类型(图3)。
UNIFI科学数据库中有100余种转化类型,并且支持自定义生物转化类型的存储和编辑。UNIFI科学数据库是软件包的一部分,不仅能够存储结构,还能够保留与单独数据库条目相关的信息。用户还可将母体化合物和目标代谢物的数据存储在数据库中,以备日后搜寻和检索。
接下来本文将以阿特拉津代谢数据集为例,介绍图2中的一些工作流程步骤。
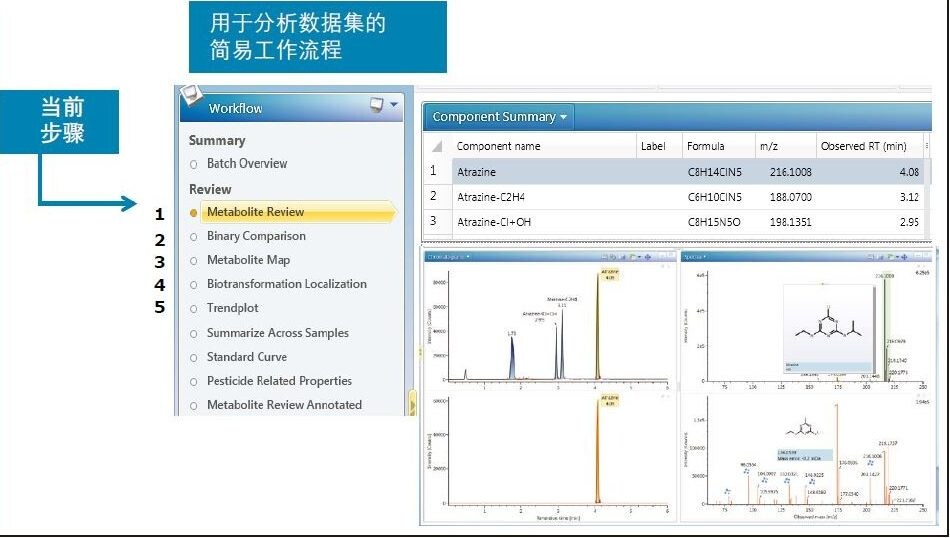
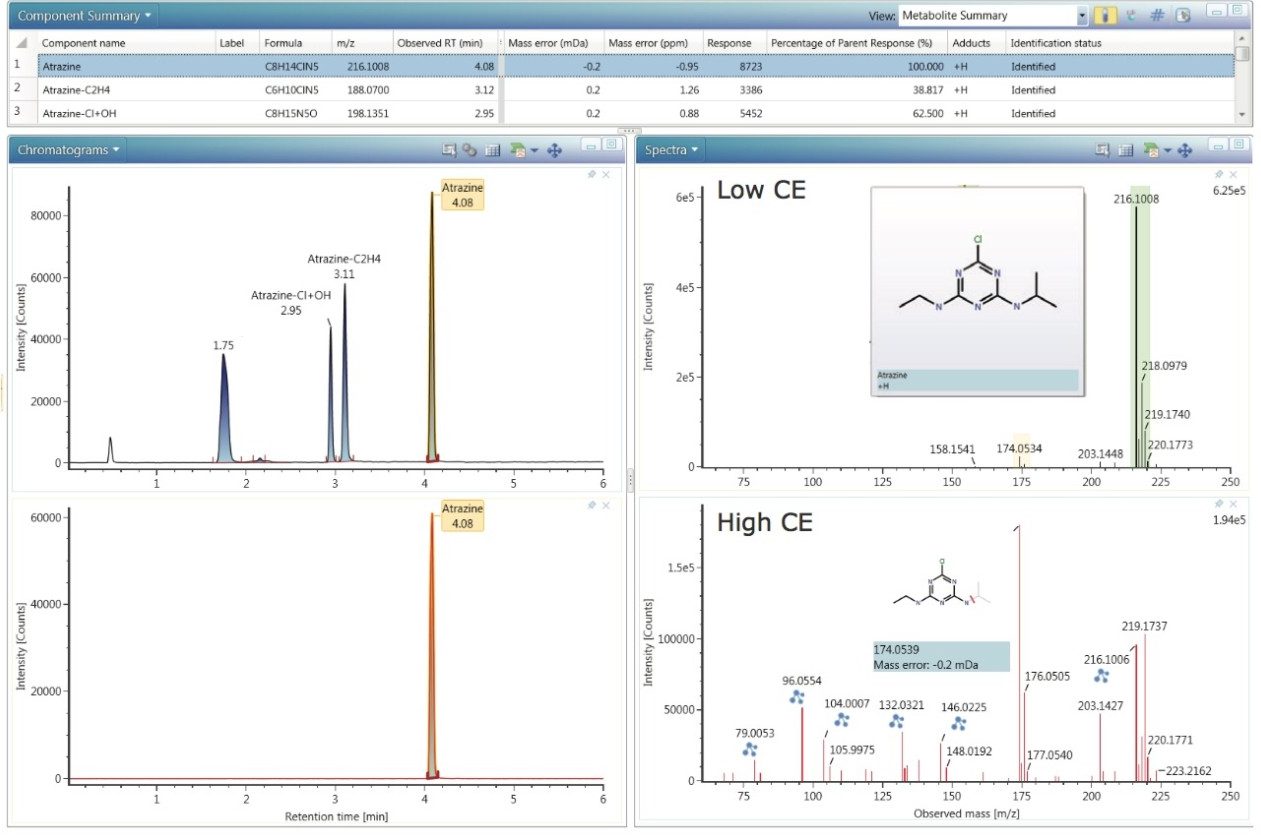
单击左键可在工作流程步骤设置的预定义显示画面中调用审查鉴定出的代谢物所需的信息。“代谢物鉴定审查”(图4)将显示“组分汇总”、“色谱图”和“质谱图”窗口。“组分汇总”窗口列出了软件鉴定出的代谢物列表。上方色谱图显示的是所有鉴定出的代谢物的XIC,下方的迹线显示的是选定组分的XIC(在本示例中为母体农药阿特拉津)。右侧谱图显示的是低碰撞能量和高碰撞能量(CE) MSE数据,以及可能的产物离子结构匹配结果。在汇总表中,阿特拉津的下方列出了2种代谢物。选定代谢物后即可查看相应的色谱图、质谱图以及产物离子结构匹配结果。
在该工作流程步骤中,用户可利用二元比较功能自动对比对照/参比样品与一种未知样品。“组分汇总”中鉴定出了保留时间(tR)2.95 min处m/z为198.1351的代谢物,该物质经历了-Cl+OH的结构修饰(图5)。此转化表明该代谢物可能是2-羟基阿特拉津。该代谢物是未知测试样品中独有的物质,因为上方的迹线图中并未出现该物质的色谱峰或质谱峰。可查看高CE数据或低CE数据质谱图。本应用纪要中展示的是低CE谱图。
“代谢物层级图”可根据代谢物鉴定结果提供整体的代谢机制概览。位于图中央的是母体农药,它的四周是鉴定出的代谢物。用户在同一位置即可看到完整的代谢途径以及获取有用的数据,包括MSE谱图数据、母体物质及其全部代谢物的质谱碎片离子匹配结果,还可以使用“转化定位”功能(图7)。

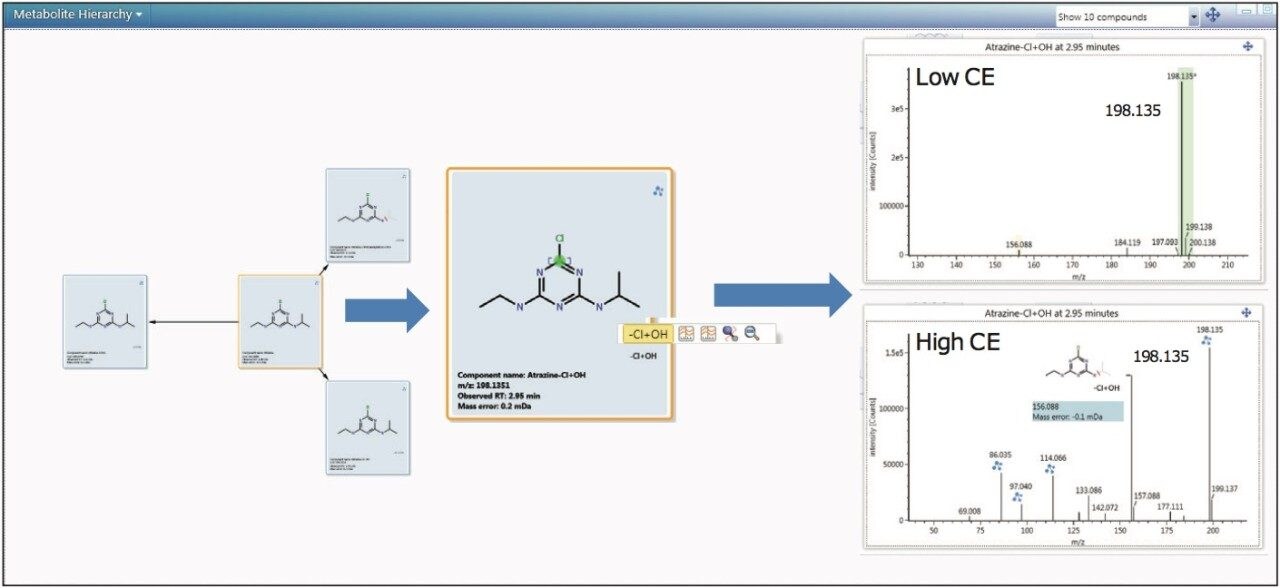
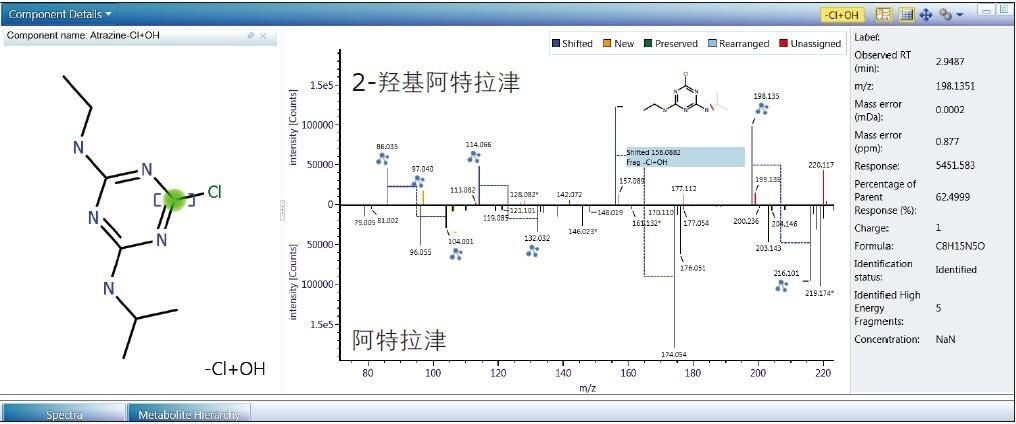
软件将自动执行转化定位功能,为用户提供可能发生代谢转化的位置的相关信息。交互式界面非常便于数据审查(图7)。软件会使用绿色突出显示物质结构中最有可能发生生物转化的位置,指示该位点发生转化的几率极高。软件会将上方的代谢物高能量MSE谱图与下方的母体农药质谱图进行比对。谱图间的差异将突出显示,并且软件将根据UNIFI鉴定出的转化关系给出由颜色编码的匹配结果。本示例中存在若干质谱转换,在图7中由连接代谢物质谱图中的质谱峰与母体农药质谱图中的质谱峰的深蓝色虚线表示。本示例中导致质谱转换的原因是–Cl+OH结构变化。
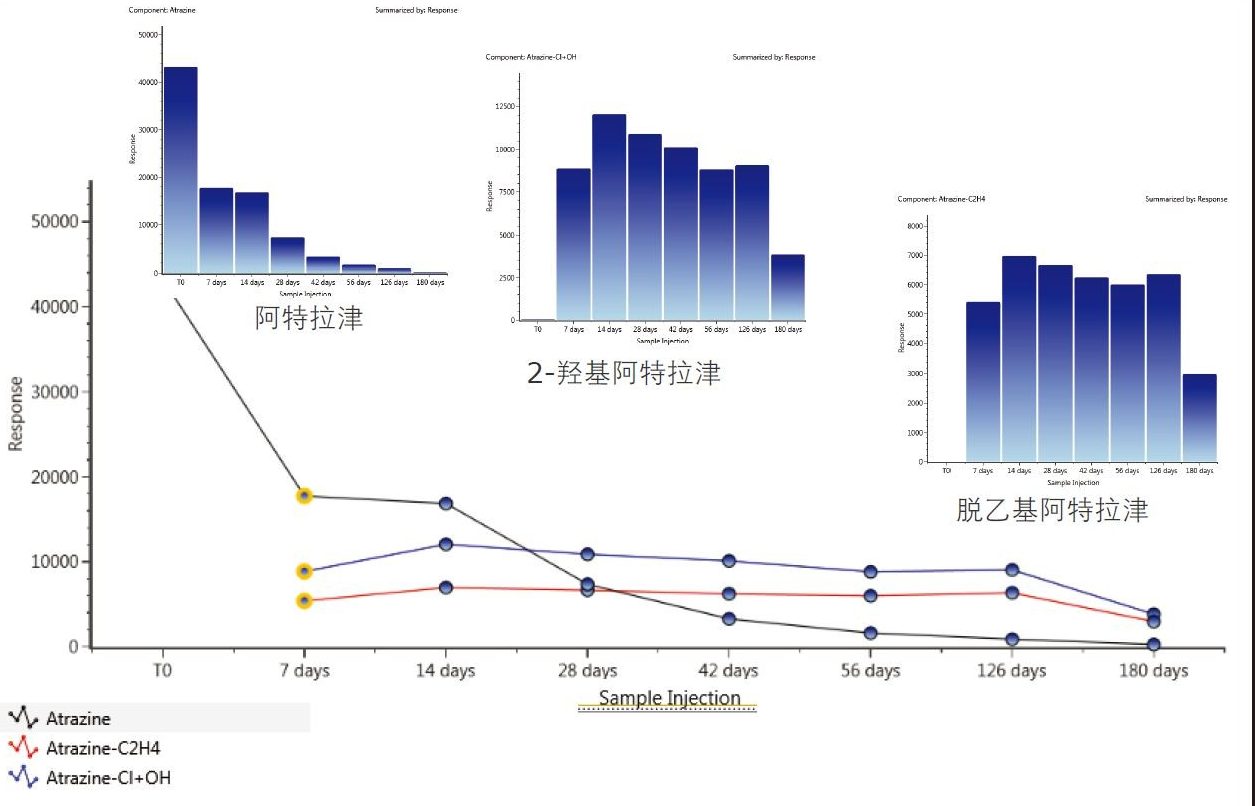
利用UNIFI代谢物鉴定工作流程,用户能够可靠地查询样品中鉴定出的所有组分。用户可利用UNIFI的TrendPlot功能观察代谢物相对于母体农药的转化趋势(图8)。为说明TrendPlot的用途,本应用纪要采用不同加标浓度的样品以及多个时间点设计了一组生物转化动力学研究实验。图中展示了如何利用UNIFI的软件功能监测样品中主要代谢物(随着母体化合物随时间的代谢而出现)的归趋。
趋势图中的每个数据点在UNIFI中都有对应的色谱图、质谱图和结构信息记录。该数据可以表格形式或图片形式显示。图8中的数据以柱状图形式显示并叠加了折线图。
通过工作流程步骤完成代谢物鉴定结果的审查之后,用户即可利用解析工具集研究数据。利用UNIFI中的解析工具集能够更加集中地查询代谢物信息,该工具集包括全套的结构解析工具,包括英国皇家化学学会的ChemSpider搜索(www.chemspider.com)、同位素建模以及元素组成工具。此外还包括利用质量缺失、共有碎片离子和中性丢失搜索相关离子数据的工具。
MSE数据的采集是非靶向的,因此无需详细的分析物信息。用户可利用碎片离子数据搜索样品中的结构相关组分。
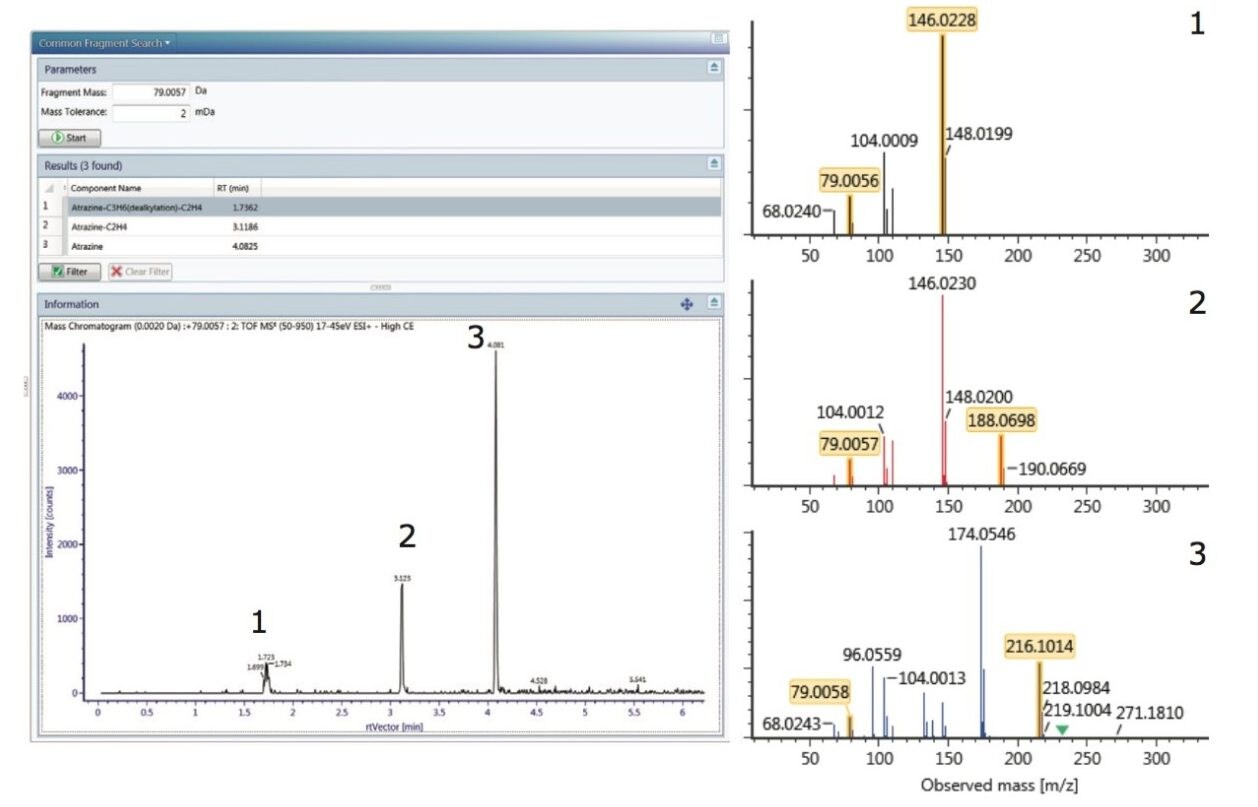
我们采用转化定位功能对鉴定结果为阿特拉津-C2H4(或脱乙基阿特拉津)的代谢物进行研究,结果表明一些产物离子继续出现在了代谢物中(数据未显示)。母体农药和脱乙基阿特拉津都具有m/z 79.005的碎片离子。
利用UNIFI的共有碎片搜索功能,可高效地从数据中提取出具有共同结构特征的成分(图9)。m/z 79.005的碎片存在于阿特拉津(峰3)、脱乙基阿特拉津(峰2)以及另外一种保留时间为(tR) 1.74 min的物质(峰1)的产物离子谱图中。经鉴定,该组分经历了2种脱烷基化作用,C3H6和C2H4(脱乙基脱异丙基阿特拉津)。高能量碎片离子数据以及完整母离子数据所提供的信息让用户仅通过单次进样就能建立代谢物之间的结构关系。
代谢物一经鉴定即可被标记并输入UNIFI科学数据库,数据库可存储tR、碎片离子精确质量数以及质谱图等信息以供未来分析使用。
完成数据分析之后,UNIFI可组织数据,生成内容清晰且可完全自定义的报告。数据审查时可获取的全部信息均可导出到报告中,包括数字、图片、表格、曲线以及结构信息等,如图10所示。UNIFI能够为具体分析生成多种报告,用户可根据内部或外部报告的需求自定义每种报告中所包含信息的详细程度。

采用基于UNIFI的代谢物鉴定应用解决方案成功鉴定了加标到土壤样品中的阿特拉津及其代谢物。可自定义的工作流程步骤让用户能够以最少的用户干预更轻松、更高效地完成数据分析。使用UNIFI的数据评估工具能够轻松将阿特拉津与其代谢物之间的关系可视化。本应用纪要中所展示的阿特拉津及其代谢物的分析工作流程也可应用于类似的农药代谢物鉴定研究,以显著加快新产品注册进程。本研究中所使用的样品是为测试整体分析方法、软件和硬件以及验证原理分析而配制。
720005357ZH,2015年4月