waters_connect für Biopharmazeutika
Für schnellere Biopharma-Analysen
Die Analyse komplexer Biotherapeutika und neuer Modalitäten erfordert effiziente Workflows zur Integration von Aufnahme, Verarbeitung, Überprüfung und Berichterstellung von Analysedaten und Ergebnissen. Analysen für Biologika finden auf mehreren Ebenen statt: dem intakten Molekül, den Untereinheiten, Sequenz-Mapping (Peptid oder Oligo) und markierten freigesetzten Glykanen.
waters_connect für Biopharmazeutika bietet eine Compliance-fähige, vernetzte Informatikplattform und optimierte biopharmazeutische Workflows für die Integration der LC-MS-Systeme eines Unternehmens. Von der Produkt- und Prozesscharakterisierung bis zur gezielteren Attributüberwachung und Release-Assays – der Weg von der Probe zum umsetzbaren Ergebnis war noch nie schneller und zuverlässiger.
Überblick
- Bewältigen Sie Herausforderungen bei der Charakterisierung und Überwachung kritischer Qualitätsmerkmale mit app-basierten integrierten Workflows
- Maximieren Sie die Benutzerfreundlichkeit und beschleunigen Sie die Entscheidungsfindung mit kompletten Workflowlösungen, die optimierte Datenaufnahme, Verarbeitung, Überprüfung und Berichterstellung bieten.
- Reduzieren Sie die Zeit bis zur Erstellung von Ergebnissen und erhöhen Sie die Flexibilität beim Screening einer großen Anzahl von Biomolekülen, einschließlich Proteinen, RNA-Molekülen, Peptiden, Oligonukleotide und Metaboliten aus verbrauchten Medien.
- Überwachen Sie routinemäßig die Qualitätsmerkmale biopharmazeutischer Prozesse und Produkte auf Peptidebene und identifizieren Sie unerwartete Peaks mit der Funktion New Peak Detection (NPD; Erkennung neuer Peaks)
- Kombinieren Sie die Produkt- und Prozesscharakterisierung mit Überwachungs- und Freigabe-Assays mithilfe einer einzigen, Compliance-fähigen Softwarelösung.
Empfohlene Verwendung: Biopharmazeutische LC-MS-Workflows für Protein- und Nukleinsäure-basierte Therapeutika, Impfstoffe und Metaboliten aus Zellkulturen
Features Header
Moderne, Compliance-fähige Netzwerkstruktur
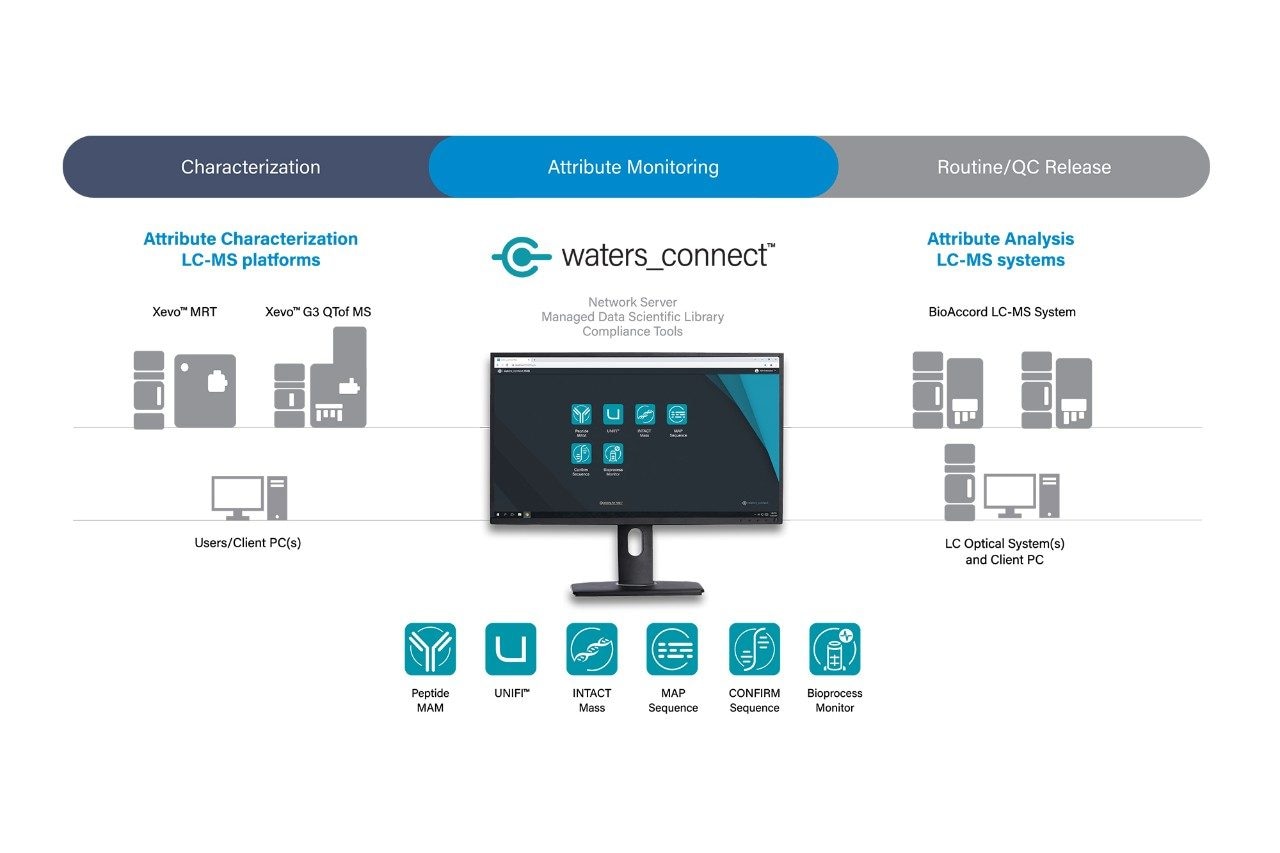
waters_connect für Biopharmazeutika wurde speziell für den netzwerkbasierten Zugriff auf Ihre Systeme und Daten entwickelt und ermöglicht einen effizienten Laborbetrieb und ein effizientes Informationsmanagement.
- Vernetzte Schnittstelle, die eine Vielzahl von Massenspektrometern, wie Quadrupol-Time-of-Flight (QTof) und BioAccord LC-TOF MS Systeme, sowie eigenständige UPLC-Systeme mit optischer Detektion unterstützt
- Kontrollierter Zugriff auf Ihre Daten in Ihrem Labor, im Büro oder an anderen vernetzten Orten
- Branchenführende Compliancetools und professionelle Dienstleistungen, die Sie bei Ihren Bemühungen um Datenintegrität und bei der Einhaltung von Vorschriften unterstützen
- API-Schnittstelle und Exporttools für die Interaktion mit Software von Drittanbietern
Spezielle Workflows für biopharmazeutische Applikationen
waters_connect bietet integrierte App-basierte Workflows, die Sie direkt für die Attributcharakterisierung und Attributüberwachung einsetzen können, einschließlich:
- Analyse der intakten Massen (Proteine, Untereinheiten, Nukleinsäuren oder Peptide)
- Peptidkartierung durch datenabhängige Aufnahme (DDA) und MSE-basierte datenunabhängige Aufnahme (DIA)
- Peptid-Multi-Attribut-Methode (MAM)
- Analyse abgespaltener N-Glykane (Fluoreszenz (FLR)/MS)
- Oligonukleotid-Mapping und Sequenzbestätigung
- Analyse des Proteintiters und des verbrauchten Mediums
- Metabolitanalyse von verbrauchten Medien
Analyse intakter Massen
Die Analyse der intakten Masse von Biologika ermöglicht Anwendern Folgendes:
- die Optimierung der Massenbestätigung und Reinheitsbestimmung von Biomolekülen, einschließlich Proteinen, Peptiden, Oligonukleotiden und Konjugaten
- die Beurteilung kritischer Eigenschaften von sgRNA- und mRNA-Molekülen
- die Steigerung der Laborproduktivität für die Analyse intakter Massen dank intelligenter, automatisierter Dekonvolution
- einen schnelleren Erhalt von Ergebnissen und größere Flexibilität dank automatisierter Aufnahme und Prozessierung
- die Nutzung der branchenführenden spektralen Dekonvolutionsalgorithmen BayesSpray oder MaxEnt1, um monoisotope oder durchschnittliche Massen für Ihre Moleküle zu erhalten
- sowohl die MS- als auch die UV-basierte Quantifizierung von Verunreinigungen und die Identifizierung von Verunreinigungen
- die Massenbestätigung mit hoher Kapazität und hohem Durchsatz und die Reinheitsbestimmung von Hunderten von eindeutigen Biomolekülen pro Tag
- die automatische Berechnung des Wirkstoff-zu-Antikörper-Verhältnisses (Drug-Antibody-Ratio, DAR) und der Wirkstoffverteilung für konjugierte Biomoleküle
Bioprocess Monitor
Die App Bioprocess Monitor sorgt für eine einfache Prozessierung von LC-MS- und LC-UV-Daten und kann flexibel in verschiedene Workflows und Softwarepakete integriert werden. Sie wurde speziell für Labore mit Bioprozesssystemen entwickelt und ermöglicht Ihnen Folgendes:
- Unterstützen Sie die routinemäßige Probenanalyse von Metaboliten aus verbrauchten Medien oder die Proteintiteranalyse mit schnellen und einfach einzurichtenden Methoden.
- Generieren Sie nahezu in Echtzeit Ergebnisse für analytische Walk-up-Systeme, die Proben schnell quantifizieren können, und sorgen Sie für eine zukunftssichere Integration mit At-Line-/Online-Ansätzen.
- Erhalten Sie einfach Messwerte für Assays wie LC-basierte Titer und verbrauchte Medienkomponenten (Aminosäuren, Vitamine, Metaboliten usw.)
- Direkte Integration in Automatisierungsysteme von Waters, die einfache und komplexe Probenvorbereitungen ermöglichen, wie z. B. OneLab und Andrew Alliance Systeme.
- Einfache Integration in externe, gängige Plattformen wie Ambr® 15 oder Ambr 250 HT-Systeme oder die JMP® Software
Analyse abgespaltener N-Glykane
Der Workflow für abgespaltene N-Glykane automatisiert die Aufnahme, Verarbeitung und Berichterstellung von Profilen markierter N-Glykane mithilfe von FLR- und MS-basierter Detektion:
- Automatisierter Abgleich von FLR- und MS-Chromatogrammen
- Dextran-Kalibrierung für die auf der Retentionszeit basierende Zuordnung von Glukoseeinheiten (GU)
- Glykan GU-Bibliotheken für 2-AB, 2AA und RapiFluor-MS-markierte Glykane
- Erstellung benutzerdefinierter GU-Bibliotheken für Ihr Molekül
- FLR-Quantifizierung mit genauer Massenbestätigung
Peptidkartierung
Der Peptidkartierungsworkflow automatisiert die Analyse von verdauten, Protein-basierten therapeutischen Substanzen mittels MS, DDA-MS/MS und DIA MSE-basierter Akquisition und ermöglicht:
- Peptididentifizierung basierend auf genauer Massen- und Fragmentbestätigung
- Sequenzabdeckung auf Peptid- und Fragmentebene
- Zuordnung von Proteinmodifikationen und Berechnung der prozentualen Modifikationsgrade
- Sammeln von m/z, Retentionszeit und Informationen zum Signal des Ladungszustands, um Bibliotheken für die gezielte Analyse mittels Peptide MAM zu erstellen
Peptide MAM
Die Applikation Peptide MAM bietet einen automatisierten Workflow für die Überwachung von Attributen der Produktqualität, die ID-Bestätigung und neue Reinheitstests auf Basis der Erkennung neuer Peaks. So können Anwender:
- Datenaufnahme, Systemeignung, Datenüberprüfung und Reporterstellung effizient in ein einfaches grafisches Workflow-Design integrieren
- Die Liste der Zielattribute direkt aus der wissenschaftlichen Bibliothek in waters_connect, der Importdatei oder durch manuelle Eingabe ausfüllen
- Fehlende Peaks vermeiden und die Quantifizierung mit erweiterten RT-Ausrichtungstools verbessern
- Die Produktivität durch eine robuste, neue Peakerkennung für potenzielle Verunreinigungen verbessern, die eine hohe Empfindlichkeit bei niedrigen falsch-positiven Erkennungsraten bietet
Oligo-Kartierung und Sequenzierung verdauter RNA
Die waters_connect MAP Sequence App ist eine speziell für waters_connect entwickelte Anwendung zur beschleunigten Charakterisierung größerer RNA-Moleküle (z. B. sgRNA und mRNA) durch die Verarbeitung von LC-MS- and LC-MSE-Karten, die aus enzymatisch verdauten RNA-Proben gewonnen wurden. Dadurch sind Anwender zu Folgendem in der Lage:
- Optimieren der Bestätigung der RNA-Sequenz und -Modifikationen.
- Durchführen einer Oligo-Kartierungsanalyse von RNA-Molekülen, die modifizierte Backbones, Nukleoside und Verknüpfungen enthalten, einschließlich der Möglichkeit, benutzerdefinierte Moleküle zu verwenden.
- Kombinieren von Ergebnissen mehrerer Oligokarten, die mit dem traditionellen RNase T1-Verdau und dem wachsenden Toolset von RNA-Verdauenzymen wie Waters RapiZyme RNasen und benutzerdefinierten Enzymen mit alternativen Verdauspezifitäten erstellt wurden, um eine maximal zuverlässige Sequenzabdeckung zu erhalten.
- Verringern des Zeitaufwands für manuelle Datenanalyse mit Datenvisualisierungstools und optimierten Ausnahme-Datenüberprüfungsfunktionen, die den Anwender auf Bereiche mit potenziell mehrdeutigen Ergebnissen aufmerksam machen.
Sequenzierung von Oligonukleotiden
Die Applikation CONFIRM Sequence ist der erste Workflow, der für die schnellere Charakterisierung synthetischer Oligonukleotide und verdauter RNA-Fragmente entwickelt wurde. Sie ermöglicht Anwendern:
- die Optimierung der Bestätigung von Sequenzen und der Charakterisierung von Verunreinigungen mit einem speziellen Workflow
- Effizienzsteigerungen durch schnelle Analyse und Berechnung der Sequenzabdeckung aus mehreren gezielten (MS/MS) oder ungezielten (MSE) Experimenten
- eine höhere Konfidenz durch automatisierte Lokalisierung von Modifikationen
- eine schnellere Beurteilung der Daten dank einzigartiger Datenvisualisierungstools und umfassender Funktionen zur Überprüfung von Rohdaten
- eine Einhaltung der regulatorischen Vorschriften mit der Compliance-fähigen waters_connect Plattform